- Распределение нормированных весов находок в протеомах двух групп бактерий
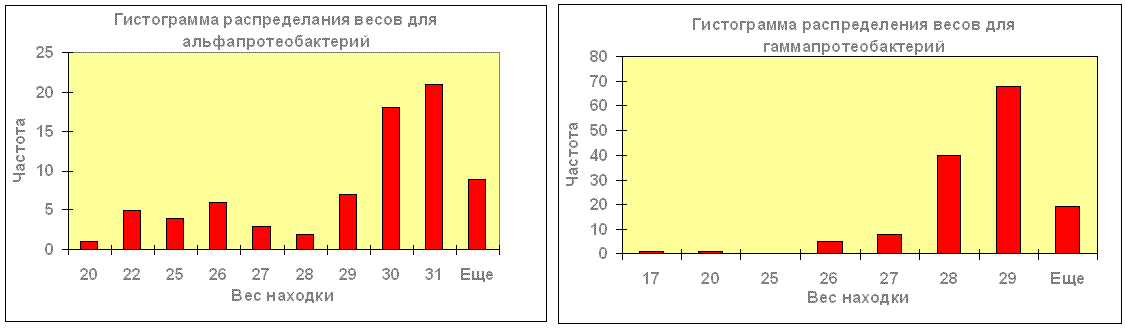
На приведённых гистограммах можно увидеть распределение весов найденных белков среди альфа- и гаммапротеобактерий. При сравнении гистограмм заметно, что веса находок среди альфапротеобактерий больше, чем среди гаммапротеобактерий; при том, что число находок среди гаммапротеобактерий почти в 2 раза больше, диапазон распределения весов среди альфапротеобактерий больше.
Рабочие файлы:
- Тест Вилкоксона
Для сравнения выборок полученных весов был проведён тест Вилкоксона. Результаты теста представлены ниже:
Вилкоксон=1.06E4, Z=-5.123, Значимость=0, степ.своб = 76,142
Гипотеза 1: <Есть различия между медианами выборок>
Сравним медианы каждой из выборок с помощью описательной статистики:
Alpha: Gamma:
Размер <---Диапазон---> Среднее---Ошибка Дисперс Ст.откл Сумма Размер <---Диапазон---> Среднее---Ошибка Дисперс Ст.откл Сумма
76 19.86 33 28.42 0.3606 9.884 3.144 2160 142 16.88 29.64 27.89 0.1289 2.36 1.536 3960
Медиана <--Квартили--> ДовИнтСр. <-ДовИнтДисп-> Ош.СтОткл Медиана <--Квартили--> ДовИнтСр. <-ДовИнтДисп-> Ош.СтОткл
29.45 26.73 30.27 0.709 8.254 9.884 0.8976 28.16 27.58 28.56 0.2515 1.377 1.377 0.3747
Асимметр. Значим Эксцесс Значим Асимметр. Значим Эксцесс Значим
-1.253 0 3.658 0.0742 -4.808 0 33.65 0
Различия, замеченные нами при построении гистограмм подтвердил тест Вилкоксона. Проанализировав данные описательной статистики о различии между средним значением и медианой, можно заключить, что выборка из альфапротеобактерий ближе к изучаемой выборке.
- Филогенетический анализ рибосомальных белков S7 из эукариотических митохондрий и 3-х групп бактерий
Получим fasta-последовательности белков по выбранным нами идентификаторам для альфа- и гаммапротеобактерий. С помощью поиска SRS получим также последовательности белков из рибосом фирмикут. Объединив все полученные последовательности с исходными митохондриальными построим множественное выравнивание с помощью muscle .
Описание выборки в виде таблицы:
Группа источников рибосом Число последовательностей
альфапротеобактерии 76
гаммапротеобактерии 142
фирмикуты 7
митохондрии 10
На основе построенного выравнивания методом максимального правдоподобия построим филогенетическое дерево для всех белков:
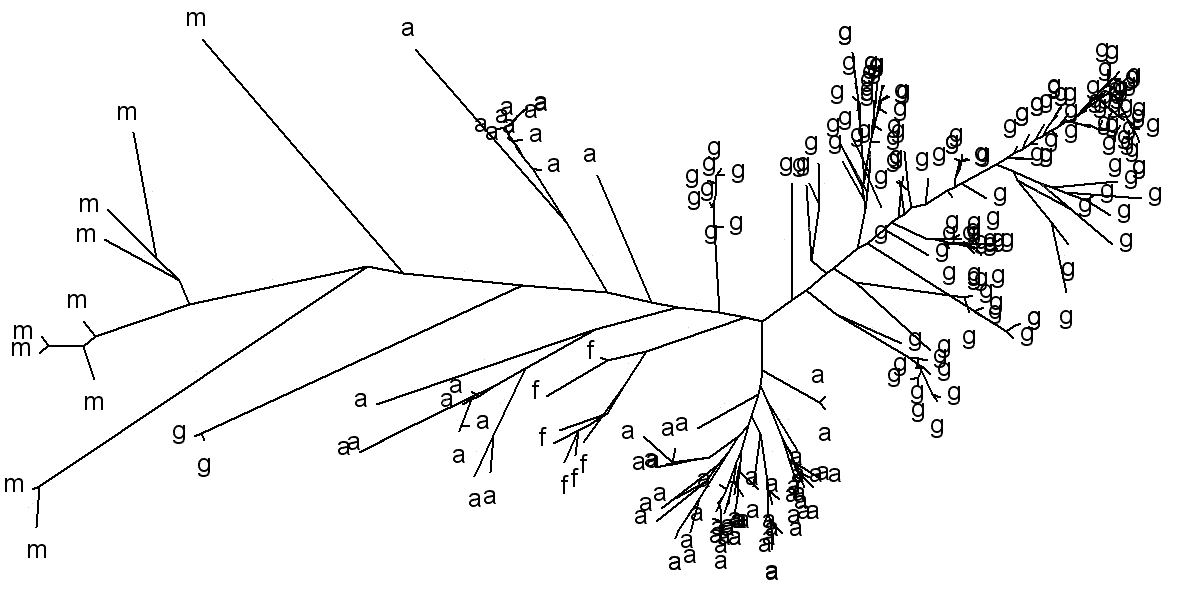
На представленном дереве последовательности из митохондрий эукариот обозначены буквой "m", белки из альфа- и гаммапротеобактерий - буквами "a" и "g" соответсвтенно, из фирмикут - "f". Длина ветвей сохранена, расстояния не отображаются.
За исключением двух веток с гаммапротеобактериями, все последовательности из этой группы сосредоточены на одном конце дерева, группа из фирмикут находится на отдельной ветке, а группа митохондриальных белков ближе к группе альфапротеобактерий, что подтверждает существующее мнение о том, что митохондриальные белки произошли от белков альфапротеобактерий.
Определим попарные эволюционнные расстояния между группой митохондриальных белков и группами белков из протеомов альфа- и гаммапротеобактерий с помощью программы protdist пакета phylip.
Ниже приведены гистограммы распределений этих расстояний:
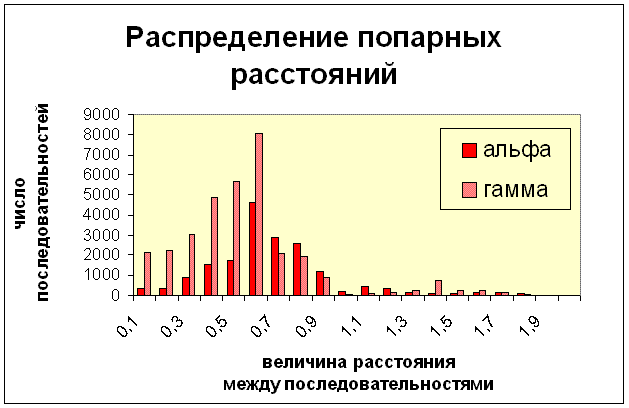
Можно заметить, что медианы распределений для альфа- и гаммапротеобактерий лежат в одном интервале, лишь для гаммапротеобактерий существует хвост нормального распределения, что достаточно любопытно, возмозжно, это объясняется тем, что гаммапротеобактерии удалены от остальных групп сильнее всего, поэтому расстояние в районе 1,5 столь "популярно".
Рабочий файл:
- Вывод
Таким образом, по результатам проделанной работы, можно заключить, что наши исследования подтверждают мнение о том, что митохондриальные белки эукариот ближе к альфапротеобактериям, чем к гаммапротеобактериям. На это указывают результаты описательной статистики, построенное дерево и гистограмма попарных расстояний, на которой заметно, что гаммапротеобактерии удалены от эукариотических белков сильнее, чем альфапротеобактерии. При этом, нельзя считать полученные нами результаты абсолютно правильными, в силу малой выборки эукариотических белков, большой разницы между выборками, например, из гаммапротеобактерий и митохондрий эукариот.



