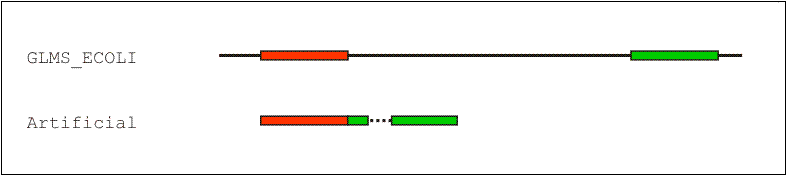
Выравнивание проводилось с помощью программы Needle при gap_penalty = 10.0 и extend_penalty = 0.5. Совпадающим участкам последовательностей соответствуют цветные прямоугольники, гэпы обозначены пунктиром (см. выравнивание №4).
Главная страница > Второй семестр > Программы выравнивания
Было проведено глобальное и локальное выравнивание последовательностей GLMS E.coli и
GLMS Thiobacillus ferrooxidans (использовалась матрица замен аминокислотных остатков BLOSUM62;
gap_penalty = 10.0; extend_penalty = 0.5). Глобальное выравнивание, осуществленное с помощью программы
Needle, показало, что последовательности гомологичны друг другу (см. выравнивание №1).
Локальное выравнивание, осуществленное с помощью программы Water (см. выравнивание №2), совпало с глобальным. Это можно объяснить тем,
что выравниваемые последовательности гомологичны друг другу на всем своем протяжении. Таким образом, наилучший сходный
участок последовательностей совпадает с самой последовательностью.
Было проведено глобальное выравнивание последовательности GLMS E.coli и искусственной последовательности,
составленной из двух ее фрагментов длиной по 12 аминокислотных остатков (использовалась программа Needle, параметры
выравнивания: матрица замен аминокислотных остатков BLOSUM62; gap_penalty = 10.0; extend_penalty = 0.1). Глобальное выравнивание позволило установить, каким
участкам последовательности GLMS соответствует каждый из фрагментов искусственной последовательности (см. выравнивание №3). При попытке
проведения выравнивания с параметрами, задаваемыми по умолчанию (gap_penalty = 10.0, extend_penalty = 0.5), один из
фрагментов искусственной последовательности был ошибочно сопоставлен с участком последовательности GLMS (рис. 1, см. выравнивание №4).
Ошибка была устранена путем уменьшения значения параметра extend_penalty до 0.1. В данном случае на вес
глобального выравнивания оказывает влияние только длина центрального гэпа, так как концевые гэпы программой Needle не
учитываются. Таким образом, наиболее предпочтительным (при достаточно высоком значении extend_penalty и небольшой
длине совпадающих участков) оказывается выравнивание с коротким центральным гэпом (см. рис. 1). При уменьшении значения
extend_penalty в формировании веса выравнивания начинают преобладать баллы, начисляемые за сходные участки
последовательностей, и программа выводит правильное выравнивание.
 |
|
Рис. 1. Результаты глобального выравнивания последовательности GLMS E.coli (GLMS_ECOLI) и искусственной последовательности, составленной из двух ее фрагментов (Artificial). Выравнивание проводилось с помощью программы Needle при gap_penalty = 10.0 и extend_penalty = 0.5. Совпадающим участкам последовательностей соответствуют цветные прямоугольники, гэпы обозначены пунктиром (см. выравнивание №4). |
Также было проведено локальное выравнивание этих же последовательностей с использованием программ
Water и Matcher (BLOSUM62; gap_penalty = 10.0; extend_penalty = 0.5, сохранены три наилучших выравнивания, полученных
с помощью Matcher). Выравнивание с помощью Water (см. выравнивание №5) позволило установить один из совпадающих участков последовательностей
(вес выравнивания данного участка с самим собой при использовании матрицы весов замен BLOSUM62 превосходит вес
выравнивания другого совпадающего участка с самим собой).
Выравнивание с помощью Matcher (см. выравнивание №6) позволило выявить три сходных участка. Два из них представляют собой
совпадающие фрагменты последовательностей (веса выравниваний составляют 73 и 59). Появление третьего участка со
значительно меньшим весом выравнивания (17) обусловлено случайными совпадениями аминокислотных остатков
последовательности GLMS и искусственной последовательности (сходство не имеет биологического смысла).
Было проведено глобальное выравнивание последовательности GLMS E.coli и искусственной последовательности
помощью программы Needle при различных значениях параметра gap_penalty (матрица замен аминокислотных остатков BLOSUM62; gap penalty = 1.0, 5.0, 10.0; extend_penalty = 0.2). Значение параметра extend_penalty было выбрано меньшим 1.0 по причинам, описанным выше.
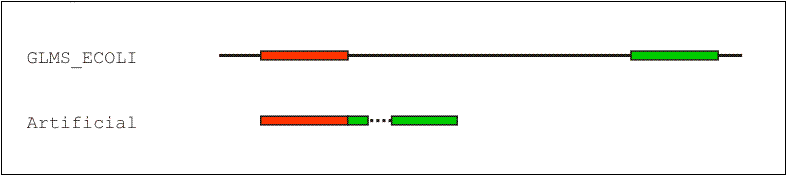
При gap_penalty = 10 один из фрагментов искусственной последовательности (обладающий наибольшим весом выравнивания с
самим собой) был выровнен правильно, а другой (обладающий меньшим весом выравнивания с самим собой) был сопоставлен
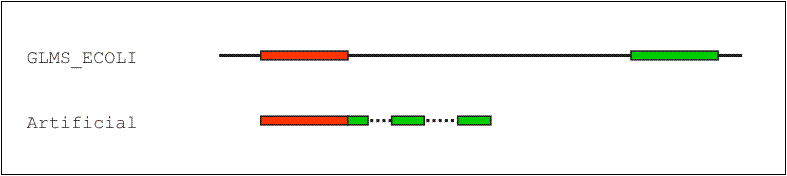
с близлежащим участком последовательности GLMS (рис. 2, см. выравнивание №7). При gap_penalty = 5 фрагмент искусственной последовательности
с наибольшим весом выравнивания с самим собой выровнен правильно, а другой фрагмент разбит двумя гэпами и сопоставлен
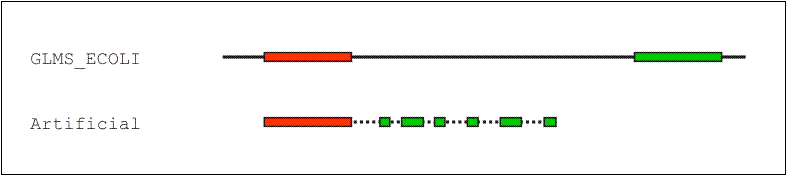
близлежащему участку последовательности GLMS (рис. 3, см. выравнивание №8). При gap_penalty = 1 один из фрагментов
искусственной последовательности выровнен правильно, а другой разбит множеством гэпов на отдельные аминокислотные остатки,
сопоставленные совпадающим аминокислотным остаткам последовательности GLMS (рис. 4, см. выравнивание №9).
На основании проведенных экспериментов можно сделать следующие выводы:
Правильное выравнивание может быть получено при уменьшении extend_penalty до 0.1 и gap_penalty = 10.0, см. выше.
 |
|
Рис. 2. Результаты глобального выравнивания последовательности GLMS E.coli (GLMS_ECOLI) и искусственной последовательности, составленной из двух ее фрагментов (Artificial). Выравнивание проводилось с помощью программы Needle при gap_penalty = 10.0 и extend_penalty = 0.2. Обозначения как на рис. 1 (см. выравнивание №7). |
 |
|
Рис. 2. Результаты глобального выравнивания последовательности GLMS E.coli (GLMS_ECOLI) и искусственной последовательности, составленной из двух ее фрагментов (Artificial). Выравнивание проводилось с помощью программы Needle при gap_penalty = 5.0 и extend_penalty = 0.2. Обозначения как на рис. 1 (см. выравнивание №8). |
 |
|
Рис. 2. Результаты глобального выравнивания последовательности GLMS E.coli (GLMS_ECOLI) и искусственной последовательности, составленной из двух ее фрагментов (Artificial). Выравнивание проводилось с помощью программы Needle при gap_penalty = 1.0 и extend_penalty = 0.2. Обозначения как на рис. 1 (см. выравнивание №9). |
Построение карт локального сходства последовательности GLMS E.coli и искусственной последовательности было осуществлено с помощью программы Dotmatcher. Построение проводилось при различных значениях параметров windowsize и threshold, использовалась матрица замен аминокислотных остатков BLOSUM62.
Чтобы установить влияние параметра windowsize на построение карт локального сходства, были проведены три выравнивания при различных значениях windowsize (5, 10, 15) и постоянном значении threshold = 23 (рис. 5). Выравнивания показали, что увеличение значения параметра windowsize приводит к увеличению длин выявляемых программой сходных участков последовательностей. Следовательно, windowsize определяет длину выявляемых сходных участков.
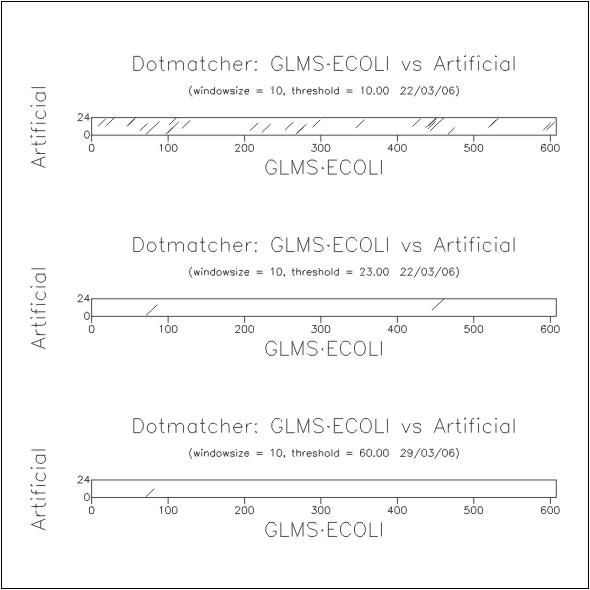
Чтобы установить влияние параметра threshold на построение карт локального сходства, были проведены три выравнивания при различных значениях threshold (10, 23, 60) и постоянном значении windowsize = 10 (рис. 6). Выравнивания показали, что увеличение значения параметра threshold приводит к уменьшению количества выявляемых программой сходных участков (при threshold = 10 выявлено около 20 сходных участков, при threshold = 23 — два сходных участка, при threshold = 60 — один сходный участок). Можно предположить, что эта закономерность обусловлена тем, что threshold определяет пороговое значение веса выравнивания выявляемых сходных участков. Так как вес выравнивания одного из совпадающих фрагментов последовательностей с самим собой равен 59 (см. "Выравнивание последовательностей, содержащих общие участки"), программа Dotmatcher при threshold = 60 не позволяет обнаружить этот участок. С другой стороны, существует достаточно большое количество участков последовательностей, вес выравнивания которых в результате случайных совпадений между аминокислотными остатками превышает 10 (в данном случае программа выявила около 20 таких участков).
Таким образом, правильная карта локального сходства может быть получена при windowsize = 12 и, например, threshold = 59 (так как совпадающие участки аминокислотных последовательностей имеют длину 12 аминокислотных остатков и наименьший из весов выравнивания таких участков с самими собой равен 59, рис. 7). Такая карта состоит из двух штрихов, соответствующих двум совпадающим участкам последовательностей, причем проекции этих штрихов на ось ординат не пересекаются друг с другом и образуют непрерывный отрезок.
 |
|
Рис. 5. Карты локального сходства последовательности GLMS E.coli и искусственной последовательности, построенные с помощью программы Dotmatcher при различных значениях параметра windowsize (5, 10, 15) и постоянном значении threshold (23).
|
 |
|
Рис. 6. Карты локального сходства последовательности GLMS E.coli и искусственной последовательности, построенные с помощью программы Dotmatcher при различных значениях параметра threshold (10, 23, 60) и постоянном значении windowsize (10).
|
 |
|
Рис. 7. Карта локального сходства последовательности GLMS E.coli и искусственной последовательности, построенная с помощью программы Dotmatcher при threshold = 59 и windowsize = 12.
|
© Куравский Михаил Львович, 2006