Исследование структуры тРНК
- Краткое описание структуры в файле 1I9V.pdb
Организм: SACCHAROMYCES CEREVISIAE
В файле приведены координаты атомов следующих молекул:
PHENYLALANINE TRANSFER RNA (цепь A) - 1 молекула
Для исследования была выбрана цепь A, представляющая собой фенилаланин-тРНК со следующей последовательностью:
[1] 5' -
GCGGAUUUAGCUCAGUUGGGAGAGC
GCCAGACUGAAYGAUCUGGAGGUCC
UGUG5MUUCGAUCCACAGAAUUCGC
ACCA
- 3' [76]
где YG - вибутозин, 5MU - 5-метилуридин
На 3' конце последовательности находится триплет CCA, к которому присоединяется аминокислота - фенилаланин, в PDB файле приведены координаты его атомов.
- Исследование вторичной структуры
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи
между азотистыми основаниями (выходной файл программы TRNK.out). Руководствуясь полученными данными,
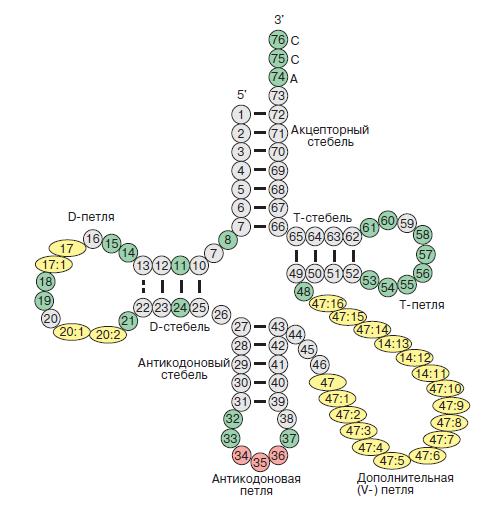
а также схемой строения тРНК (источник):
RMSD of the bases
(----- for WC bp, + for isolated bp, x for helix change)
Strand I Strand II Helix
1 (0.010) A:...1_:[..G]G-----C[..C]:..72_:A (0.013) |
2 (0.011) A:...2_:[..C]C-----G[..G]:..71_:A (0.010) |
3 (0.018) A:...3_:[..G]G-----C[..C]:..70_:A (0.008) |
4 (0.014) A:...4_:[..G]G-*---U[..U]:..69_:A (0.006) |
5 (0.018) A:...5_:[..A]A-----U[..U]:..68_:A (0.008) |
6 (0.009) A:...6_:[..U]U-----A[..A]:..67_:A (0.006) |
7 (0.013) A:...7_:[..U]Ux----A[..A]:..66_:A (0.017) |
8 (0.017) A:..49_:[..C]C-----G[..G]:..65_:A (0.021) |
9 (0.009) A:..50_:[..U]U-----A[..A]:..64_:A (0.012) |
10 (0.013) A:..51_:[..G]G-----C[..C]:..63_:A (0.010) |
11 (0.007) A:..52_:[..U]U-----A[..A]:..62_:A (0.023) |
12 (0.012) A:..53_:[..G]G----xC[..C]:..61_:A (0.008) |
13 (0.011) A:..54_:[5MU]ux**-xA[..A]:..58_:A (0.016) x
14 (0.020) A:..36_:[..A]Ax*---U[..U]:..33_:A (0.011) |
15 (0.018) A:..38_:[..A]Ax*--xC[..C]:..32_:A (0.004) |
16 (0.014) A:..40_:[..C]C-----G[..G]:..30_:A (0.015) |
17 (0.008) A:..41_:[..U]U-----A[..A]:..29_:A (0.014) |
18 (0.008) A:..42_:[..G]G-----C[..C]:..28_:A (0.009) |
19 (0.011) A:..43_:[..G]G-----C[..C]:..27_:A (0.014) |
20 (0.018) A:..44_:[..A]Ax*---G[..G]:..26_:A (0.013) |
21 (0.005) A:..10_:[..G]G-----C[..C]:..25_:A (0.004) |
22 (0.021) A:..11_:[..C]C-----G[..G]:..24_:A (0.014) |
23 (0.019) A:..12_:[..U]U-----A[..A]:..23_:A (0.013) |
24 (0.011) A:..13_:[..C]C----xG[..G]:..22_:A (0.020) |
25 (0.020) A:..14_:[..A]A-**-xU[..U]:...8_:A (0.015) |
26 (0.019) A:..15_:[..G]Gx**+xC[..C]:..48_:A (0.009) x
27 (0.048) A:..19_:[..G]G-----C[..C]:..56_:A (0.014) + |
 |
можно сделать вывод о том, что:
акцепторный стебель состоит из участка 1-7 и комплементарного ему участка 66-72.
Т-стебель состоит из участка 49-52 и комплементарного ему участка 62-65.
D-стебель состоит из участка 10-13 и комплементарного ему участка 22-25.
антикодоновый стебель состоит из участка 39-43 и комплементарного ему участка 27-31
(пара U39-A31 не представлена с списке водородых взаимодействий, хотя в файле pdb координаты этой пара описаны, и в схеме строения тРНК эта пара должна входить в состав антикодонового стебля)
Cкрипт для получения в RasMol изображения остова исследуемой тРНК, где
акцепторный стебель выделен красным, Т-стебель - зеленым, D-стебель - синим, антикодоновый - оранжевым, вариабельная петля - фиолетовым, антикодон - желтым, а также иллюстрация:
|
Рис.1. Вторичная структура
|
Скрипт trna.spt для получения изображения
|
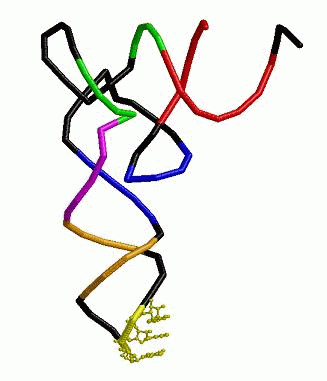 |
restrict none
select all
backbone 150
color black
background white
select 1-7, 66-72
color red
select 49-52, 62-65
color green
select 10-13, 22-25
color blue
select 39-43, 27-31
color orange
select 44-48
color magenta
select 34-36
cpk 120
wireframe 50
color yellow |
Структуру стеблевых дуплексов поддерживают 18 канонических и 1 неканоническая (G4-U69, акцепторный стебель)пара оснований.
Изображение неканонической пары:
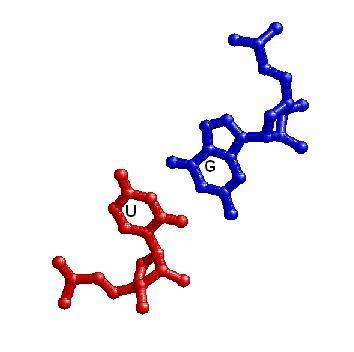
Особенности:
а)Вероятно, в цепи присутствует вариабельная петля (44-48 нуклеотиды);
а)В Т-петле нет тимидина, однако есть 5-метилуридин (он идентичен тимидину за исключением лишней ОН группы в рибозе 5-метилуридина);
а)В D-петле нет дигидроуридина;
Поиск антикодона в антикодоновой петле.
Фенилаланину соответствуют 2 кодона: UUU и UUC. Следовательно, антикодонами будут являться AAA и AAG. В антикодоновой петле подряд идут 2 аденозина и гуанозин - это и есть антикодон, он обозначен на изображении выше желтой шарнирной моделью.
- Исследование третичной структуры
Стекинг взаимодействия.
С помощью данных файла TRNK.out и stacking.pdb было построено изображение стекинг-взаимодействий:
- между концами акцепторной и Т петель, перекрывание слабое:
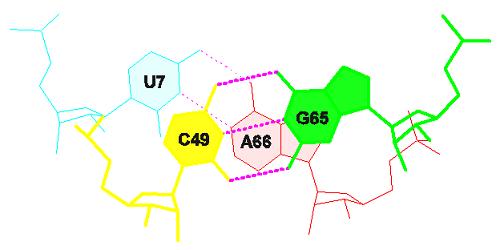
- самое сильное стекинг-взаимодействие - между парами U54-A58 и С61-G53, они находятся в Т-петле:
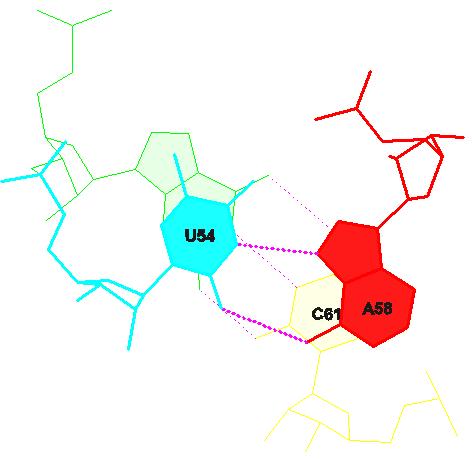
Дополнительная водородная связь между основаниями D- и Т-петель: С56-А19, это каноническое взаимодейтсвие, изображение взаимодействующих оснований:
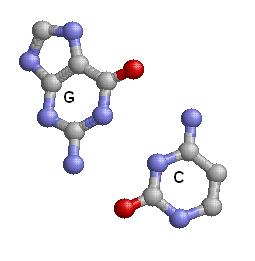
- Предсказание вторичной структуры тРНК
1.Предсказание вторичной структуры тРНК путем поиска инвертированных повторов
При помощи программы einverted из пакета EMBOSS были найдены инвертированные
участки в исследуемой тРНК. Результаты по данным выходного файла trna.einverted занесены в таблицу ниже.
Результаты этой программы базируются на "выравнивании" нуклеотидов относительно комплементраных им, в зависимости от параметров выравниваются разные части тРНК. Наилучший результат был достигнут при штрафе за гэп равном 12, минимальном весе выравнивания 2, весе совпадения 5, весе несовпадения -5.
2.Предсказание вторичной структуры тРНК по алгоритму Зукера.
Программа mfold из пакета EMBOSS реализует алгоритм Зукера.
В программе я подбирала параметр P, наиболее близкое к реальной струтуре предсказание появилось при значении P=10 ( при этом значении создавалось 3 схемы, подходила третья):
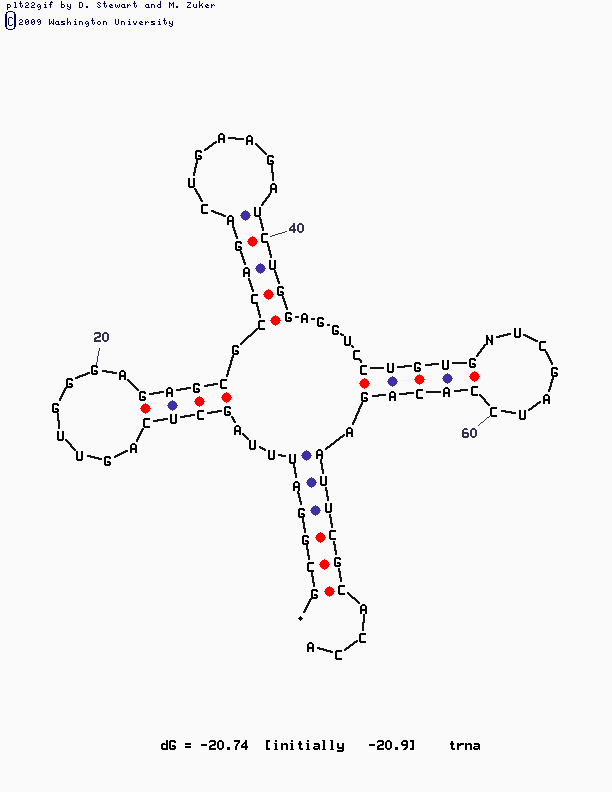
Реальная и предсказанная вторичная структура тРНК из файла 1I9V.pdb
| Участок структуры)
|
Позиции в структуре
(по результатам find_pair)
|
Результаты предсказания
с помощью einverted
|
Результаты предсказания
по алгоритму Зукера
|
| Акцепторный стебель
|
5' 1-7 3'
5' 66-72 3'
Всего 7 пар
|
предсказано 0 пар
|
предсказано 6 пар из 7 (кроме 7-66)
|
| D-стебель
|
5' 10-13 3'
5' 22-25 3'
всего 4 пары
|
предсказано 0 пар
|
все 4 пары предсказаны
|
| T-стебель
|
5' 49-52 3'
5' 62-65 3'
всего 4 пары
|
предсказано 5 пар (дополнительная 53-61)
|
предсказаны все 4 пары + 1 лишняя (53-61)
|
| Антикодоновый стебель
|
5' 39-43 3'
5' 27-31 3'
всего 5 пар
|
предсказано 5 пар
|
все 5 пар предсказаны
|
| Общее число канонических пар нуклеотидов
|
20
|
10
|
20 |
Таким образом, наиболее точными способами определения вторичной структуры тРНК являются программа find_pair и алгоритм Зукера. Второй является предпочтительней, так как создает схему строения тРНК, что нагляднее списка водородных связей. При помощи einverted можно предсказать только часть вторичной структуры тРНК, результаты могут сильно меняться от введения разных параметров.
|