| Организм | Молекула | Количество молекул | Цепь |
| Trichomonas vaginalis | Инициатор связывания 39 кДа (IBP39) | 6 | U, P, F, V, M, O |
| Trichomonas vaginalis | α-SCS элемента промотора INR | 4 | E, I, Y, K, T, R, J, G |
| Trichomonas vaginalis | Сульфат анион | 10 | O, V |
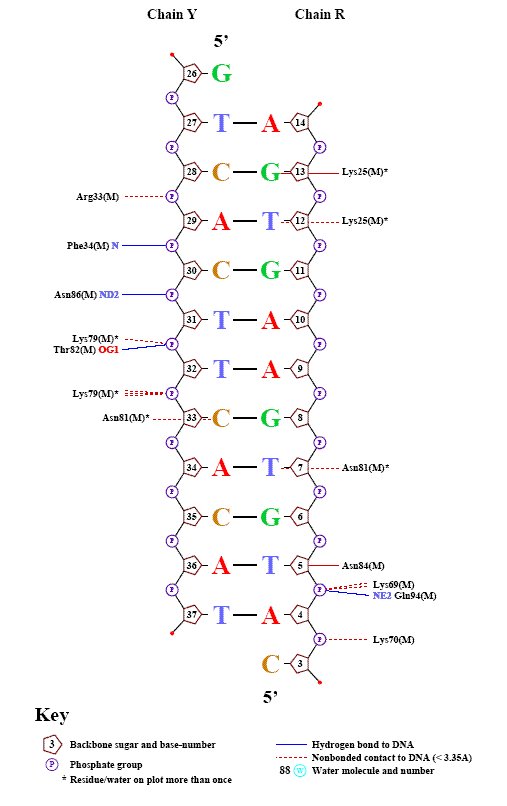
цепь Y: [26] 5` - gtсaсttсaсat - 3`[37] ||||||||||| цепь R: [14] 3` - agtgaagtgtac - 5`[3]
| Контакты атомов белка с | Полярные | Неполярные | Всего |
| остатками 2'-дезоксирибозы | 1 | 11 | 12 |
| остатками фосфорной кислоты | 8 | 14 | 22 |
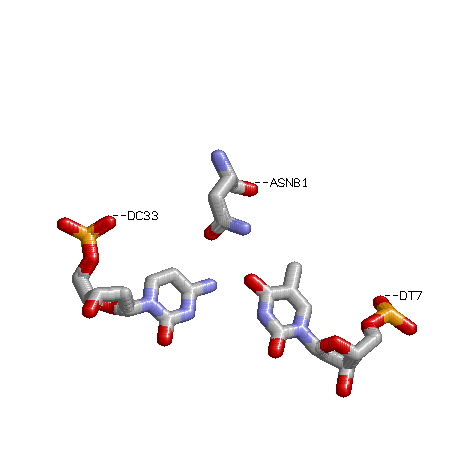
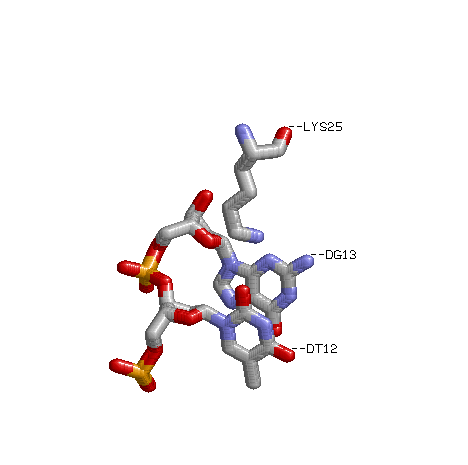
| остатками азотистых оснований со стороны большой бороздки | 2 | 5 | 7 |
| остатками азотистых оснований со стороны малой бороздки | 1 | 0 | 1 |


Доменная структура белка

2009 ©