Исследование ДНК-белковых взаимодействий в структуре комплекса регуляторного белка PHO4 с молекулой ДНК
- Краткое описание структуры в файле
1A0A.pdb
В файле приведены координаты атомов следующих молекул
1) две цепи ДНК -(фрагмент - UPSTREAM ACTIVATION SITE)
2) белок (позитивный регулятор фосфатной системы PHO4), фрагмент - связывающий ДНК домен
Организм - Saccaromyces cerevisiae (Пекарские дрожжи)
Для исследования были выбраны цепи ДНК со следующей последовательностью:
цепь C [1] 5' - CTCACACGTGGGACTAG - 3' [17] ||||||||||||||||| цепь D [17] 3' - GAGTGTGCACCCTGATC - 5' [1], - Функции белка, структура которого представлена в файле 1A0A.pdb В соответствующем документе UniProt описаны функции белка: активатор транскрипции, который регулирует образование ингибируемой фосфатазы в условиях дефицита фосфатов. Связывается с активирующей последовательностью в начале гена (upstream activating sequence - UAS) нескольких фосфатаз, включая гены PHO. Ингибируется комплексом циклина с циклинзависимой киназой при избытке фосфатов. Этот белок связывается с ДНК как гомодимер.
- Исследование структуры ДНК
Сначала преобразуем файл в старый формат с помошью программы remediator
remediator --pdb --old 1A0A.pbb > 1A0A_old.pdb
и с помошью RasMol вырежем только ДНК в отдельный файл DNA.PDB, но и с ним не все просто - все нуклеотиды обозначены двумя буквами - DA, DT и так далее. Чтобы использовать find_pair, удалим все буквы D в редакторе Far.
Теперь можно использовать команду:
find_pair -t DNA.PDB stdout | analyze
Мы определили:
Тип формы ДНК - правозакрученная спираль:
This is a right-handed nucleic acid structure
Cредние значения торсионных углов для внутренних нуклеотидов (кроме краевых). Исследуем и сравним их с помошью средств Excel. Получим таблицы и диаграммы. Файл в формате excel - angles.xls Найдем самый "кривой" нуклеотид со значениями торсионных углов, наиболее отклоняющимися от средних. Это G-11.Кроме этого сильно изменены нуклеотиды под номерами 2, 4, и 12. На рисунке они отмечены красным цветом на зеленой спирали ДНК. Видно, как альфа-спирали белка "вклиниваются" между цепями ДНК, заставляя молекулу изгибаться в соответствующих местах. Тем не менее, в моем случае нельзя сказать, что белок сильно изменяет конформацию ДНК, скорее слегка корректирует ее для успешного взаимодействия.
- Исследование природы ДНК-белковых контактов
Для исследования ДНК-белковых контактов используем скрипт my_dna.def,
в котором определятся необходимые множества атомов. Cкрипт работает со старым форматом - с
файлом 1A0A_old.pdb. При выполнении
скрипт последовательно показывает атомы, взаимодействующие с остатками
фосфорной кислоты (множества pa_pol и pa_npol), остатками дезоксирибозы
(множество dr) , с большой и малой бороздками. Полярные взаимодействия - красные
, неполярные - оранжевые. Замечу, что взаимодействий в целом не очень много,
и нет никаких взаимодействий с малой бороздкой, что вполне очевидно и так
(см картинку выше). Количество взаимодействий занесем в таблицу:
Таблица. Контакты разного типа в комплексе 1А0А.pdb
Контакты атомов белка с Полярные Неполярные Всего остатками 2'-дезоксирибозы 6 17 23 остатками фосфорной кислоты 16 24 40 остатками азотистых оснований со стороны большой бороздки 8 8 16 остатками азотистых оснований со стороны малой бороздки 0 0 0 - Получение популярной схемы ДНК-белковых контактов с помощью nucplot
Используем старый формат PDB
nucplot 1A0A_old.pdb
Получили картинку
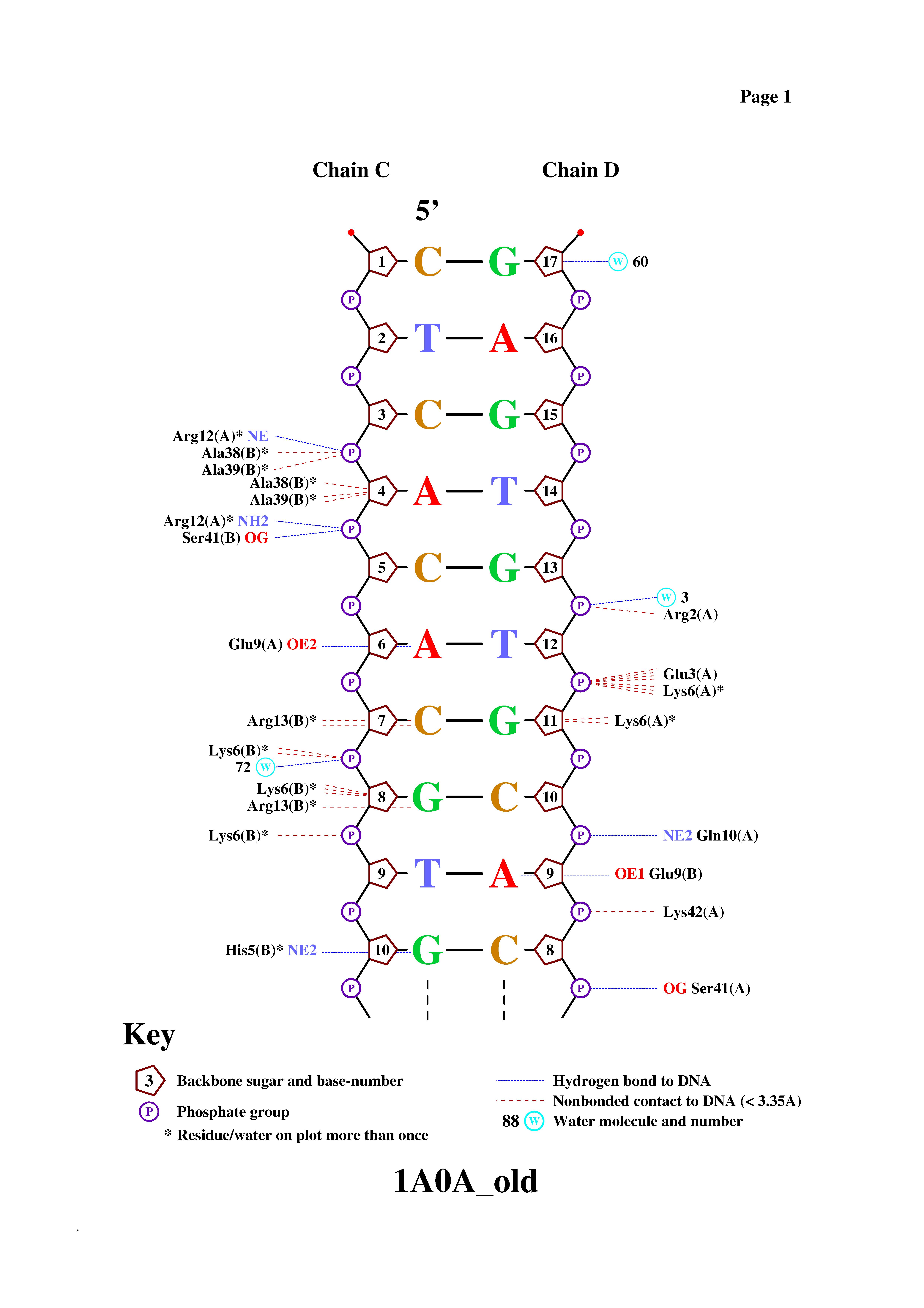
Сравнивая эти контакты с теми, которые мы обнаружили с помощью RasMol, можно заметить, что хотя число контактов отличается (мы с программой по-разному понимаем критерии взаимодействия) общие тенденции сохраняются, то есть мало контактов с основаниями, больше с остовом (с остатками фосфорной кислоты больше, чем с сахаром). Если сравнить взаимодействия белка с основаниями, то в обоих случаях связи предполагаются между одними и теми же АК и нуклеотидами. Из значимых различий отмечу, что nucplot, в отличие от меня, учел три молекулы воды (2 взаимодействуют с фосфорной кислотой и одна с сахаром).
- Возможный распознающий контакт
Аминокислоты Arg13 (с нуклеотидами С7 и G8), GLU9 с А9 и His5 с G10. Эти контакты можно считать распознающими,
потому что они во-первых локализованы все внутри большой бороздки, во-вторых, остальные
аминокислоты контактируют не с основаниями, а с остовом (то есть взаимодействие
неспецифично для данной последовательности нуклеотидов).
На картинке темно-синим показан фрагмент остова белка, голубым - упомянутые аминокислоты,
красным - атомы, контактирующие с ДНК на растоянии меньше 3х Ангстрем (еще одно
доказательство в пользу распознающего контакта - очень тесное взаимодействие - значит
достаточно прочные связи.)
- Характеристика ДНК-связывающего домена P07270 (PHO4_YEAST)
Исследуемый белок содержит классический домен - Basic helix-loop-helix, домен связывания ДНК у эукариот.
Этот домен характерен для многих белков - факторов транскрипции и высоко консервативен.
Этот домен обеспечивает специфическую димеризацию и облегчает переход неактивных мономеров
к активирующим транскрипцию димерам на соответствующих стадиях развития.
Схема из Pfam
описание из InterPro - IPR001092 Basic helix-loop-helix dimerisation region bHLH
На пространственной структуре это выглядит так (домен - зеленый) :