Совмещение структур
Поиск структурных гомологов
Были найдены гомологи для E цепи белка 1RDQ в PDBeFold.
Белки:
1rdq - мутантная мышиная цАМФ-зависимая протеинкиназа (взятая изначально);
1rek - другой мутант мышиной цАМФ-зависимой протеинкиназы;
1ctp - вепрева цАМФ-зависимая протеинкиназа.
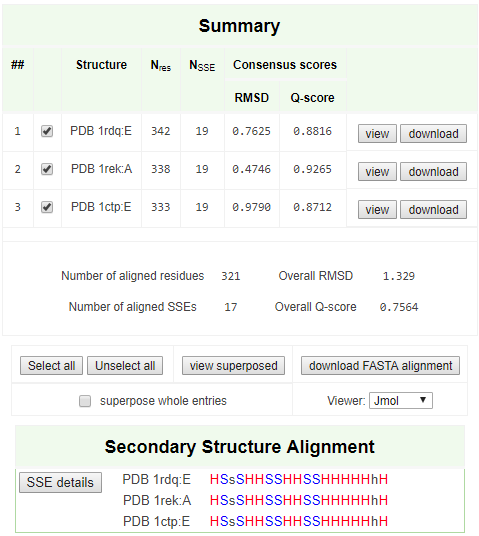
Рис 1. Гомологичные цАМФ-зависимые протеинкиназы.
Файлы со структурным выравниванием: FASTA, PDB
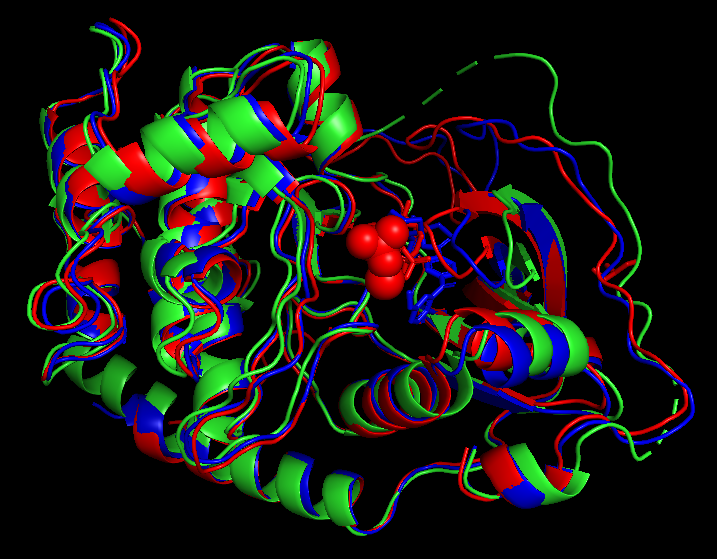
Рис 2. Совмещение структур: зеленым - вепрева структура, синим и красным - мышиные.
Построил выравнивание с помощью Muscle и сравнил его со структурным.
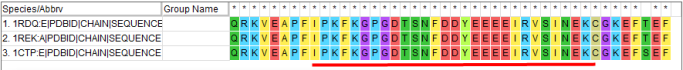

Рис 3. Участок различия двух выравниваний: сверху - Muscle, снизу - структурное; красной линией обозначен отличающийся участок.
Эти выравнивания имеют одно крупное отличие - ближе к концу. Во первых, в структурном выравнивании есть участок (у самого начала красной линии), где 315, 316 и 317 остатки вепревой последовательности смещены на один остаток относительно мышиных, а дальше есть невыровненный участок. В данном случае, это не ошибка выравнивания и на совмещении видно, что в этом месте петля в вепревой последовательности не совмещается с мышиными.
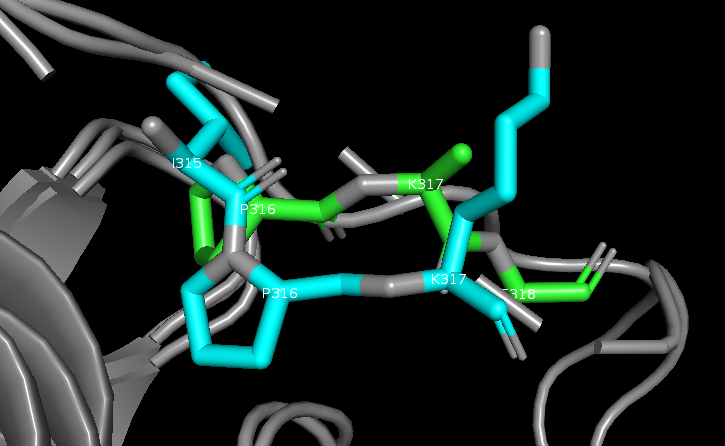
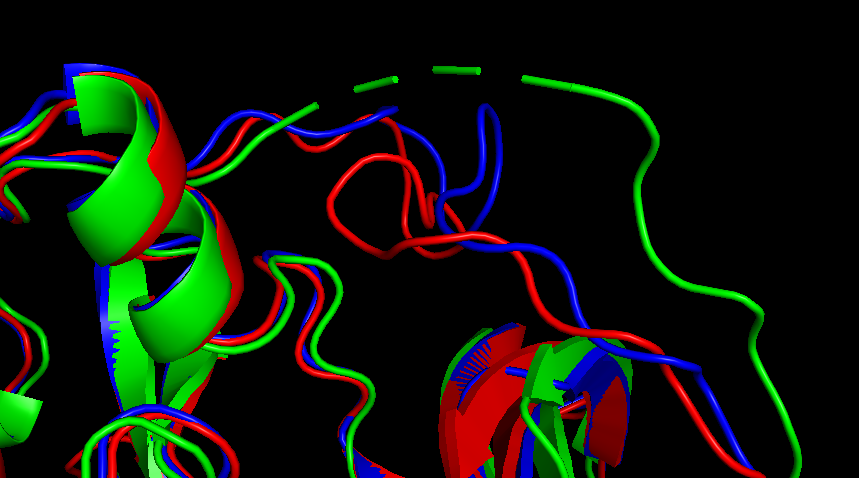
Рис 4. Иллюстрация различий: сверху - 315, 316, 317 остатки вепревой последовательности, смещенные относительно мышиных, снизу - петля вепревой последовательности, не выровненная с мышиными.
Совмещение по заданному выравниванию
Домены Т-клеточного рецептора:
α-цепь - 1qrn, region d:118-206; (выделил в объект obj02)
β-цепь - 2esv, region e:119-247. (выделил в объект obj01)
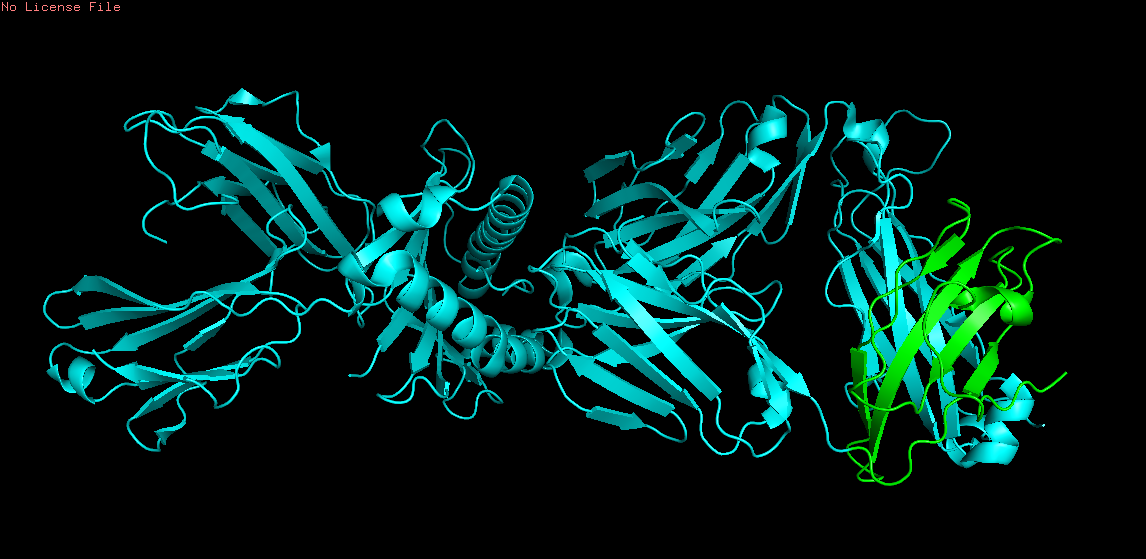
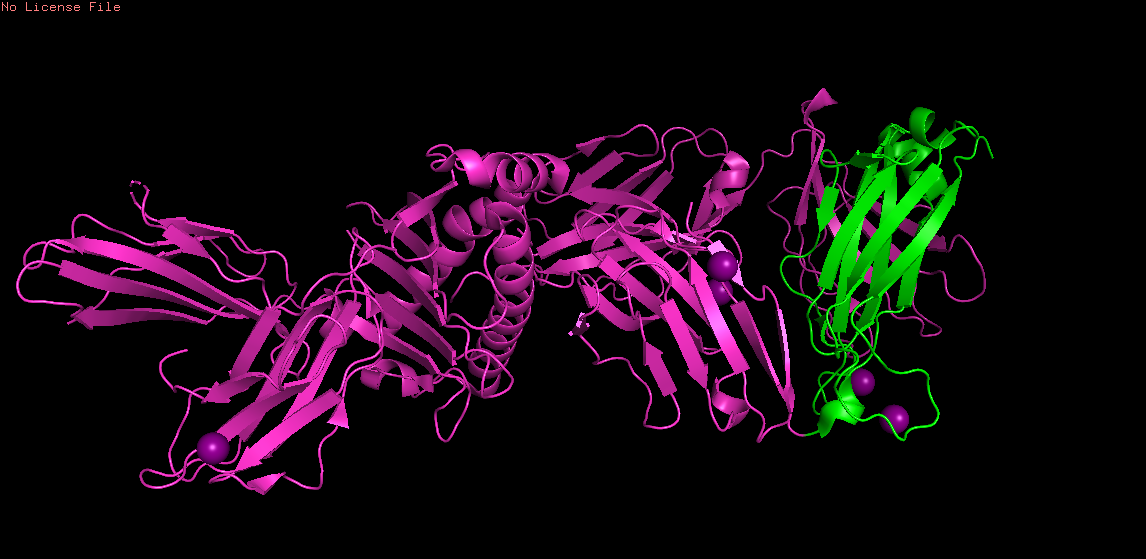
Рис 5. Структуры 1qrn (сверху) и 2esv (снизу). Обозначенные выше регионы выделены зеленым.
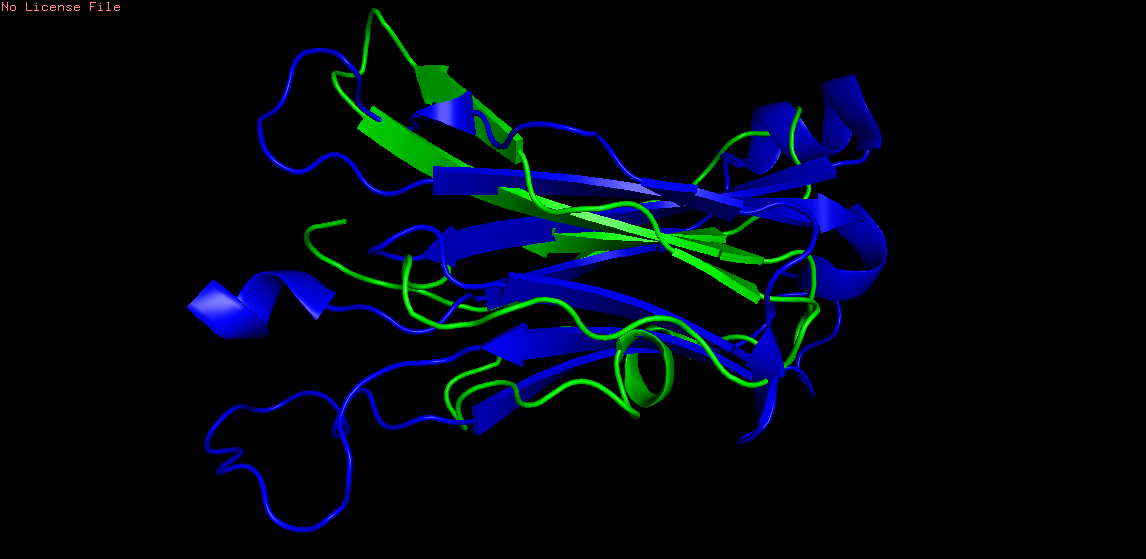
Рис 6. Совмещение регионов с помощью align в PyMol: зеленый - из альфа цепи, синий - из бета.
В SheeP построил карты:
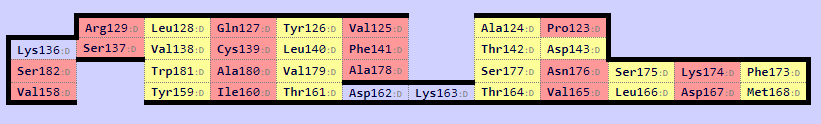
Рис 7а. Карта альфа цепи; Cys139 - цистеин, образующий дисульфидную связь.
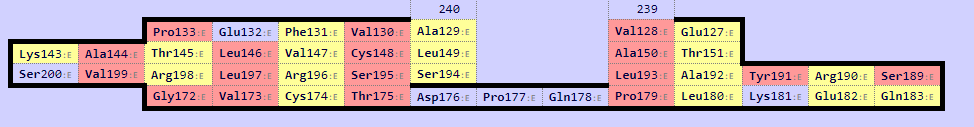
Рис 7б. Карта листа бета цепи, который соответствует листу альфа цепи; Cys148 образует дисульфидную связь.
Команда pair_fit для совмещения структур:
select alpha, obj02 and resi 126-128+138-140+179-181 and name CA
select beta, obj01 and resi 129-131+147-149+194-196 and name CA
pair_fit alpha, beta
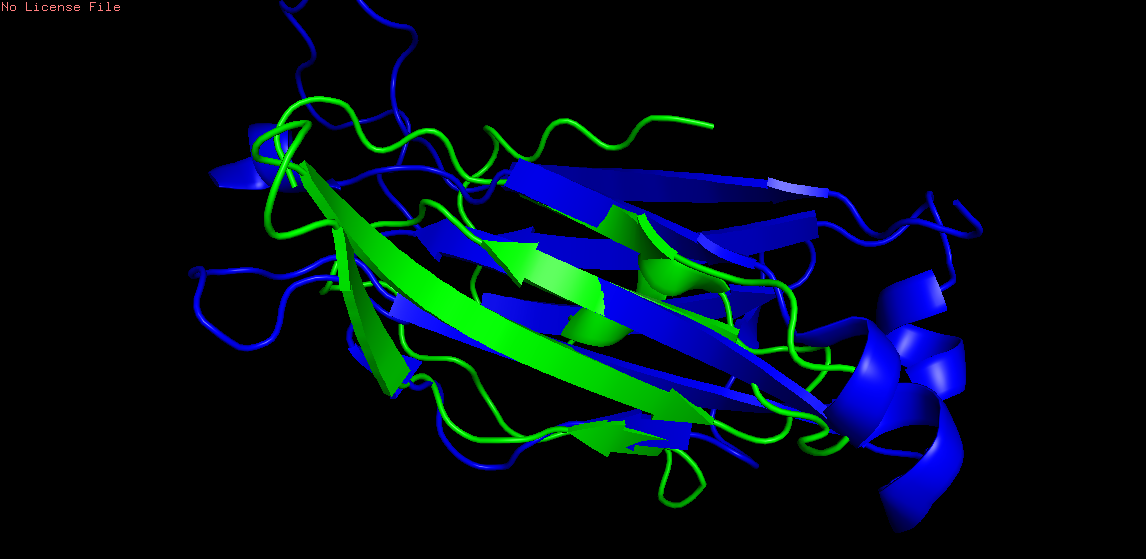
Рис 8. Выравнивание с помощью pair_fit. RMS = 0.448
Совмещение получилось довольно неплохим. Кроме того, что выбранные тяжи совместились, есть соответствия между другими элементами альфа и бета цепей (петли, идущие от тяжей частично совпадают), но не знаю, можно ли эти совмещения считать значительными, поскольку набор вторичных структур в этих цепях разный: там, где в цепи альфа просто петля, то в цепи бета петля с альфа-спиралью или там, где в цепи бета лежат бета тяжи, в цепи альфа - альфа спираль. Но при разлячиях в структурах, их расположения довольно неплохо совпадают. В любом случае, совмещение получилось намного качественнее, чем в случае исполозования команды align.
