EMBOSS
Упражнения:
1. C помощью команды "seqret *.fasta all.fasta" объединил все файлы (1,2,3) формата fasta в один.
2. C помощью команды "seqretsplit all.fasta" разделил полученный в 1 упражнении файл на три отдельных (1, 2, 3), которым программа дала имена последовательностей.
4. Взял ген ribosomal protein S4 (fasta) мха из 7 практикума. Белок
5. Взял тот же ген ribosomal protein S4 (fasta). Результат

6. Взял выравнивание с прошлого семестра (fasta). Результат

Задания
Организм – архея из 1-го семестра Vulcanisaeta moutnovskia 768-28
getorf -table 11 -minsize 180 -find 0 NC_015151.1.fasta output.fasta ## Получение открытых рамок, параметры: таблица генетического кода, минимальная длина открытой рамки, трансляция открытых рамок от стоп кодона до стоп кодона на выход
infoseq -only -name -length -sprotein1 -description output.fasta > output.txt ## имя рамки, длина трансляции, описание с координатами
Таблица открытых рамок: one.xlsx
Таблица анотированных генов: anot.xlsx
Сводная таблица: compl.xlsx
Что я увидел:
1) Открытых рамок гораздо больше, чем анотированных генов. Видимо, не каждая открытая рамка гарантирует ген.
2) Аннотации длиной менее 60 а.о. не имеют соответсвующих ORF-ов, так как мы искали только длинные открытые рамки (больше 180 п.н.)
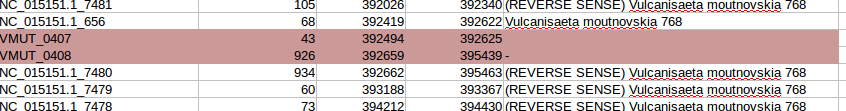
Рис.1
3) ORF-ы начинаются раньше, чем соответствующие им аннотации (от двух до пары десятков нуклеотидов)

Рис.2
4) Перекрывание антипараллельных рамок (более 150 п.н.)

Рис.3
5) Не для всех белков были найдены открытые рамки. Возможно это из-за того, что ORF-ы были определены от стоп- до стоп-кодонов и программа не обращала внимания на старт кодоны, с которых действительно начинаются последовательности кодирующие эти белки.

Рис.4
