На страницу шестого семестра
Докинг низкомолекулярных лигандов в структуру белка
Задание 1
1) В банке pdb нашли SMILES аннотацию для NAG.2)C помощью obgen построим 3D структуру этого сахара в pdb формате.
3)Скриптом prepare_ligand4.py из пакета Autodock tools создали pdbqt файл лиганда.obgen nag.smi > nag.mol babel -imol nag.mol -opdb nag.pdb
4)Так же, скриптом prepare_receptor4.py из пакета Autodock tools создали pdbqt файл белка.prepare_ligand4.py -l nag.pdb
Проведем первый докинг с фиксированными аминокислотами белка и подвижным лигандом. Запустим командуprepare_receptor4.py -r seq.B99990001.pdb cp seq.B99990001.pdbqt prot.pdbqt
vina --config vina.cfg --receptor seq5.pdbqt --ligand nag.pdbqt --out nag_prot.pdbqt --log nag_prot.log
На выходе имеем два файла nag_prot.pdbqt, содержащий информацию о положениях лиганда, и nag_prot.log, содержащий информацию о энергиях каждой модели. Все состояния на одной картинке.
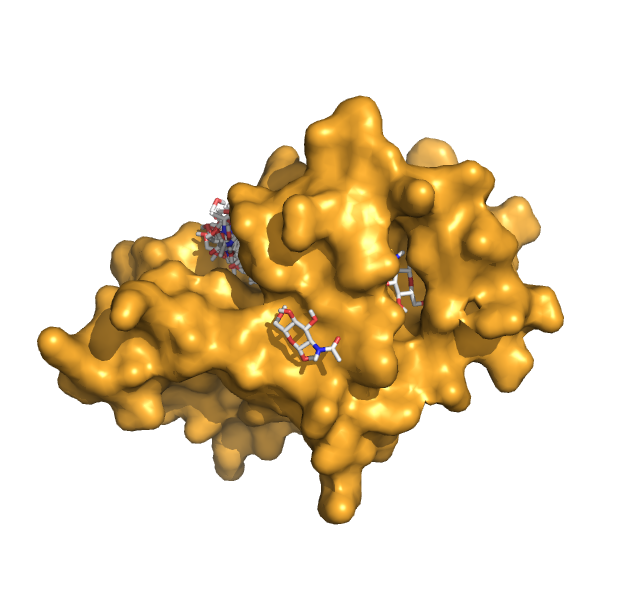
Энергии 3ёх лучших расположений и геометрическая разницу между ними:
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -6.5 0.000 0.000
2 -6.3 2.045 3.771
3 -5.6 14.708 16.058
Докинг, при учёте подвижности некоторых боковых радикалов белка
Сначала разобьем белок на две части, подвижную и неподвижную. Для подвижной части выберем 3 аминокислоты которые использовали в прошлом задании для позиционирования лиганда- LEU70,GLU136,LYS138.
python /usr/share/pyshared/AutoDockTools/Utilities24/prepare_flexreceptor4.py -r re.pdbqt -s LEU70_GLU136_LYS138
vina --config vina.cfg --receptor prot_rigid.pdbqt
--flex prot_flex.pdbqt --ligand nag.pdbqt --out vina_prot_flex.pdbqt --log vina_prot_flex.log
Результаты докинга, энергии 3ёх лучших расположений и геометрическая разница между ними. Время докинга больше, чем у докинга без подвижных радикалов.
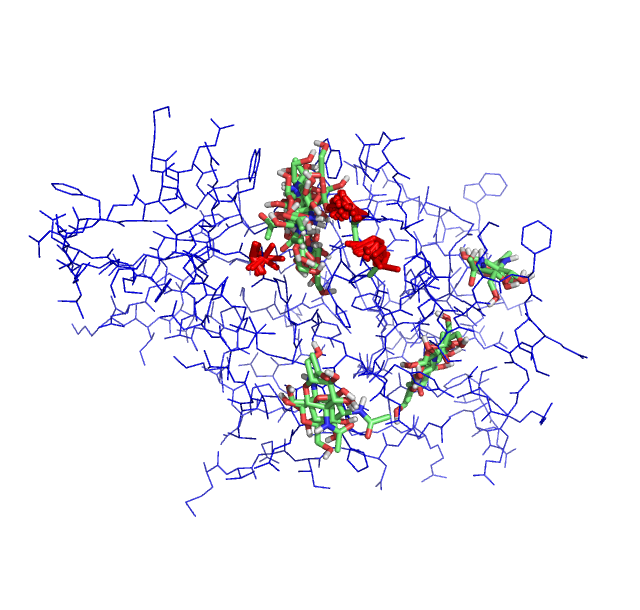
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -6.4 0.000 0.000
2 -6.2 1.579 2.985
3 -6.0 1.531 3.024
На изображени подвижные остатки изображены красным. Разброс в позиционировании лиганда увеличился.
Выводы о докинге
Может ли докинг расположить лиганд наиболее близким образом к тому, что было получено в моделировании. В принципе, среди множества вариантов, предлагаемых докингом, можно подобрать и такие, которые будут отражать ситуацию , полученную при помощи гомологичного моделирования. На картинке совокупность результатов. Синее- по моделированию, зелёное -по докнгу с подвижными радикалами, расскраска по атомам- обычный докинг.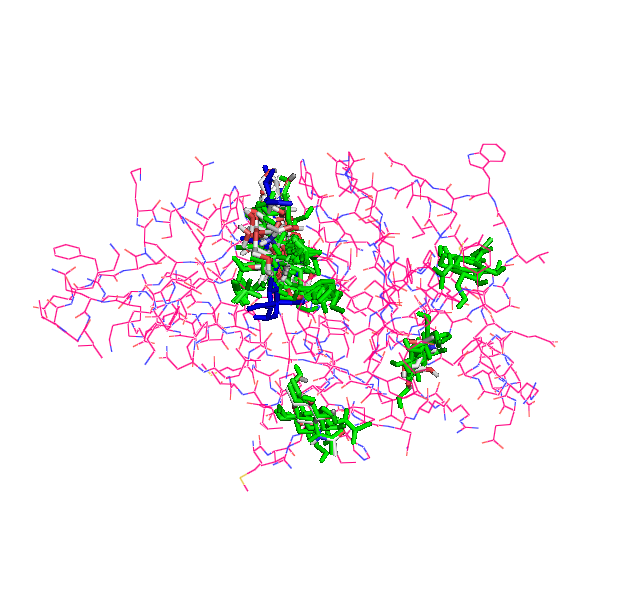
NAG, заменяем метильный радикал.
Сравнение энергий для обычного докинга с лигандом где метильный радикал этой группы будет заменён на :OH mode | affinity | dist from best mode | (kcal/mol) | rmsd l.b.| rmsd u.b. -----+------------+----------+---------- 1 -5.5 0.000 0.000 2 -5.4 1.557 3.085 3 -5.2 1.734 3.055 NH2 mode | affinity | dist from best mode | (kcal/mol) | rmsd l.b.| rmsd u.b. -----+------------+----------+---------- 1 -5.7 0.000 0.000 2 -5.5 1.755 3.081 3 -5.3 2.976 4.855 Ph mode | affinity | dist from best mode | (kcal/mol) | rmsd l.b.| rmsd u.b. -----+------------+----------+---------- 1 -7.1 0.000 0.000 2 -7.0 3.039 7.198 3 -6.4 2.152 3.595
И для докинга с подвижными радикалами.OH mode | affinity | dist from best mode | (kcal/mol) | rmsd l.b.| rmsd u.b. -----+------------+----------+---------- 1 -5.7 0.000 0.000 2 -5.7 1.332 2.413 3 -5.4 0.890 2.185 NH2 mode | affinity | dist from best mode | (kcal/mol) | rmsd l.b.| rmsd u.b. -----+------------+----------+---------- 1 -5.9 0.000 0.000 2 -5.8 1.349 2.111 3 -5.5 1.950 3.809 Ph mode | affinity | dist from best mode | (kcal/mol) | rmsd l.b.| rmsd u.b. -----+------------+----------+---------- 1 -7.6 0.000 0.000 2 -7.6 2.413 5.749 3 -6.7 1.398 2.470