|
Главная
I Семестр
II Семестр
III Семестр
IV Семестр
V Семестр
Проекты
Обратная Связь
|
Введение в PyMol
Работа с записью 1KMY банка PDB
C сайта RCSB был получен PDB-файл записи
1KMY.
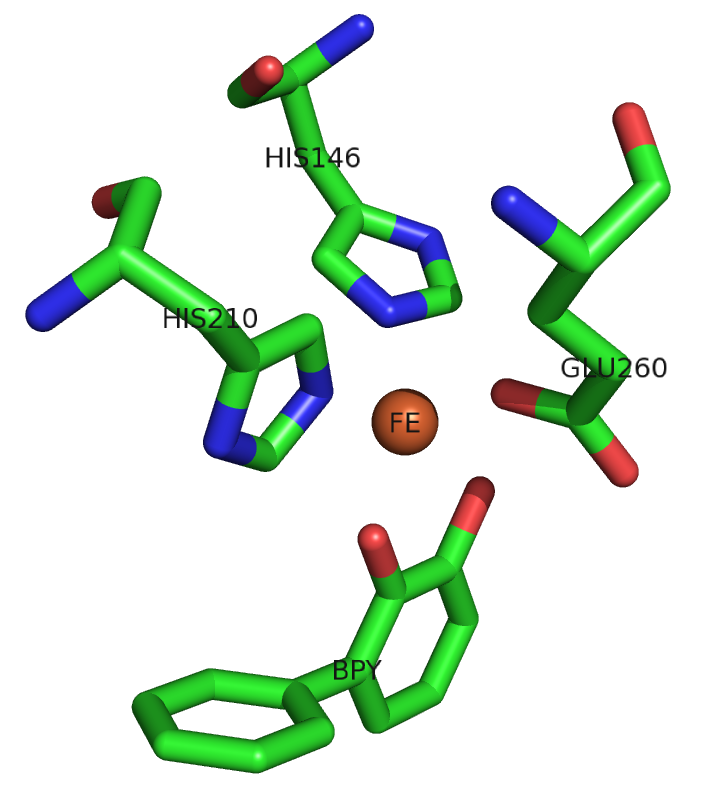
По полю HETNAM было определено, что в структуре присутствует ион двухвалентного железа.
C помощью PyMOL было сохранено изображение иона двухвалентного железа, включая остатки и
лиганды, связанные с ионом координационными связями (то есть имеющие атомы,
которые находятся на растоянии не более 3.5 ангстрем от данного иона).
Среди них оказались два остатка гистидина (His146 и His210),
остаток глутаминовой кислоты (Glu260), лиганд бифенил-2,3-диол (BPY).
Работа с записью 1DLP банка PDB
Получен файл 1DLP.
Структура расшифрована в 1999 году. Разрешение 3.30 ангстрем.
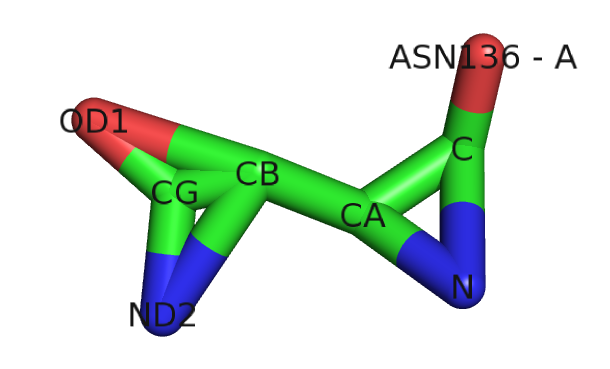
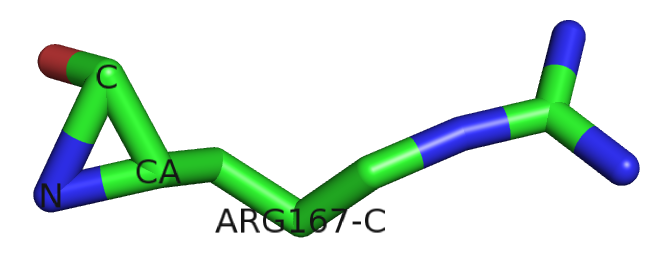
Запись содержит шесть идентичные полипептидные цепи. Остаток 136 цепи А это аспарагин, а остаток 167 цепи С - аргинин, в глаза бросается то, что
атомы этих аминокислот соединены не правильно. Посмотрев в поле REMARK, я обнаружил следующее:
REMARK 480 ZERO OCCUPANCY ATOM
REMARK 480 THE FOLLOWING RESIDUES HAVE ATOMS MODELED WITH ZERO
REMARK 480 OCCUPANCY. THE LOCATION AND PROPERTIES OF THESE ATOMS
REMARK 480 MAY NOT BE RELIABLE. (M=MODEL NUMBER; RES=RESIDUE NAME;
REMARK 480 C=CHAIN IDENTIFIER; SSEQ=SEQUENCE NUMBER; I=INSERTION CODE):
REMARK 480 M RES C SSEQI ATOMS
REMARK 480 ASN A 136 CG OD1 ND2
REMARK 480 ARG C 167 CB CG CD NE CZ NH1 NH2
Это подверждение того, что у этих остатков местоположение атомов может не соответствовать реальности.
Вот почему мы видим не правильное соединение атомов у этих остатков.
| Asn136 - A |
Arg167 - C |
|
|
Работа с записью 2B5A банка PDB
Получен файл 2B5A.
Структура расшифрована в 2005 году. Разрешение 1.54 ангстрем.
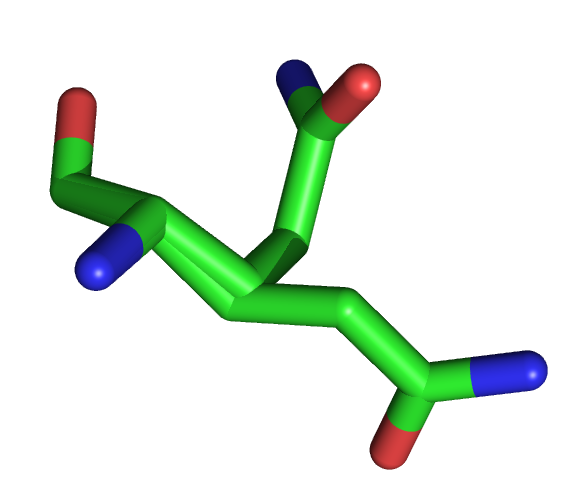
Запись содержит четыре идентичные полипептидные цепи. Остатками с номерами 21
оказались остатки глутамина. Кроме того некоторые атомы остатка в цепи C
указаны в двух экземплярах с разными координатами. Возможно, это говорит о том,
что этот остаток имеет две конформации. В поле REMARK не нашлось информации об
этой странности и вообще о данных остатках.
| Gln21 - C |
|
Работа с записью 7GPB банка PDB
Получен файл 7GPB.
Структура расшифрована в 1990 году. Разрешение 2.90 ангстрем.
Запись содержит четыре идентичные полипептидные цепи. Остатками с номерами 67
оказались остатки триптофана. Триптофан должен быть плоским, а в это файле это не так. Более того у
триптофана цепи D пятичленный цикл практически перпендикулярен шестичленному циклу.
В поле REMARK не нашлось информации об этой странности.
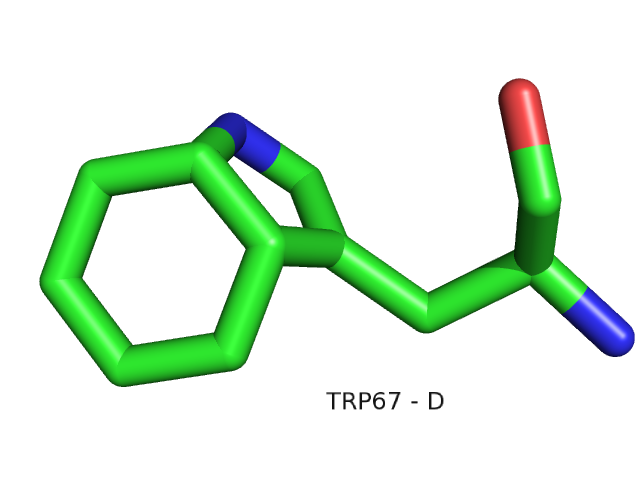
Создание скрипта для PyMOL
Создан скрипт для PyMOL, результат работы которого - изображение в графическом окне,
иллюстрирующее отчёт по предыдущему упражнению (Работа с записью 7GPB банка PDB).
Скрипт: trp67.pml
load 7GPB.pdb
hide all
select some, i. 67 & c. d
show sticks, some
orient some
util.cbag some
label i. 67 & c. d & n. cb, "%s%s - %s" % (resn, resi, chain)
bg_color white
set label_color, black
set label_size, 16
set label_position,(0,-1.5,1.75)
set depth_cue, 0
(*) Первые впечатления от PyMOL
Преимущества PyMol по сравнению с RasMol:
- Возможность создавать изображения высокого качества
- Возможность писать команды в графическом окне
- Возможность вставлять текст в командную строку
- Наличие панели для выполнения при помощи мыши различных операций с заданными там множествами
- Поддержка системы плагинов
Недостаток PyMol по сравнению с RasMol:
- Некоторая сложность в интерфейсе
|




