|
Главная
I Семестр
II Семестр
III Семестр
IV Семестр
Проекты
Обратная Связь
|
Особенности работы с нуклеотидными последовательностями. Анализ деревьев, содержащих паралоги.
1. Построение дерева по нуклеотидным последовательностям
Из записей EMBL были извлечены последовательности одной из 16S рРНК, соответствующих геномам отобранных ранее бактерий.
| Название |
Мнемоника |
| Rhizobium etli |
RHIEC |
| Ralstonia pickettii |
RALPJ |
| Neisseria meningitidis |
NEIMA |
| Yersinia pestis |
YERPE |
| Vibrio cholerae |
VIBCH |
| Haemophilus influenzae |
HAEIN |
| Pasteurella multocida |
PASMU |
| Proteus mirabilis |
PROMH |
Все последовательности были собраны в файл (my_bac.fasta).
После чего выровнены программой muscle, в результате чего получили следующее выравнивание
(mybac_muscle.fasta).
Затем выравнивание было подано на вход программам, реконструирующим филогенетическое дерево.
Сперва воспользовался программами fdnaml и fdnapars, которые на вход принимают выранивание:
fdnaml
+PASMU
+---1
| +HAEIN
+---2
| | +-YERPE
| | +--4
| +--3 +-PROMH
| |
| +--VIBCH
|
| +---NEIMA
5--6
| +---RALPJ
|
+-------RHIEC
fdnapars
+---NEIMA
+----6
| +----RALPJ
|
| +-YERPE
| +--5
| +--4 +-PROMH
| | |
1--3 +--VIBCH
| |
| | +-HAEIN
| +---2
| +PASMU
|
+--------RHIEC
С помощью программы fprotdist была получена матрица расстояний для этих последовательностей:
RHIEC 0.000000 0.242139 0.249970 0.224768 0.233155 0.231912 0.243445 0.222719
PASMU 0.242139 0.000000 0.055319 0.159438 0.145568 0.132628 0.216906 0.238637
HAEIN 0.249970 0.055319 0.000000 0.154623 0.136530 0.144546 0.221192 0.249752
VIBCH 0.224768 0.159438 0.154623 0.000000 0.108559 0.097215 0.226786 0.218918
PROMH 0.233155 0.145568 0.136530 0.108559 0.000000 0.068963 0.222052 0.217404
YERPE 0.231912 0.132628 0.144546 0.097215 0.068963 0.000000 0.217778 0.231515
RALPJ 0.243445 0.216906 0.221192 0.226786 0.222052 0.217778 0.000000 0.144074
NEIMA 0.222719 0.238637 0.249752 0.218918 0.217404 0.231515 0.144074 0.000000
Реконструировал деревья программами fneighbor, ffitch и fkitsch,
которые на вход принимают матрицу расстояний:
fneighbor (алгоритм Neighbor-Joining)
+----RALPJ
+-1
! +----NEIMA
!
! +PASMU
! +--2
! ! +HAEIN
3--4
! ! +--VIBCH
! +-5
! ! +--PROMH
! +-6
! +-YERPE
!
+------RHIEC
ffitch
+----NEIMA
+-6
! +----RALPJ
!
! +-YERPE
! +-4
! +-3 +--PROMH
! ! !
5--2 +--VIBCH
! !
! ! +PASMU
! +--1
! +HAEIN
!
+------RHIEC
fkitsch (дерево укоренено)
+---NEIMA
+--7
! +---RALPJ
!
+-6 +-YERPE
! ! +-5
! ! +-4 +-PROMH
! ! ! !
! +--3 +--VIBCH
--1 !
! ! +-HAEIN
! +-2
! +-PASMU
!
+------RHIEC
Все неукорененные деревья полученные разными програмами совпадают друг с другом по множеству нетривиальных ветвей,
но отличаются от правильного дерева (изображение).
У полученных деревьев есть нетривиальная ветвь - ({RHIEC, NEIMA, RALPJ, PASMU, HAEIN} против {VIBCH, PROMH, YERPE})в связи с этим
отсутсвует нетривиальная ветвь - ({RHIEC, NEIMA, RALPJ, VIBCH} против {PASMU, HAEIN, PROMH, YERPE}).
Нуклеотидная последовательность 16S rRNA VIBCH ближе к PROMH и YERPE, а белковая последовательность
VIBCH дальше от PROMH и YERPE, чем PASMU, HAEIN. Реконструкция филогенетических деревьев по нуклеотидным последовательностям хуже,
чем по белковым, в связи с вырожденностью генетического кода.
2. Построение и анализ дерева, содержащего паралоги
Сперва найдем последовательность белка FTSH_ECOLI:
seqret sw:ftsh_ecoli
Затем создадим базу данных:
formatdb -i proteo.fasta -n dbase -p T
Проведен поиск гомологов белка FTSH_ECOLI программой blastp:
blastall -p blastp -d dbase -i ftsh_ecoli.fasta -e 0.0001 -o mybac.out -m 8
Среди них были отобраны находки, соответствующие отобранным мною бактериям (для которых были построены предыдущие деревья).
Последовательности находок были выравнены программой muscle, затем получил матрица расстояний для этих последовательностей,
которую подал на вход программе fneighbor. В результате чего получил следующее дерево:
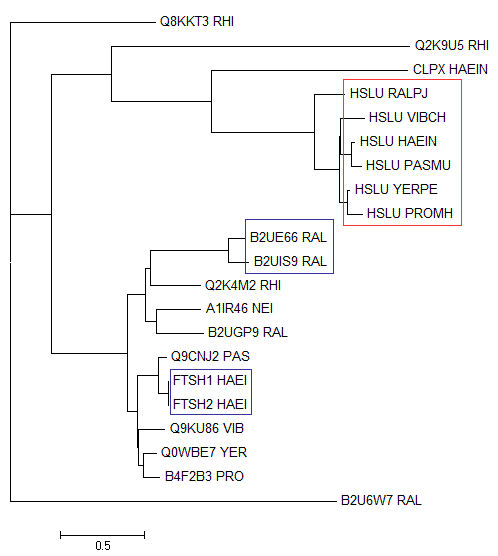
Будем считать реконструкцию дерева верной.
Ортологи - два гомологичных белка из разных организмов, разделение общего предка которых произошло
в результате видообразования. Например:
- HSLU_HAEIN и HSLU_PASMU
- HSLU_YERPE и HSLU_PROMH
Паралоги - два гомологичных белка из одного организма. Например:
- B2UE66_RALPJ и B2UIS9_RALPJ
- FTSH1_HAEIN и FTSH2_HAEIN
|
