|
Главная
I Семестр
II Семестр
III Семестр
IV Семестр
Проекты
Обратная Связь
|
Построение филогенетического дерева
1. Отобранные бактерии
| Название |
Мнемоника |
| Rhizobium etli |
RHIEC |
| Ralstonia pickettii |
RALPJ |
| Neisseria meningitidis |
NEIMA |
| Yersinia pestis |
YERPE |
| Vibrio cholerae |
VIBCH |
| Haemophilus influenzae |
HAEIN |
| Pasteurella multocida |
PASMU |
| Proteus mirabilis |
PROMH |
2. Скобочная формула дерева
(RHIEC, ((RALPJ, NEIMA), (VIBCH, ((PASMU, HAEIN), (PROMH, YERPE)))));
3. Изображение дерева
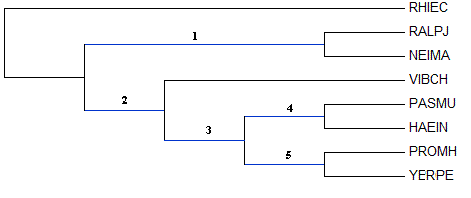
Синим выделены нетривиальные ветви дерева.
4. Нетривиальные ветви
- {RALPJ, NEIMA} против {RHIEC, VIBCH, PASMU, HAEIN, PROMH, YERPE}
- {RHIEC, RALPJ, NEIMA} против {VIBCH, PASMU, HAEIN, PROMH, YERPE}
- {RHIEC, RALPJ, NEIMA, VIBCH} против {PASMU, HAEIN, PROMH, YERPE}
- {RHIEC, RALPJ, NEIMA, VIBCH, PROMH, YERPE} против {PASMU, HAEIN}
- {RHIEC, RALPJ, NEIMA, VIBCH, PASMU, HAEIN} против {PROMH, YERPE}
Продолжение
1. Таксономия бактерий
При помощи таксономического сервиса NCBI: http://www.ncbi.nlm.nih.gov/taxonomy/;
определил, к каким таксонам относятся отобранные мною бактерии.
| Название |
Таксономия |
| Rhizobium etli |
Bacteria; Proteobacteria; Alphaproteobacteria; Rhizobiales; Rhizobiaceae;
Rhizobium/Agrobacterium group; Rhizobium |
| Ralstonia pickettii |
Bacteria; Proteobacteria; Betaproteobacteria; Burkholderiales;
Burkholderiaceae; Ralstonia |
| Neisseria meningitidis |
Bacteria; Proteobacteria; Betaproteobacteria; Neisseriales;
Neisseriaceae; Neisseria |
| Vibrio cholerae |
Bacteria; Proteobacteria; Gammaproteobacteria; Vibrionales;
Vibrionaceae; Vibrio |
| Pasteurella multocida |
Bacteria; Proteobacteria; Gammaproteobacteria; Pasteurellales;
Pasteurellaceae; Pasteurella |
| Haemophilus influenzae |
Bacteria; Proteobacteria; Gammaproteobacteria; Pasteurellales;
Pasteurellaceae; Haemophilus |
| Proteus mirabilis |
Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales;
Enterobacteriaceae; Proteus |
| Yersinia pestis |
Bacteria; Proteobacteria; Gammaproteobacteria; Enterobacteriales;
Enterobacteriaceae; Yersinia |
Далее приведу таксоны которые отделяют нетривиальные ветви (нумерацию как в предыдущем задание)
- Отделяет Betaproteobacteria
- Отделяет Gammaproteobacteria
- -
- Отделяет Pasteurellales; Pasteurellaceae
- Отделяет Enterobacteriales; Enterobacteriaceae
2. Семейство белков для реконструкции филогенитического дерева
Я взял следующее семейство белков (Фактор элонгации трансляции Ts) получил с помощью
команды seqret аминокислотные последовательности белков, выбранных мной бактерий. Затем воспользовался
программой Mafft в результате чего получил множественное выравнивание в fasta-формате
(mp_mafft.fasta)
3. Реконструкцию дерева программой fprotpars
Программа fprotpars выдала всего одно дерево.
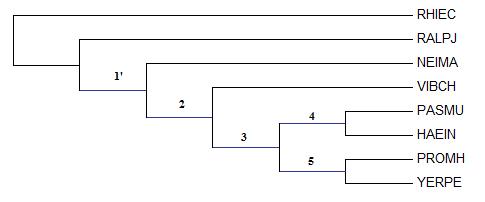
Полученное дерево имеет следующую скобочную формулу:
(RHIEC, (RALPJ, (NEIMA, (VIBCH, ((PASMU, HAEIN), (PROMH, YERPE))))));
Полученное дерево и дерево из предыдущего задание отличаются. В полученном дереве есть
нетривиальная ветвь 1' - ({RALPJ, RHIEC} против {NEIMA, VIBCH, PASMU, HAEIN, PROMH, YERPE})в связи с этим
отсутсвует нетривиальная ветвь 1 - ({RALPJ, NEIMA} против {RHIEC, VIBCH, PASMU, HAEIN, PROMH, YERPE}).
4. Оценка эволюционных расстояния между последовательностями программой fprotdist
С помощью программы fprotdist была построенна матрица расстояний по множественному выравниванию.
RHIEC 0.000000 0.952542 0.853172 0.911172 0.788978 0.809924 0.870521 0.865587
RALPJ 0.952542 0.000000 0.651425 0.866461 0.846062 0.829169 0.869665 0.859547
NEIMA 0.853172 0.651425 0.000000 0.873420 0.731265 0.763463 0.793638 0.822947
VIBCH 0.911172 0.866461 0.873420 0.000000 0.359863 0.385027 0.425615 0.426919
PASMU 0.788978 0.846062 0.731265 0.359863 0.000000 0.193014 0.291674 0.301713
HAEIN 0.809924 0.829169 0.763463 0.385027 0.193014 0.000000 0.335116 0.353217
PROMH 0.870521 0.869665 0.793638 0.425615 0.291674 0.335116 0.000000 0.189185
YERPE 0.865587 0.859547 0.822947 0.426919 0.301713 0.353217 0.189185 0.000000
Проверим данную матрицу на ультраметричность и аддитивность.
Для выполнения принципа ультраметричности необходимо, чтобы для трёх расстояний между тремя листьями два всегда равны между собой и не меньше третьего.
Возьмем VIBCH, PROMH и YERPE имеем следующие расстояния: 0.425615 (между VIBCH и PROMH), 0.426919 (между VIBCH и YERPE) и
0.189185 (между PROMH и YERPE), принцип выполняется, отклонение от равенства 0.001304.
Возьмем еще одну тройку - RALPJ, PASMU и HAEIN имеем следующие расстояния: 0.846062 (между RALPJ и PASMU), 0.829169 (между RALPJ и HAEIN) и
0.193014 (между PASMU и HAEIN), принцип выполняется, отклонение от равенства 0.016893.
Для выполнения принципа аддитивности необходимо, чтобы для четырех последовательностей A,B,C,D были три суммы расстояний 1) d(A,B) + d(C,D);
2) d(A,C) + d(B,D); 3) d(A,D) + d(B,C) причем, две равны между собой и больше третьей.
Возьмем PASMU, HAEIN, PROMH и YERPE имеем следующие расстояния : d(PASMU, HAEIN) + d(PROMH, YERPE)=0.382199,
d(PASMU, PROMH) + d(HAEIN, YERPE)=0.644891 и d(PASMU, YERPE) + d(HAEIN, PROMH)=0.636829, принцип выполняется, отклонение от равенства 0.008062.
5. Реконструкцию дерева программой fneighbor
Были получите две реконструкции дерева программой fneighbor, используя два алгоритма: UPGMA и Neighbor-Joining
Neighbor-Joining:
((RALPJ:0.35784,NEIMA:0.29358):0.11388,(VIBCH:0.23290,((PASMU:0.08202,
HAEIN:0.11100):0.03618,(PROMH:0.09062,YERPE:0.09856):0.09315):0.00624):0.20623,RHIEC:0.46326);

UPGMA:
(RHIEC:0.43228,((RALPJ:0.32571,NEIMA:0.32571):0.08707,(VIBCH:0.19968,
((PASMU:0.09651,HAEIN:0.09651):0.06371,(PROMH:0.09459,YERPE:0.09459):0.06562):0.03946):0.21310):0.01950);

Деревья полученные программой fneighbor полностью совпадают с правильным деревом и отличаются от дерева, полученного с помощью
программы fprotpars, различия в нетривиальных ветвях 1 и 1'.
|



