Чтобы оценить качетсво структуры записи 1А40 воспользуемся программой PROCHECK. Для этого на странице базы данных PDBSum со страницы структуры 1А40 нужно пройти по гиперссылке "PROCHECK".
Выдача программы в файле procheck_statistics.txt.
Ниже приведена полученная карта Рамачандрана.
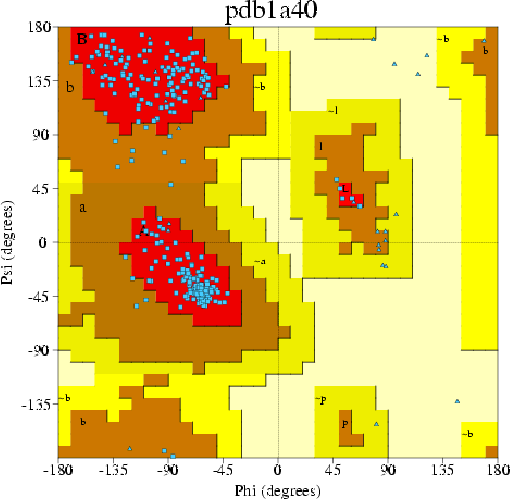
Рисунок 1. Карта Рамачандрана.
PROCHECK statistics
No. of
residues %-tage
------ ------
Most favoured regions [A,B,L] 253 93.7%
Additional allowed regions [a,b,l,p] 17 6.3%
Generously allowed regions [~a,~b,~l,~p] 0 0.0%
Disallowed regions [XX] 0 0.0%
---- ------
Non-glycine and non-proline residues 270 100.0%
End-residues (excl. Gly and Pro) 2
Glycine residues 34
Proline residues 15
----
Total number of residues 321
В данной структуре процент остатков (отличных от Gly и Pro), попавших в предпочитаемые области на карте - 93.7%
(253 из 270 остатков). Более того, все остатки (отличные от Gly и Pro) попали в разрешенные области.
Таким образом, можно сделать вывод, что структуру можно считать хорошей по этому показателю (торсионные углы).
Заходим на страницу структуры 1А40, затем по гиперссылке "Significant regions". Получаем картинку, на которой наличие пика говорит о том, что соответствующий пику аминокислотный остаток имеет значительно меньшую плотность по сравнению со средней величиной для такого типа остатков с близким разрешением.
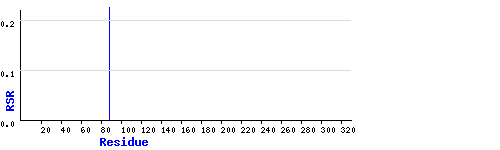
Рисунок 2. Significant Regions.
Пик на картинке соответствует лейцину 88
RSR = 0.226
Z-score пространственного R-фактора = 3.469816
Коэффициент корреляции (Real-space correlation coefficient) = 0.783
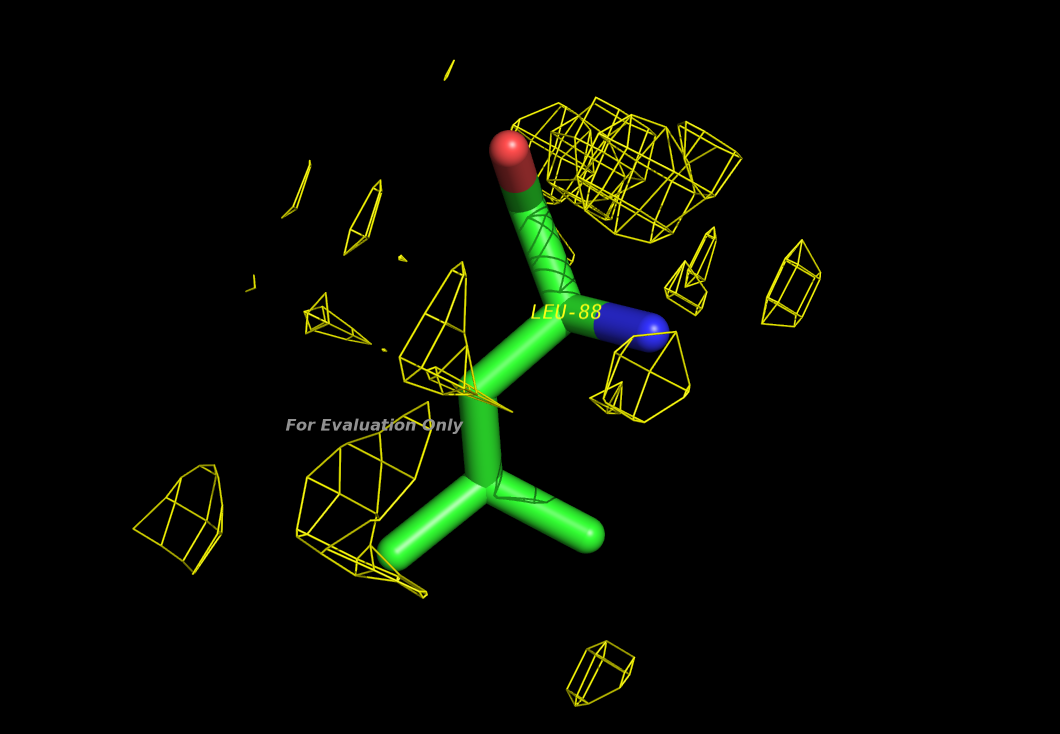
Рисунок 3. Лейцин 88
Приведенные значения сильно отличаются от допустимых для этих характеристик, поэтому достоверность такого положения этого остатка сомнительна. Рисунок 3 также показывает, что структура плохо вписана в электронную плотность.
R-values etc. From PDB header Calculated from data After conservative optimisation After full optimisation R 0.2030 0.1919 0.1745 0.2654 R-free 0.2690 0.1934 0.2060 0.2780 σR-free 0.0033 0.0036 0.0048 R-free Z-score 10.82 0.64 8.10
После оптимизации улучшилось значение для Z-score, при этом ухудшились остальные показатели.
WHAT_CHECK validation Original PDB entry Conservatively optimised Fully optimised 1st generation packing quality1 -0.059 0.058 0.052 2nd generation packing quality1 -1.429 -1.514 -1.301 Ramachandran plot appearance1 -0.599 -0.456 -0.571 Chi-1/Chi-2 rotamer normality1 -1.891 -1.365 -0.300 Backbone conformation1 0.060 0.088 0.100 Bond length RMS Z-score2 0.589 0.839 0.788 Bond angle RMS Z-score2 0.741 0.871 0.802 Total number of bumps3 24 34 27 Unsatisfied H-bond donors/acceptors3 11 14 12 1 Higher is better 2 Should be lower than 1.000 3 Fewer is better
Улучшились показатели для карты Рамачандрана, 1st generation и 2nd generation packing quality, конформации остова. Ухудшились все остальные показатели.
Для остатка лейцин 88 в PyMOL была создана картинка 4.
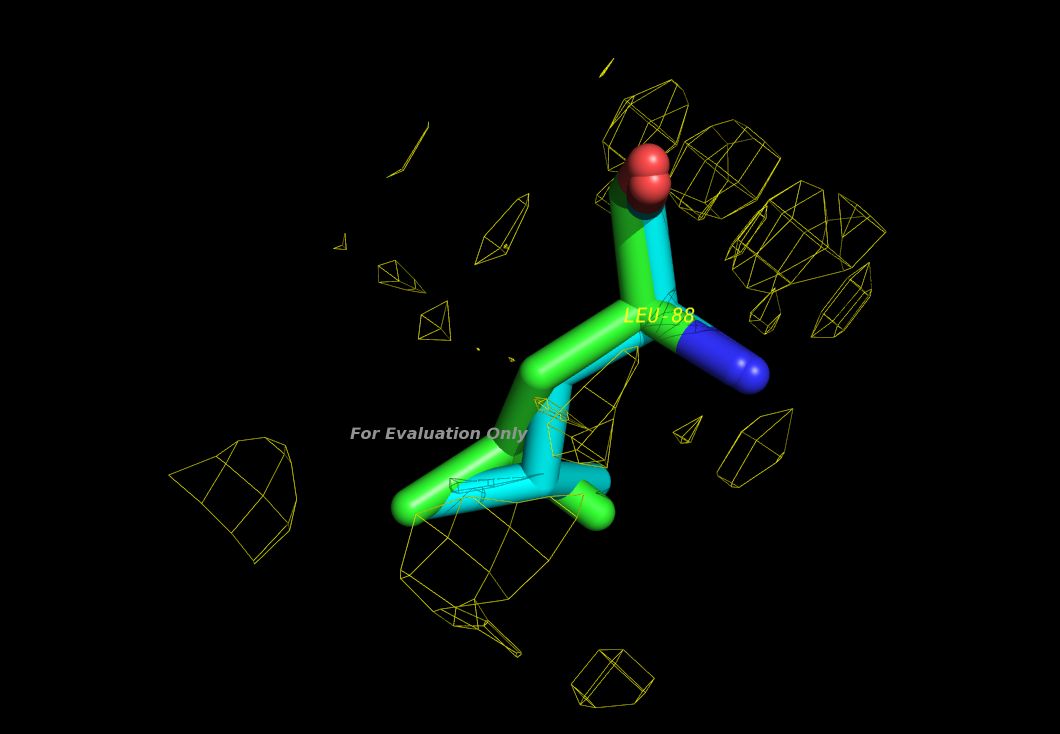
Рисунок 4. Лейцин 88 (оптимизированная структура)