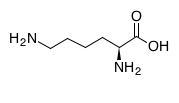
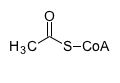
Функции, продолжение. Ферменты и метаболические пути.
EC 4.3.1.1
Для получения информации об этом коде воспользуемся ссылкой International Union of Biochemistry and Molecular Biology и далее следуем по ссылкам - в соответствии с кодом фермента.
EC 4 Lyases Лиазы(ферменты, катализирующие расщепление связей С-С, С-О, С-N или др., сопровождающееся образованием двойных связей, а также обратные реакции - присоединения по двойным связям или образовывающие связи без участия окислительно-восстановительных процессов или гидролиза) EC 4.3 Carbon-Nitrogen Lyases Углерод-азот лиазы(расщепление связи C-N) EC 4.3.1 Ammonia-Lyases Аммиак-лиазы EC 4.3.1.1 Aspartate ammonia-lyase Аспартат-аммиак-лиазы(дезаминирование аспартата) Катализируемая реакция: L-аспартат = фумарат + NH3Этому коду соответствуют 2 записи PBD:
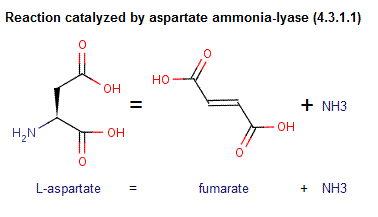
рисунок взят из БД BRENDA
Другие названия фермента: aspartase; fumaric aminase; L-aspartase; L-aspartate ammonia-lyase Систематическое название: L-aspartate ammonia-lyase (fumarate-forming) Ссылки на БД: BRENDA, EXPASY, GTD, KEGG, PDB, CAS registry number: 9027-30-9
1j3u (организм Bacillus sp. YM55-1, длина 468, Uniprot: Q9LCC6),
1jsw (организм Escherichia coli (strain K12), длина 478, Uniprot: P0AC38 )
На главной страничке БД KEGG провели поиск гена по названию его локуса "eco:b4139" (в KEGG идентификатор штамма Escherichia coli K12 - eco).
Описание метаболических путей
ID Definition -------------------------------------------------------------------------------------------------------------------------------------------- eco00250 Alanine, aspartate and glutamate metabolism - Escherichia coli K-12 MG1655 метаболизм аланина, аспартата, глутамата eco00910 Nitrogen metabolism - Escherichia coli K-12 MG1655 метаболизм азота eco01100 Metabolic pathways - Escherichia coli K-12 MG1655 метаболические путиГрафический файл с картой пути eco00250
Провели поиск по L-лизин(L-Lysine) и ацетил-СоА(acetyl-CoA).
L-лизин: ацетил-СоА: английское название: L-Lysine (Lysine acid; 2,6-Diaminohexanoic acid) Acetyl-CoA (Acetyl coenzyme A) идентификатор: C00047 C00024 структурная формула: C6H14N2O2 C23H38N7O17P3S
В поле запроса ввели найденные в упр.3 идентификаторы соединений. Первое соединение раскрасили красным, а второе - зеленым, промежуточные соединения (кратчайший путь)- желтым, другие возможные пути (более длинные) отмечены другими цветами.
Было найдено несколько путей, при том как от L-лизина к ацетил-СоА, так и наоборот.
Цепочка ферментативных реакций 1 (9 интермедиатов):
путь: биосинтез лизина
цепочка: ацетил-СоА→L-лизин
В данном примере желтым отмечен кратчайший путь, но он не реализуется из-за недостатка 5 ферментов. Реализуется другой путь, более долгий(отмечен розовым, переходит в желтый).
Цепочка ферментативных реакций 2:
путь: распад лизина
цепочка: L-лизин→ацетил-СоА
| Организм | Возможна ли цепочка реакций (да/нет/неизвестно) |
Обоснование |
| Escherichia coli K-12 MG1655 | нет | отсутствуют все ферменты, необходимые для осуществления цепочки реакций |
| Archaeoglobus fulgidus | нет | отсутствуют все ферменты, необходимые для осуществления цепочки реакций |
| Arabidopsis thaliana | нет | отсутствуют все ферменты, необходимые для осуществления выбранной цепочки реакций |
| Homo sapiens | нет | отсутствуют 6 ферментов, необходимых для осуществления цепочки реакций |
Как показывает результат, ни в одном из предложенных организмов нет выбранной цепочки ферментативных реакций. Вероятно, это связано с недостаточной степенью исследования ферментов, необходимых для осуществления этой цепочки реакций.
- С помощью запроса к SRS находим ферменты с кодом ЕС2.7.7.1 у человека и археи Archaeoglobus fulgidus.
- Сравнение доменной организации (по PFAM) найденных белков. Для этого используем режим SW_InterProMatches. для просмотра находок.
- Определение % совпадения последовательностей гомологичных доменов из археи и человека.
- С помощью инструментов KEGG найти для выбранного человеческого белка лучшего ортолога из архей, а для архейного –- лучшего ортолога у эукариот.
ЕС2.7.7.1 - никотинамиднуклеотид-аденилилтрансфераза;
реакция: ATP + nicotinamide ribonucleotide = diphosphate + NAD+
запрос: (([uniprot-ID:*_arcfu] | [uniprot-ID:*_human]) & [uniprot-ECNumber:2.7.7.1])
Было найдено 4 белка, три из которых - у человека:
NMNA1_HUMAN, NMNA2_HUMAN, NMNA3_HUMAN;
один - у археи Archaeoglobus fulgidus:
NADM_ARCFU.
При поиске нужно снять опцию использования маски, чтобы исключить лишние находки.
Для всех белков найден единственный домен:
PFAM PF01467 цитидилилтрансфераза(Cytidylyltransferase) код этого фермента EC:2.7.7 В InterPro Relationships указано, что никотинамиднуклеотид-аденилилтрансфераза относится к дочерним ферментам Children: IPR006418 Nicotinamide-nucleotide adenylyltransferase, archaeal type
Если осуществлять поиск по ID белков, то PFAM выдает тот же домен, но у каждого белка своя длина этого домена. Также в поле Description для белков из организма человека указан еще один код EC 2.7.7.18 (nicotinate-nucleotide adenylyltransferase).
Для дальнейшей работы были выбраны домены
NMNA1_HUMAN(279aa) 
NADM_ARCFU(174aa) 
Построили выравнивание последовательностей двух доменов белков NMNA1_HUMAN и NADM_ARCFU: domain.needle
Идентичность последовательностей 13.3% NADM_ARCFU 1 ------------------------MRAFFVGRFQPYHLGHHEVVKNVLQK 26 :|.|.:.:......|.:.|||.::.. NMNA1_HUMAN 1 MENSEKTEVVLLACGSFNPITNMHLRLFELAKDYMNGTGRYTVVKGIISP 50 NADM_ARCFU 27 VDELIIGIGSAQESHSLENPFTAGERVLMIDRAVDEIK------------ 64 | |.|.:...| ..|..||:|.:.|....| NMNA1_HUMAN 51 V-------GDAYKKKGL---IPAYHRVIMAELATKNSKWVEVDTWESLQK 90 NADM_ARCFU 65 ----------------------------------------------RELG 68 ::.. NMNA1_HUMAN 91 EWKETLKVLRHHQEKLEASDCDHQQNSPTLERPGRKRKWTETQDSSQKKS 140 NADM_ARCFU 69 IDKKVYIIP----------LEDIYRNSLWVAH-VCSMVPPFDVVYTN--- 104 ::.|...:| ||.....:||.:. :..:|..:.::... NMNA1_HUMAN 141 LEPKTKAVPKVKLLCGADLLESFAVPNLWKSEDITQIVANYGLICVTRAG 190 NADM_ARCFU 105 ---NPLVYR---LFKEAGFKVMHTKMYNRNEYHGTEIRRKMLEGEDWEKY 148 ...:|. |:|... .:.....:..|:...|:|||.:..|:..... NMNA1_HUMAN 191 NDAQKFIYESDVLWKHRS-NIHVVNEWIANDISSTKIRRALRRGQSIRYL 239 NADM_ARCFU 149 VPESVAEIIKEIDGIKRLRDISGRDF------------------ 174 ||:.|.|.|:: ..|......|. NMNA1_HUMAN 240 VPDLVQEYIEK----HNLYSSESEDRNAGVILAPLQRNTAEAKT 279
Интересно, что в Uniprot для белка NADM_ARCFU имя локуса AF_2315, если осуществлять поиск по этому имени, то находка явно странная: AF_2315,
не имеет никакого отношения к исходному белку и организму.
Если же искать AF2315, тогда имеем подходящий результат: AF2315.
Лучший ортолог из эукариот для гена белка NADM_ARCFU KEGG ID: afu:AF2315 ген: BC1G_14085(белок гипотетический) организм: Botryotinia fuckeliana B05.10 длина: 245 идентичность: 0.327 рассмотрим еще одну находку (с реальным продуктом): ген: 100197028 ( сходен с АТФ-зависимой ДНК хеликазой PIF) организм: Hydra magnipapillata длина: 1559 идентичность: 0.298 Лучший ортолог из архей для гена белка NMNA1_HUMAN не найден. Если выбрать не Best-best при поиске, а Forward-Best: KEGG ID: hsa:64802 (279 a.a.) ген: MFS40622_1131 (белок гипотетический) организм: Methanocaldococcus sp. FS406-22 длина: 447 идентичность: 0.237 ген: Nmar_1577 (термосома - шаперонный белковый комплекс) организм: Nitrosopumilus maritimus длина: 540 идентичность: 0.250
Как можно заметить, идентичность последовательностей довольно низкая, но наблюдается некоторое сходство доменной структуры,
которое, видимо, указывает на сохранение функции даже в таких далеких организмах.
По результатам видно, что самые лучшие находки - гены, продукты которых - гипотетические белки, что, вероятно, связано с недостаточной изученностью организмов и соответствующих белков.
© Ксения Лежнина 2008-2010