показывает полное название файла, если же таких файлов несколько, то программа покажет все файлы.
Построение и сравнение оптимальное глобальное и оптимальное локальное выравнивание двух последовательностей
Построение полного (глобального) оптимального выравнивания с помощью программы needle пакета EMBOSS
Сначала создаем файл с последовательностью родственного белка, введя команду "seqret sw:o05870 -auto".
Затем вводим команду needle psts_ecoli.fasta psts2_myctu.fasta comparing.needle -auto. В файле содержится информация следующего рода:
- Название программы: needle
- Дата запуска программы
- Документы, с которыми работает программа
- Количество выравниваемых последовательностей, в данном случае 2
- Название белков: PSTS_ECOLI
PSTS2_MYCTU - Матрица замен EBLOSUM62
- Штраф за открытие гэпа: 10.0
- Штраф за каждый дополнительный гэп: 0.5
- Длина выравнивания 395
- Количество совпавших остатков 29.9%
- Сходство 41.3%
- Доля гэпов 18.7%
- Вес выравнивания 329.0
- Последовательности
Задание штрафов, отличных от заданных по умолчанию
Команда needle psts_ecoli.fasta psts2_myctu.fasta comparing2.needle. Выравнивание изменилось. Все показатели: совпадение, сходство, вес, длина, доля гэпов - уменьшились, т.е. выравнивание ухудшилось, несмотря на уменьшение числа гэпов;
Получение выдачи в формате, пригодном для импорта в GeneDoc
Команда needle psts_ecoli.fasta psts2_myctu.fasta comparing3.msf: создает файл в формате msf;
Так выглядит глобальное оптимальное выравнивание в GeneDoc с штрафом за открытие гэпа 10.0, за новые гэпы 0.5
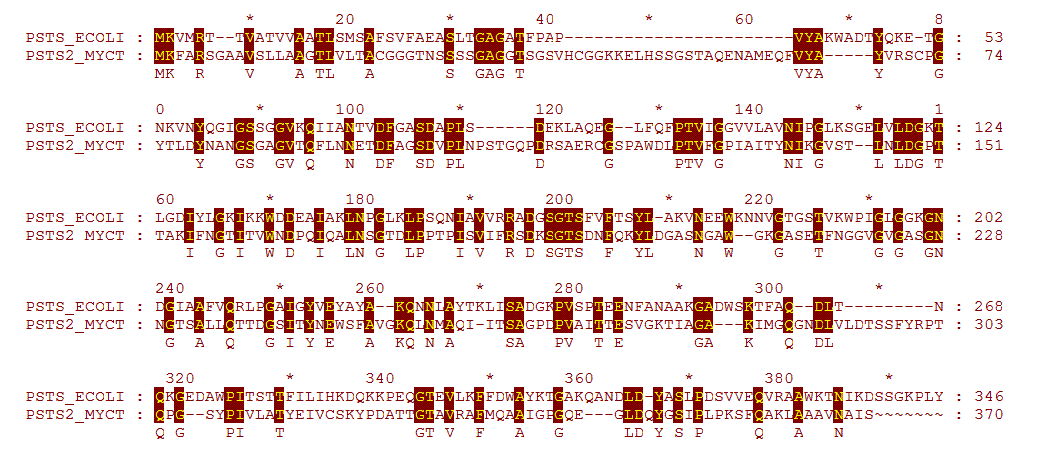
Файл с этим выравниванием.
Глобальное оптимальное выравнивание с штрафом за открытие гэпа 20.0, за новые гэпы 1.0

Файл с этим выравниванием.
Построение локального оптимального выравнивания с помощью программы water пакета EMBOSS
Команда water psts_ecoli.fasta psts2_myctu.fasta scomparing.water -auto: создает файл с локальным оптимальным выравниванием. В файле, аналогично программе needle, есть следующая информация:
- Название программы: water
- Дата запуска программы
- Документы, с которыми работает программа
- Количество выравниваемых последовательностей, в данном случае 2
- Название белков: PSTS_ECOLI
PSTS2_MYCTU - Матрица замен EBLOSUM62
- Штраф за открытие гэпа: 10.0
- Штраф за каждый дополнительный гэп: 0.5
- Длина выравнивания 380
- Количество совпавших остатков 30.3%
- Сходство 42.4%
- Доля гэпов 16.6%
- Вес выравнивания 336.0
- Последовательности
Локальное оптимальное выравнивание с штрафом за открытие гэпа 10.0, за новые гэпы 0.5
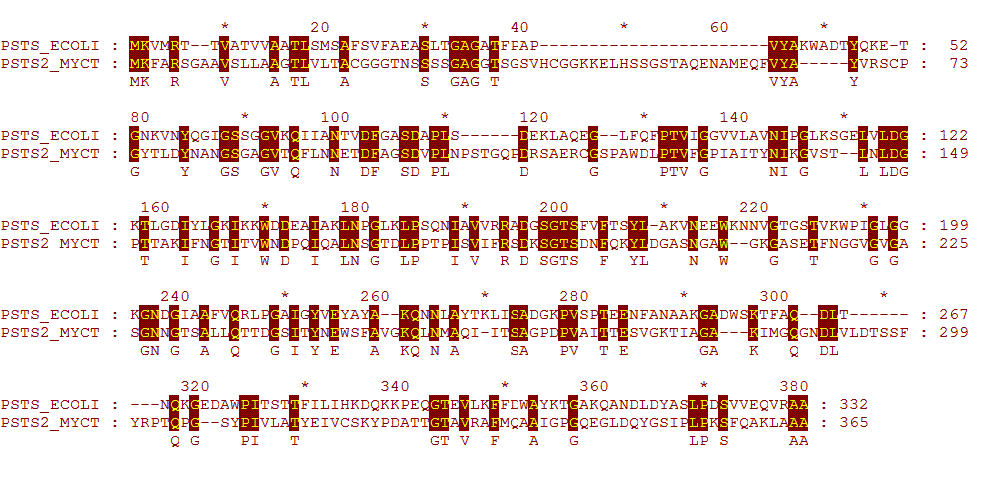
Файл с этим выравниванием.
Увеличим штраф за гэпы: water psts_ecoli.fasta psts2_myctu.fasta scomparing2.water
В итоге, получаем следующие данные:
вес выравнивания, количество гэпов, длина выравнивания уменьшились;
количество совпавших и схожих остатков увеличилось;
Локальное оптимальное выравнивание с штрафом за открытие гэпа 20.0, за новые гэпы 1.0
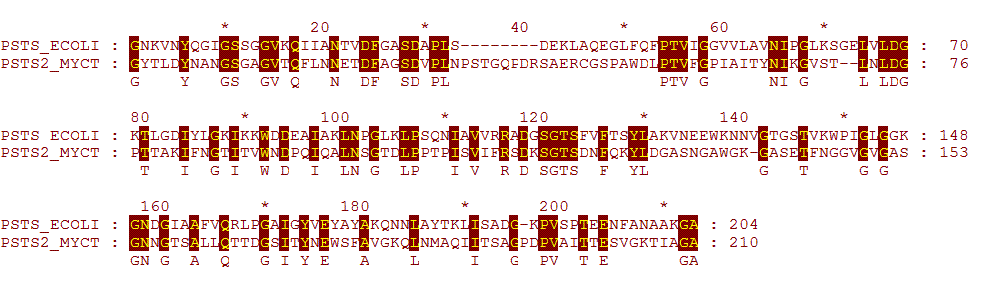
Файл с этим выравниванием.
Уменьшим запрашиваемые параметры вдвое: water psts_ecoli.fasta psts2_myctu.fasta scomparing3.water
В результате:
Все показатели увеличились
Локальное оптимальное выравнивание с штрафом за открытие гэпа 5.0, за новые гэпы 0.25

Файл с этим выравниванием.
Сравнение полученных выравниваний
есть пример того, что одной и той же позиции первой последовательности в разных локальных выравниваниях сопоставлены разные позиции второй последовательности: в выравнивании с штрафом 10.0. и 0.5 на 48-ой позиции белка psts_ecoli находится Y, и ему соответствует Y на 68 позиции белка psts2_myctu,
а в выравнивании с штрафом 20.0 и 1.0 этой же позиции белка PSTS_ECOLI соответствует H на 50 позиции белка PSTS2_MYCTU.
есть пример того, что одной и той же позиции первой последовательности в разных локальных выравниваниях сопоставлены разные позиции второй последовательности:
в выравнивании с штрафом 10.0 и 0.5 на 86 позиции белка PSTS_ECOLI стоит D, ему соответствует D на позиции 113 белка PSTS2_MYCTU, в выравнивании с штрафом 20.0 и 1.0 этой же позиции соответствует S на 114 позиции белка PSTS2_MYCTU (нумерация позиций отличается, так как при открытии файла с помощью GeneDoc теряется информация о том, с какого аминокислотного остатка началось выравнивание),
в выравнивании с штрафом 5.0 и 0.25 ему соответствует P на позиции 112 белка PSTS2_MYCTU.
есть пример того, что в одном глобальном выравнивании какой-либо позиции первой последовательности сопоставлена некоторая позиция второй, а в другом выравнивании против той же позиции оказался пропуск: в выравнивании с штрафом 20.0. и 1.0 на 182 позиции белка PSTS_ECOLI стоит K, ему соответствует W на 209 позиции белка PSTS2_MYCTU,
а в выравнивании с штрафом 10.0 и 0.5 этой же позиции соответствует пропуск.
пример того, что в одном локальном выравнивании какой-либо позиции первой последовательности сопоставлена некоторая позиция второй, а в другом выравнивании против той же позиции оказался пропуск: в выравнивании с параметрами 5.0 и 0.25 312 остатку Q белка PSTS_ECOLI соответствует пропуск PSTS2_MYCTU, в выравнивании с параметрами 10.0 и 0.5 этой позиции соответствует
343 Q белка PSTS2_MYCTU.
Как можно заметить из представленных выравниваний, оптимальные локальные выравнивания соответствуют разным фрагментам последовательностей. Это явно видно при сравнении выравниваний с штрафом за открытие гэпа 5.0, за новые гэпы 0.25 и с штрафом за открытие гэпа 10.0, за новые гэпы 0.5 с выравниванием с штрафом за открытие гэпа 20.0, за новые гэпы 1.0. Выравнены разные фрагменты последовательности. Видимо, при выравнивании с параметрами 20.0 и 1.0 первые 52 аминокислотных остатка оказались неподходящими, поэтому они не входят в полученный результат.
Но при этом, можно заметить, что другие два локальных выравнивания отличаются совсем незначительно и в целом очень похожи.
Локальные выравнивания совпадают с соответствующими частями глобального выравнивания.
Построим карту локального сходства заданных последовательностей с помощью программы dotmatcher пакета EMBOSS
Сначала с помощью dotmatcher создаем файл dotmatcher.ps, затем с помощью GSviewer получаем картинку.
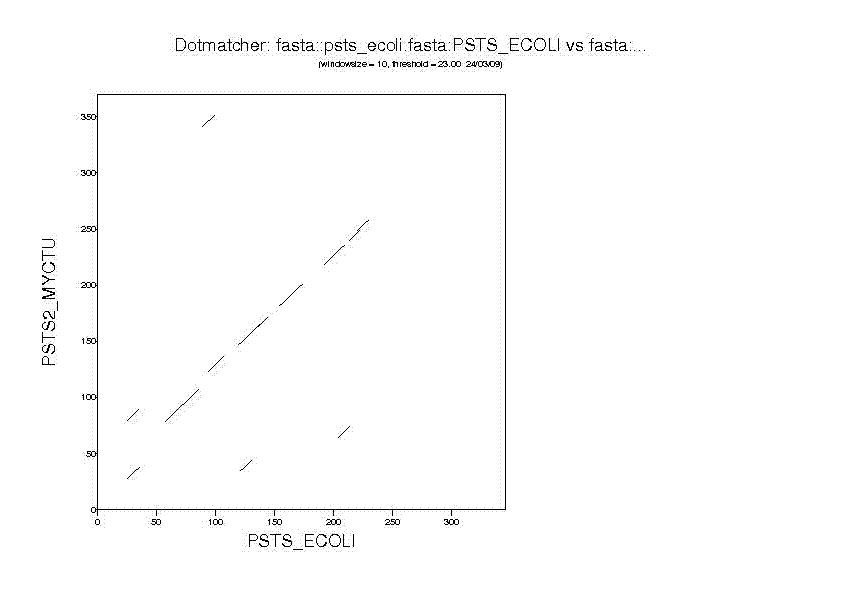
Из представленной карты можно предположить, что белки родственны, но "родственники" они не близкие.
И действительно, PSTS2_MYCTU принадлежит таксону Actinobacteria, который не самый близкий PSTS_ECOLI.
Получим несколько субоптимальных локальных выравниваний заданных последовательностей с помощью программы matcher пакета EMBOSS.
Выберем лучшие из них, в которых фрагменты последовательностей не совпадают с выровненными фрагментами в оптимальном выравнивании.
Вводим команду matcher sw:p0ag82 sw:o05870 align.matcher -alternatives 3 и получаем файл с субоптимальными локальными выравниваниями.
Вот лучшие из них:
Gap_penalty: 14
Extend_penalty: 4
Length: 214
Identity: 75/214 (35.0%)
Similarity: 105/214 (49.1%)
Gaps: 14/214 ( 6.5%)
Score: 239
=======================================
60 70 80 90
PSTS_E GNKVNYQGIGSSGGVKQIIANTVDFGASDAPLS------DEKLAQEG--L
: ..: :: :: : . : :: :: ::. : . :
PSTS2_ GYTLDYNANGSGAGVTQFLNNETDFAGSDVPLNPSTGQPDRSAERCGSPA
80 90 100 110 120
100 110 120 130 140
PSTS_E FQFPTVIGGVVLAVNIPGLKSGELVLDGKTLGDIYLGKIKKWDDEAIAKL
. ::: : . . :: :. . : ::: : :. : : :.: : :
PSTS2_ WDLPTVFGPIAITYNIKGVST--LNLDGPTTAKIFNGTITVWNDPQIQAL
130 140 150 160 170
150 160 170 180 190
PSTS_E NPGLKLPSQNIAVVRRADGSGTSFVFTSYL-AKVNEEWKNNVGTGSTVKW
: : :: :.:. :.: :::: : :: : : : :
PSTS2_ NSGTDLPPTPISVIFRSDKSGTSDNFQKYLDGASNGAWGK--GASETFNG
180 190 200 210
200 210 220 230 240
PSTS_E PIGLGGKGNDGIAAFVQRLPGAIGYVEYAYAKQNNLAYTKLISADGK-PV
.:.: ::.: .: .: :.: : :...: : ..:.. : ::
PSTS2_ GVGVGASGNNGTSALLQTTDGSITYNEWSFAVGKQLNMAQIITSAGPDPV
220 230 240 250 260
250
PSTS_E SPTEENFANAAKGA
. : :. ::
PSTS2_ AITTESVGKTIAGA
270 280
=======================================
Gap_penalty: 14
Extend_penalty: 4
Length: 59
Identity: 14/59 (23.7%)
Similarity: 23/59 (39.0%)
Gaps: 0/59 ( 0.0%)
Score: 43
=======================================
280 290 300 310 320
PSTS_E AWPITSTTFILIHKDQKKPEQGTEVLKFFDWAYKTGAKQANDLDYASLPD
..:: :. .. :: : : : : . . ::
PSTS2_ SYPIVLATYEIVCSKYPDATTGTAVRAFMQAAIGPGQEGLDQYGSIPLPK
310 320 330 340 350
330
PSTS_E SVVEQVRAA
: .. ::
PSTS2_ SFQAKLAAA
360