Учебный сайт Шиндяпиной А.В.
Работа в командной строке Linux. Программы выравнивания последовательностей пакета EMBOSS.
I.Работа в командной строке Linux.
Было предложено для ознакомления с командной строкой Linux выполнить несколько команд, ну и проверить,
что получилось. Для этого использовалась программа Putty. Результаты трудов описаны ниже.
1. Смена активной директории и просмотр содержимого директорий.
- при выполнении команды "ls" putty показывает все поддиректории и файлы активной директории.
- команда "ls .." делает активной предыдущую директорию и показывает её содержимое
- при введении "cd", а затем "ls .." несколько раз (пока картина не перестанет меняться), поднимаешься
"выше" по дереву директорий.
- ввела команды "cd" и "pwd". Команда выдала путь к моей папке (/home/students/y08/student08fbb).
2. Создание и просмотр файлов.
Все команды вводила при активной директории Practice5.
- при введении "seqret sw:p0aa04 -auto" в активной директории создается новый файл формата fasta с записью
аминокислотной последовательности моего белка pthp_ecoli, чьим кодом доступа является p0aa04. После команды
"more pthp_ecoli.fasta" показывается содержимое файла.
- при введении команды "entret sw:p0aa04 -auto" тоже появился файл, но формата entret. Файл содержит информацию о моем
белке, подобную той, что в записях на сайте Uniprot. (см. Блок 1)
II. Построение и сравнение оптимальных глобальных и оптимальных локальных выравниваний 2-х последовательностей.
1. Построение полного (глобального) оптимального выравнивания с помощью программы needle пакета EMBOSS:
- создав файл с помощью программы seqret с последовательностью родственного моему белка (pthp_chltr), выполнила следующие команды:
"needle pthp_ecoli.fasta pthp_chltr.fasta glob.needle -auto" - в результате получила файл glob.needle с выравниванием и расчетами веса выравнивания и др.
двух последовательностей.
"needle pthp_ecoli.fasta pthp_chltr.fasta glob2.needle", после введения этой команды задала штраф за открытие гэпа 20 (вместо 10) и
штраф за продолжение гэпа 1 (вместо 0.5). После изменения параметров получила файл с выравниванием, отличающимся от предыдущего;
- с помощью команды "needle pthp_ecoli.fasta pthp_chltr.fasta glob.msf -auto -aformat msf" получила файл, который можно открыть в GeneDoc
glob1.msf и , задав параметры 20.0 и 1.0, получила файл glob2.msf .
2.Построение локального (частичного) оптимального выравнивания с помощью программы water пакета EMBOSS
- проделала аналогичные действия, только заменяя в командах needle на water + задала параметры штрафов в два раза меньшие
автоматических. Получила файлы под названиями loc1, loc2, loc3, а также два файла для работы в GeneDoc: loc.msf (параметры [10.0][0.5],[20.0][1.0]) и loc1.msf ([5.0][0.25]).
3. Сравнение полученных выравниваний
- есть ли хотя бы один пример того, что одной и той же позиции первой последовательности в разных глобальных выравниваниях
сопоставлены разные позиции второй последовательности?
Да, есть несколько примеров. Например для позиций 26-27 последовательности белка pthp_chltr. Ниже на изображениях:
слева показана часть с 26 и 27 а.о. глобального выравнивания с автоматически заданными параметрами, справа часть выравнивания с параметрами
[20.0] [1.0].
- есть ли хотя бы один пример того, что одной и той же позиции первой последовательности в разных локальных выравниваниях
сопоставлены разные позиции второй последовательности?
Таких примеров нет.
- есть ли хотя бы один пример того, что в одном глобальном выравнивании какой-либо позиции первой последовательности сопоставлена
некоторая позиция второй, а в другом выравнивании против той же позиции оказался пропуск?
Да, т.к. есть гэпы. Как пример, позиции 28-29. На изображениях ниже: слева - выравнивание с автоматически заданными параметрами, справа - с
параметрами [20.0],[1.0].
- есть ли хотя бы один пример того, что в одном локальном выравнивании какой-либо позиции первой последовательности сопоставлена
некоторая позиция второй, а в другом выравнивании против той же позиции оказался пропуск?
Таких примеров нет.
- соответствуют ли оптимальные локальные выравнивания, построенными с использованием разных параметров, одним и тем же
фрагментам последовательностей?
Нет. Соответствуют одним и тем же фрагментам выравнивания с автоматически заданными параметрами [10.0],[0,5] и параметрами [20.0],[1.0]. Выравнивание с параметрами
[5.0],[0.25] построено для большего числа а.о. и имеет два гэпа. Это видно на изображениях.
Из полученных msf-файлов видно, что в локальных выравниваниях меньше гэпов(например, присутствующего в начале глобального выравнивания гэпа нет в локальных выравниваниях), в моих двух из трех локальных выравниваниях с более жесткими параметрами
их вообще нет. Локальные выравнивания меньшей длины, что очевидно из его предназначения, ведь в локальном выравниваются не последовательности полностью, а наиболее гомологичные их участки.
Дополнительные задания
1. Построение карты локального сходства последовательностей белков pthp_ecoli и pthp_chltr с помощью программы dotmatcher пакета EMBOSS.
С помощью команды
dotmatcher pthp_ecoli.fasta pthp_chltr.fasta -graph cps -options
и далее задав параметры для размера окна - 15 (соответсвенно задав и длину участков отрезков последовательности, которые выравниваются) и параметр ограничения
веса последовательностей - 23 (насколько я поняла из информации в интернете это довольно стандартные параметры).
В результатев в активной директории Practice5 программой был сохранен файл dotmatcher.ps , в котором и была карта
локального сходства. Не хитрым образом была получена следующее изображение.
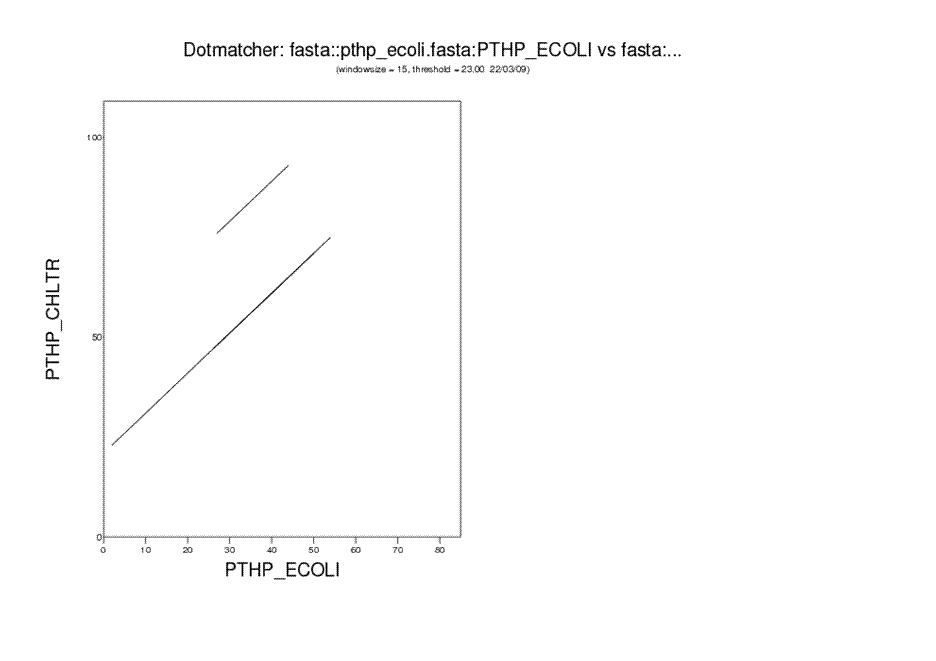
Видно, что для заданных параметров было найдено 3 выравнивания, при этом два из них перекрываются. а третье, видео было
построено с помощью сильного смещения последовательности pthp_ecoli вправо относительно pthp_chltr. Очевидно, лучше использовать в дальнейшем данные по первым двум локальным выравниваниям.
2. получение субоптимельных локальных выравниваний последовательности pthp_ecoli и pthr_chltr.
С помощью программы matcher пакета EMBOSS было получено заданное количество сублокальных выравниваний. После введения команды
matcher pthp_ecoli.fasta pthp_chltr.fasta sybloc.matcher -options
и введения параметров о количестве выравниваний - 7, штрафов за гэпы и каждый пробел - 10 и 1 соотвестсвенно, получила файл
sybloc.matcher . Из 7 выравниваний выбрала 2 наиболее лучших и не совпадающих с уже приведенными
выше локальными.
# Aligned_sequences: 2
# 1: PTHP_ECOLI
# 2: PTHP_CHLTR
# Matrix: EBLOSUM62
# Gap_penalty: 10
# Extend_penalty: 1
#
# Length: 40
# Identity: 10/40 (25.0%)
# Similarity: 16/40 (40.0%)
# Gaps: 1/40 ( 2.5%)
# Score: 31
#
#
#=======================================
10 20 30 40
PTHP_E EVTIT-APNGLHTRPAAQFVKEAKGFTSEITVTSNGKSAS
: :.: .. : . . :. : : :::
PTHP_C EATLTYLGKTVNARSVMSILMLGASYNGEVAVHIKGPSAS
60 70 80 90
#=======================================
#
# Aligned_sequences: 2
# 1: PTHP_ECOLI
# 2: PTHP_CHLTR
# Matrix: EBLOSUM62
# Gap_penalty: 10
# Extend_penalty: 1
#
# Length: 22
# Identity: 8/22 (36.4%)
# Similarity: 14/22 (63.6%)
# Gaps: 1/22 ( 4.5%)
# Score: 30
#
#
#=======================================
60 70
PTHP_E GTVVTISAEGEDEQKAVEHLVK
::.: . :::. . . .: :
PTHP_C GTIVKLF-EGEECEATLTYLGK
50 60
#=======================================
©, "ООО Шиндяпина 2008"