Исследование структуры тРНК
- Краткое описание структуры в файле 1EVV.pdb В файле приведены координаты атомов следующих молекул:
1.PHENYLALANINE TRANSFER RNA ( Фенилаланиновая тРНК ),одна цепь.
Молекула получена из организма дрожжей : SACCHAROMYCES
Для исследования была выбрана цепь A, представляющая фенилаланиновую тРНК со следующей последовательностью:
[1] 5' - G C G G A U U U A 2MG C U C A G H2U H2U G G G A G A G C M2G C C A G A OMC U OMG A A YG A PSU 5MC U G G A G 7MG U C 5MC U G U G 5MU PSU C G 1MA U C C A C A G A A U U C G C A C C A - 3' [76],
где 901 и 970 - номера первого и последнего нуклеотида.
В выбранной цепи отмечу большое количество модифицированных оснований и что на 3'-конце есть триплет CCA(C74,C75,A76), к которому присоединяется аминокислота.
В файле PDB приведены координаты атомов этого триплета. - Исследование вторичной структуры
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи
между азотистыми основаниями (1EVV.fp). В соответствии с полученными данными
акцепторный стебель состоит из участка 1-7 и комплементарного ему участка 72-66.
Т-стебель - из участков 49-53 и 65-61
D-стебель - из участков 10-13 и 25-22
антикодоновый стебель - из участков 38-44 и 32-26
Визуализация расположения стеблей т-РНК с помощью программы RasMol.
На картинке изображена т-РНК в остовной модели.
Акцепторный стебель выделен красным, T-стебель зеленым,
D-стебель синим и антикодоновый стебель оранжевым.
Структуру стеблевых дуплексов поддерживают 20 канонических и 3 неканонические пары оснований.Рис.1. Вторичная структура молекулы фенилаланиновой тРНК из SACCHAROMYCES
Скрипт для получения изображения:
background white
restrict none
select all
backbone 70
color white
select 1-7
or 66-72
color red
select 49-53
or 61-65
color green
select 10-13
or 22-25
color blue
select 38-44
or 26-32
color orange
Ниже приведено изображение неканонической пары из акцепторного стебля:
Взаимодействие остатков G4 и U69
Дополнительные характеристики т-РНК:
-в структуре нет вариабельной петли,
-в T-петле отсутствует остаток тимидинна,
-в D-петле отсутствует остаток дигидроуридина.
� - Исследование третичной структуры
- Предсказание вторичной структуры тРНК
Программа einverted.
Вводимая нуклеотидная последовательность: 1EVV.fasta
Штраф за гэп [12]: 4
(именно при этом значении получились наиболее адекватные р-ты)
Минимальное значение порога [50]:15
Пробовались значения 20,50 (улучшения эти значения не принесли)
Значение основания (канонической пары) [3]:3
Значение неканонической пары("несоответствие") [-4]:-4
Выходной файл :1EVV.inv
Выравнивание
1EVV: Score 16: 8/10 ( 80%) matches, 0 gaps 22 gagcgccaga 31 || | ||||| 48 ctggaggtct 39 1EVV: Score 15: 5/5 (100%) matches, 0 gaps 49 ctgtg 53 ||||| 65 gacac 61
Реальная и предсказанная вторичная структура тРНК из файла 1EVV.pdbУчасток структуры Позиции в структуре (по результатам find_pair) Результаты предсказания с помощью einverted Результаты предсказания по алгоритму Зукера Акцепторный стебель 5' 1-7 3'
5' 66-72 3'
Всего 7 парПредсказано 0 пар из 7 реальных Предсказано 6 пар из 7 реальных D-стебель 5' 10-13 3'
5' 22-25 3'
Всего 4 парыПредсказано 0 пар из 4 реальных Предсказано 4 пары из 4 реальных T-стебель 5' 49-53 3'
5' 61-65 3'
Всего 5 парПредсказано 5 пар из 5 реальных Предсказано 5 пар из 5 реальных Антикодоновый стебель 5' 38-44 3'
5' 26-32 3'
Всего 7 парПредсказано 5 пар из 7 реальных Предсказано 5 пар из 7 реальных Общее число канонических пар нуклеотидов 23 пары 10 пар 20 пар
Алгоритм ЗукераЗаданная команда без значения параметраа Р (default P=5)
mfold SEQ=trna.fasta
При увеличенни параметра "P" программа выдавала много структур, которые не оказались лучше первоначальной.
Изображение, полученное с помощью mfold: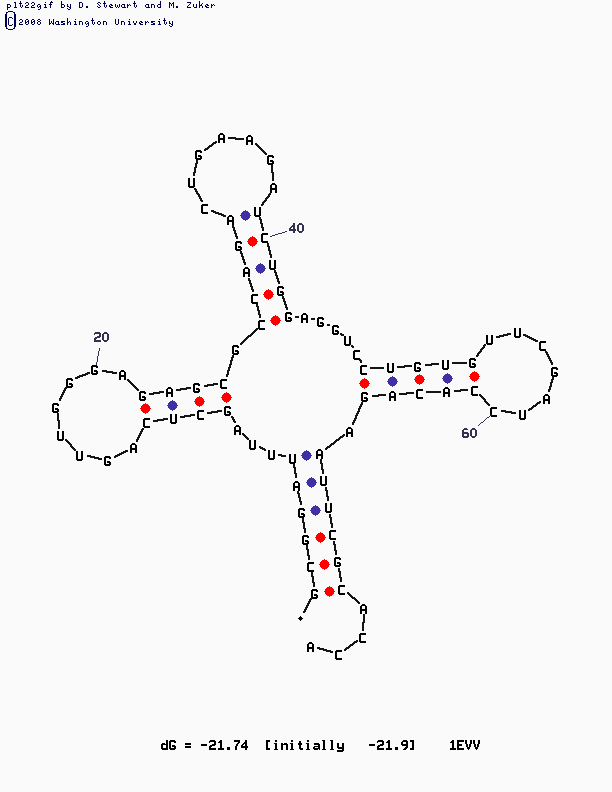
1. Данные о предположительных стрекинг-взаимодействиях получены в результате выполнения команды:
find pair -t 11QRT.pdb stdout | analyze и содержатся в выходном файле - 1EVV.out.
Всего было выдано 28 потенциально возможных взаимодействий.
Перекрытие Uc/GA, обладающее площадью (4,19 квадратных ангстрем), было замечено под номером 7.
Это - стрекинг-взаимодействие между основаниями U7 и A66 конца акцепторного стебля и с49 и G65 начала T-стебля.
Изображение соответствующего стекинг-взаимодействия получено при помощи выполнения последовательных команд:
ex_str -7 stacking.pdb step7.pdb (вырезание нужной структуры в отдельный файл),
stack2img -cdolt step7.pdb step7.ps (построение изображения).
2. Среди шести водородных взаимойдейсвий вне стеблей я выделил три, которые происходят между основаниями D и Т-петель.
Это пары нуклеотидов
U54 - a58
U55 - G18
G15 - C48
Неканоническая среди них только одна U55 - G18.