Исследование ДНК-белковых взаимодействий в структуре комплекса белков(факторы транскрипции DP-2 и E2F-4) и фрагмента Днк.
- Краткое описание структуры в файле 1CF7.pdb В файле приведены координаты атомов следующих молекул:
1.PROTEIN (TRANSCRIPTION FACTOR E2F-4), DNA-BINDING DOMAIN (ДНК-связывающий домен белка
(фактор транскрипции E2F-4).Одна цепь - A.
2.PROTEIN (TRANSCRIPTION FACTOR DP-2), DNA-BINDING DOMAIN (ДНК-связывающий домен белка
(фактор транскрипции DP-2).Одна цепь - B.
3.Фрагмент ДНК, состоящий из двух комплементарных цепей - C и D.
Молекулы взяты из организма HOMO SAPIENS (человека).
Для исследования были выбраны:
Цепь А белка(фактор транскрипции E2F-4),
Цепь B белка(фактор транскрипции DP-2) и цепи С и D, представляющие ДНК со следующей последовательностью:
цепь C [501] 5' - attttcgcgcggtttt - 3' [515] ||||||||||||||| цепь D [615] 3' - aaaagcgcgccaaaat - 5' [601],где 501 и 515 - номера первого и последнего нуклеотида.
- Функции белков, структуры которых представлены в файле 1CF7.pdb
� Белок (фактор транскрипции DP-2)
Оригинальный текст
Может стимулировать E2F-зависимую транскрипцию.Связывает ДНК совместно с членами
семейства E2F на сайте распознавания, 5'-TTTC[CG]CGC-3', найденном в промоторной
области некоторого числа генов, чьи продукты вовлекаются в регуляцию клеточного цикла или репликацию ДНК.
�
Белок (фактор транскрипции E2F-4)
Оригинальный текст
� Активатор транскрипции который связывает ДНК совместно с DP белками на сайте распознавания, 5'-TTTC[CG]CGC-3',
найденном в промотерной � области некоторого числа генов, чьи продукты вовлекаются в регуляцию клеточного цикла или репликацию ДНК.
� DRTF1/E2F комплекс функций, контролирующих развитие клеточного цикла от G1 к S фазе.
E2F-4 связывается c родственными белками RBL1 и RBL2.В некоторых случаях, могут также связывать RB белок. - Исследование структуры ДНК
1.Исследование структуры ДНК с помощью программ find_pair и analyze.
С помощью вышеуказанных программ получен файл DNA_old.out
Исходя из полученных данных мы имеем дело с ДНК в B-форме.
2.Определение средних значений торсионных углов для внутренних нуклеотидов(всех, кроме краевых).
Из файла DNA_old.out была взята информация для построения Excel-таблицы.
Таблицы в этом файле:
Первая таблица - выдержка из файла DNA_old.out.
Вторая таблица - отрицательные углы были переведены в соответствующие положительные.
Указаны средние значения каждого из углов.
Третья таблица - показывает отклонение величины каждого угла от среднего значения для этого угла.
Синим обозначен самый "кривой" нуклеотид, среднее значение которого наиболее отклоняется от других
Резюме:
По данным Excel таблицы можно сказать что деформация для всех нуклеотидов
распределена неравномерно.
Участки сильно отклоняющихся нуклеотидов чередуются со слабо отклоняющимися.
Основной участок деформации отмечен желтым.
- Исследование природы ДНК-белковых контактов
Описать ДНК-белковые контакты в заданной структуре. Сравнить количество контактов разной природы.
Будем считать полярными атомы кислорода и азота, а неполярными - атомы углерода, фосфора и серы.
Назовем полярным контактом ситуацию, в которой расстояние между полярным атомом белка и полярным атомом ДНК
меньше 3.5A. Аналогично, неполярным контактом будем считать пару неполярных атомов на расстоянии меньше 4.5A.
В скрипте my_dna.def приведены команды для выделения в RasMol множеств, необходимых для определения числа контактов.
Таблица:
Контакты разного типа в комплексе 1CF7.pdb
Контакты атомов белка с Полярные Неполярные Всего остатками 2'-дезоксирибозы 8 19 27 остатками фосфорной кислоты 17 17 34 остатками азотистых оснований со стороны большой бороздки 7 15 22 остатками азотистых оснований со стороны малой бороздки 2 1 3
Резюме
Наибольшее число контактов обнаружено с остатками фосфорной кислоты.
Скорее всего данные атомы расположены на поверхности молекулы ДНК и доступны
� для взаимодействия с атомами белка.
� Контакты атомов белка с остатками азотистых оснований со стороны бороздок
� Из таблицы видно, что атомы белка "легче" контактируют с основаниями из большой бороздки чем из малой.Это по-моему мнению связано с тем, что большая бороздка в силу своей глубины наиболее предрасположена для сближения с белком и лучшего связывания. - Получение популярной схемы ДНК-белковых контактов с помощью nucplot
Использованная в Putty команда:
>nucplot 1CF7_old.pdb
Полученные картинки
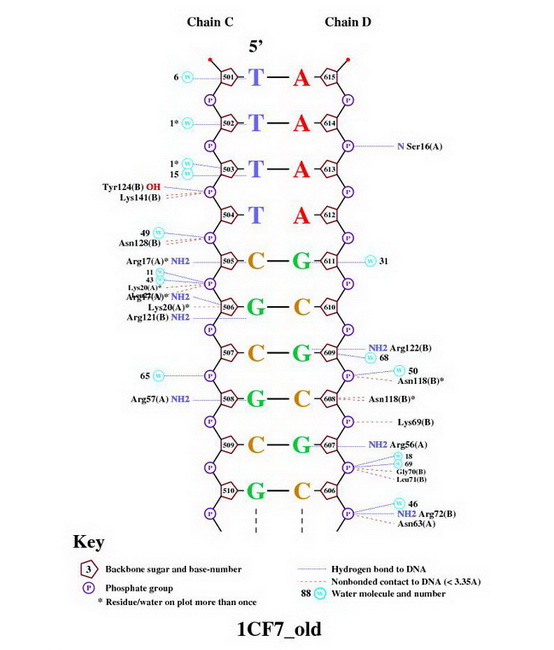
Сравнивая картинку из nucplot и расчеты из Excel файла можно сделать вывод
о том, что контакты с белком не влияют на "искривления" нуклеотидов.
� Так наиболее искривленный нуклеотид из таблицы не имеет контакта (атом белка-основание) с белком. Зона, покрашенная в таблице желтым, также не имеет контактов с атомами белка.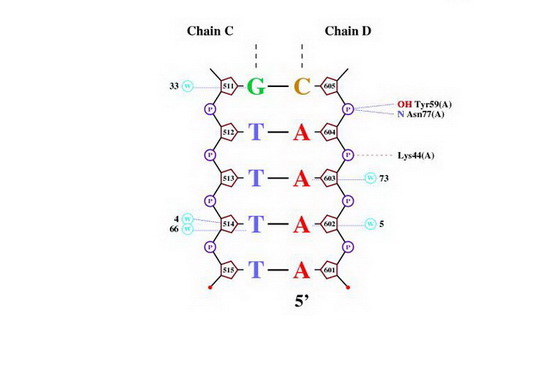
Видно что результаты, полученные с помощью программы nucplot и результаты о контактах белка и ДНК в RasMol, не совпадают.
По данным nucplot присутствуют:
16 контактов между белком и остатками фосфорной кислоты и всего 9 контактов между атомами азотистых оснований и атомами ДНК. Все это выявляет различия между алгоритмом определения контактов в этих случаях.
- Возможные распознающие контакты
� На роль распознающих я выбрал 2 контакта.
Первый контакт
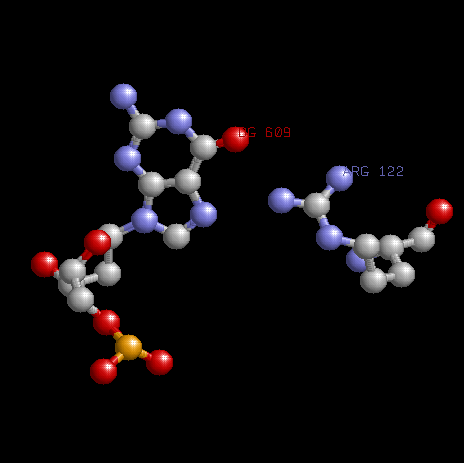
аминокислоты ARG122 (цепь B - белка (фактор транскрипции DP-2) и нуклеотида DG609 (рис.слева).
Второй контакт
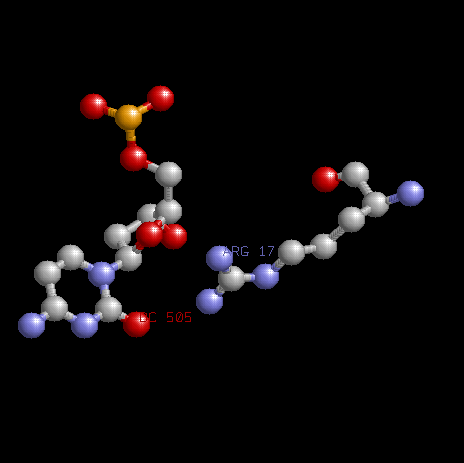
аминокислоты ARG17 (цепь A - белка (фактор транскрипции E2F4-4) и нуклеотида DC505 (рис.справа).
Причины выбора:
1.Контакт аминокислоты с основанием нуклеотида.
2.Нахождение рядом еще нескольких контактов( аминокислота-основание нуклеотида ).
Картинка взаимодействия аминокислота - нуклеотид
- Характеристика ДНК-связывающего домена E2F_TDP
Доменная структура белка из исследуемого комплекса, полученная с помощью инструментов Pfam
Белок (транскрипционный фактор DP-2)

Source Domain Description Start End Pfam-A E2F_TDP E2F/DP family winged-helix DNA-binding domain 127 208 Pfam-A DP Transcription factor DP 215 360
Белок (транскрипционный фактор E2F-4)

Source Domain Description Start End Pfam-A E2F_TDP E2F/DP family winged-helix DNA-binding domain 17 83
Семейство E2F/DP winged-helix Днк-связывающего домена.
Это семейство включает фактор транскрипции E2F и его димеризованных партнеров TDP1 и TDP2, которые стимулируют E2F-зависимую транскрипцию. E2F связывает Днк как гомодимер или как гетеродимер в комплексе с TDP1/2, гетеродимер повышает эффективность связывания. Кристалическая структура E2F4-DP2-DNA комплекса показывает, что Днк-связывающие домены E2F
и DP белков имеют сгиб, относящийся к winged-helix Днк-связывающего мотива.
Отдельно о транскрипционном факторе DP
DP формы образуют гетеродимер с E2F.Получившийся E2F1-DP1 гетеродимер регулирует гены, вовлеченные в цикл клеточного развития. Транскрипционная активность E2F ингибируется белком ретинобластомы, который связывается с E2F-DP
гетеродимером и негативно регулирует G1-S развитие.
Главная страница
©Голяев Виктор