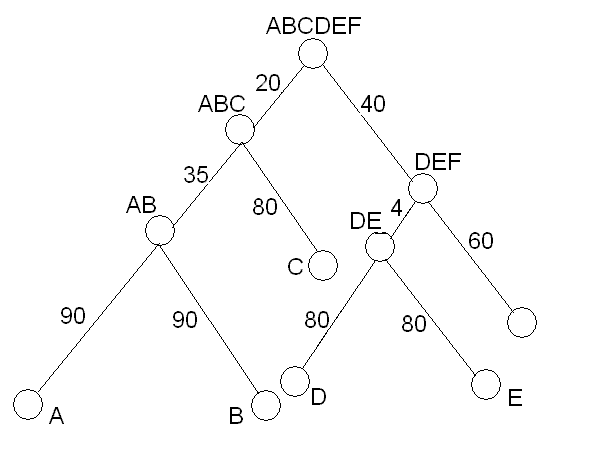
Задание 4. На основе последовательностей, соответствующих листьям, реконструировать дерево алгоритмами UPGMA,
Neighbor-joining и максимального правдоподобия.
Был создан файл, содержащий последовательности всех
листьев исследуемого дерева. Его обработали с помощью программы fdnaml для того, чтобы реконструировать дерево
алгоритмом максимального правдоподобия:
fdnaml all_ali.fasta -ttratio 1 -auto
Программа создает два файла - с расширением .treefile, в котором содержится запись дерева в формате, используемом в п.1.1
этого практикума. Второй файл - с расширением .dnaml - содержит схематичное изображение дерева и небольшое текстовое
описание(кладограмма):
+--------------------B
|
| +-------------F
| +---------------4
| | | +----------------E
1------2 +---3
| | +-----------------D
| |
| +-------------------C
|
+---------------------A
В файле содержится информация о том, что дерево неукорененное. Также там находится таблица с данными о количестве замен на
том или ином этапе эволюции и т.д.
Between And Length Approx. Confidence Limits
------- --- ------ ------- ---------- ------
1 A 0.70861 ( 0.57196, 0.84529) **
1 B 0.67832 ( 0.54392, 0.81264) **
1 2 0.22253 ( 0.09211, 0.35299) **
2 4 0.51315 ( 0.36684, 0.65935) **
4 F 0.46255 ( 0.36944, 0.55570) **
4 3 0.13243 ( 0.04359, 0.22119) **
3 E 0.56914 ( 0.47118, 0.66716) **
3 D 0.58169 ( 0.48197, 0.68140) **
2 C 0.64719 ( 0.51429, 0.78002) **
* = significantly positive, P < 0.05
** = significantly positive, P < 0.01
Результат работы программы UPGMA:
+---------------------A
+--3
+-------4 +---------------------B
! !
! +-----------------------C
--5
! +-----------------D
+-------------2
! +-----------------E
+-1
+-----------------F
В файле также помещена таблица с данными о количестве замен на
том или ином этапе эволюции и т.д.
From To Length Height
---- -- ------ ------
5 4 0.24268 0.24268
4 3 0.09830 0.34099
3 A 0.70317 1.04415
3 B 0.70317 1.04415
4 C 0.80147 1.04415
5 2 0.44187 0.44187
2 D 0.60228 1.04415
2 1 0.02604 0.46791
1 E 0.57624 1.04415
1 F 0.57624 1.04415
Neighbor-joining алгоритм
+-------------------B
!
! +------------------C
! !
1--------2 +---------------D
! ! +----3
! +-----------------4 +-------------------E
! !
! +-------------F
!
+----------------------A
И такая же таблица, как в предыдущих программах, с напоминанием о неукорененности дерева:
Between And Length
------- --- ------
1 B 0.65406
1 2 0.28146
2 C 0.61831
2 4 0.58117
4 3 0.15305
3 D 0.52128
3 E 0.65692
4 F 0.44956
1 A 0.75227
Сравнение полученных деревьев:
ABCDEF real Max UPGMA N-J
***..* + + - +
***... + + + +
**.... + + + +
****.. - - + -
Max - алгоритм максимального правдоподобия;
N-J - программа Neighbor-joining;
real - дерево, построенное вручную по скобочной схеме.
Как видно из таблицы, больше всего на исходное дерево похожи деревья, реконструированные с помощью программ N-J и Max. Скорее
всего это связано с тем, что эти алгоритмы работают, не учитывая теорию молекулярных часов - следовательно, они более пригодны
для построения деревьев неультраметрических, у которых расстояния от каждого листа до корня не равны между собой. Программа UPGMA,
напротив, работает с деревьями в соответствии с теорией молекулярных часов. Исследуемое дерево неультраметрическое, поэтому с
его реконструкцией лучше справились программы, работающие с неультраметрическими деревьями.
© Pouliakhina
All rights reserved