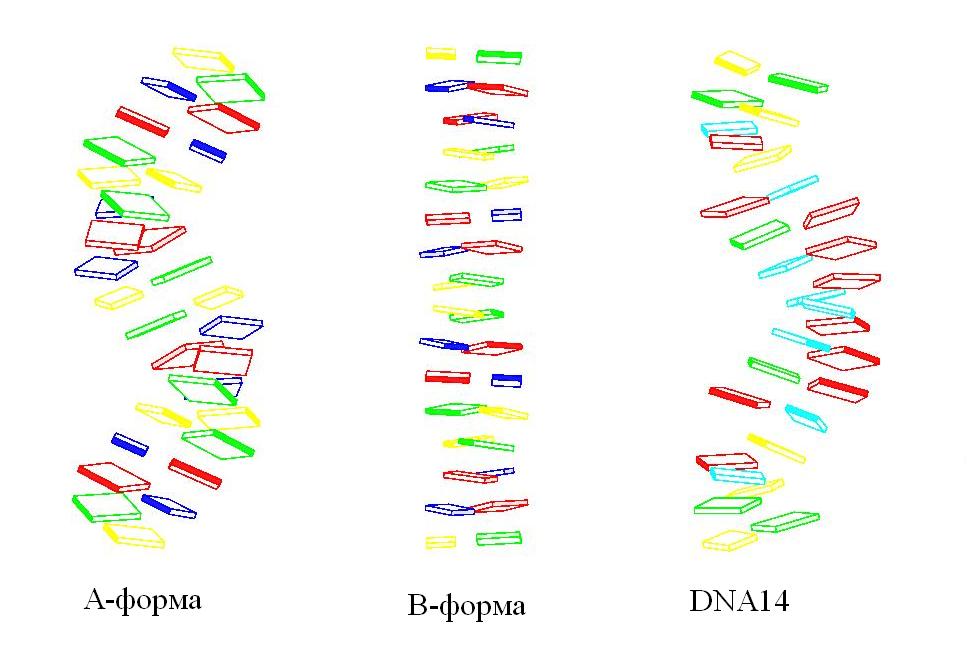
| A-форма | B-форма | DNA14.pdb | |
| Тип спирали (правая или левая) | правая | правая | правая |
| Шаг спирали (Å) | 28.02 (C16A.P-G5A.P) | 33.752 (A6A.P-C15A.P) | 29.448 (A5A.P-Ca6A.P) |
| Число оснований на виток (Å) | 11 | 10 | 11 |
| Ширина большой бороздки (Å) | 7.985 (A22B.P-G5A.P) | 17.216 (G24B.P-A6A.P) | 12.379 (A21B.P-A5A.P) |
| Ширина малой бороздки (Å) | 16.809 (G21B.P-A14A.P) | 11.693 (A10A-T27B.P) | 17.199 (A25B.P.P-A9A.P) |