1.Глутаминовая тРНК.
2.Глутаминовая тРНК синтетаза. Мутация С229R.
Выделены из организма Еrichia coli.
Для исследования была выбрана
цепь B, представляющая глутаминовую тРНК со следующей последовательностью:
[902] 5' - GGGGGUAUCGCCAAGCGGUAAGGCACCGGAUUCUGAUUCCGGCAUUCCGAGGUUCGAAUCC UCGUACCCCAGCCA- 3' [976],
где 902 и 976 - номера первого и последнего нуклеотида.В последовательности на 3'-конце есть триплет CCA, к которому присоединяется аминокислота. В PDB приведены координаты его атомов.
акцепторный стебель состоит из участка 902-907 и комплементарного ему участка 971-966.
Т-стебель 949-953 и 965-961
D-стебель 910-912 и 925-923
антикодоновый стебель 937-944 и 933-926
| Рис.1. Вторичная структура глутаминовой тРНК из Еrichia coli (2RE8) | Скрипт для получения изображения |

| select all restrict not protein select 902-907:B or 966-971:B color red select 949-953:B or 961-965:B color green select 910-912:B or 923-925:B color blue select 937-944:B or 926-933:B color orange select not(902-907:B or 966-971:B or 949-953:B or 961-965:B or 910-912:B or 923-925:B or 937-944:B or 926-933:B ) color grey select not protein backbone wireframe off select 936:b or 935:b or 934:b color cpk wireframe Данный скрипт служит для получения в RasMol изображения остова исследуемой тРНК, где акцепторный стебель выделен красным, Т-стебель - зеленым, D-стебель - синим, антикодоновый - оранжевым. В шарнирной модели выделены нуклеотиды CUG , являющимися антикодоном для кодона глутаминовой кислоты. (GAC) |
2.
| Изображение не канонической пары Аденин-Аденин в структуре исследуемой тРнк |
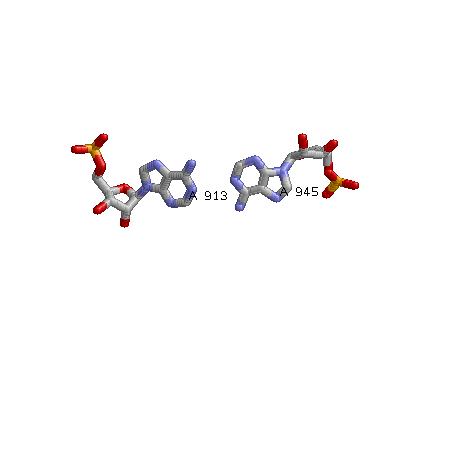
|
3)Вариабельная петля отсутствует ;
а)Тимидин в Т-петле отсутствует ;
а)Дигидроуридин в D-петле отсутствует;
| Наложение оснований конца акцепторно стебля и начала Т-стебля. | Данные в пользу стекинг-взаимодействия. | ||
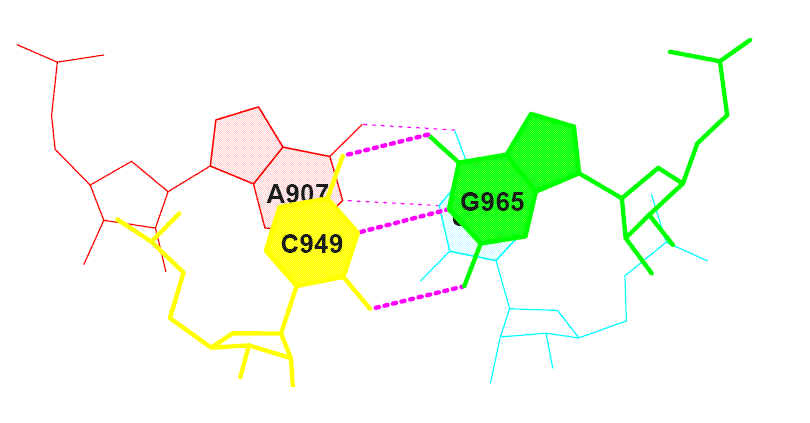
|
По данным файла 2RE8.out (результат пропраммы analyze) площадь перекрывания равна 8.9 ,когда как максимальная 12.06. Такой результат говорит о высокой вероят ности стекинг-взаимодействия между основаниями конца акцепторно стебля и начала Т-стебля. |
| Изображение неканонической пары между основаниями D- и Т-петель. | Дополнительные водородные связи между основаниями D- и Т-петель. | ||
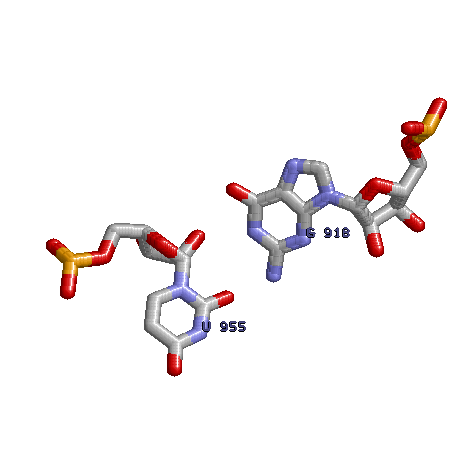
|
Есть дополнительные водородные связи между основанием аденина 955 D- петли и 918 основанием гуанина Т-петели (неканоническая пара), а также между 919 основанием гуанина D- петли и 956 Т-петели основанием цитозина (каноническая пара) |
| Участок структуры |
Позиции в структуре (по результатам find_pair) |
Результаты предсказания с помощью einverted |
Результаты предсказания по алгоритму Зукера |
| Акцепторный стебель | 5' 902-971 3' 5' 966-971 3' Всего 6 пар |
предсказано 6 пары из 6 реальных | предсказано 4; |
| D-стебель | 5' 910-912 3' 5' 923-925 3' Всего 6 пар |
предсказано 0 | предсказано 5; |
| T-стебель | 5' 945-953 3' 5' 965-961 3' Всего 7 пар |
предсказано 0 | ;предсказано 6 |
| Антикодоновый стебель | 5' 937-944 3' 5' 926-933 3' Всего 8 пар |
предсказано 0 | предсказано 5 |
| Общее число канонических пар нуклеотидов | 20 | 6 | 20 |
| Структура ,предсказанная программой Мфолд. | Комментарии. |
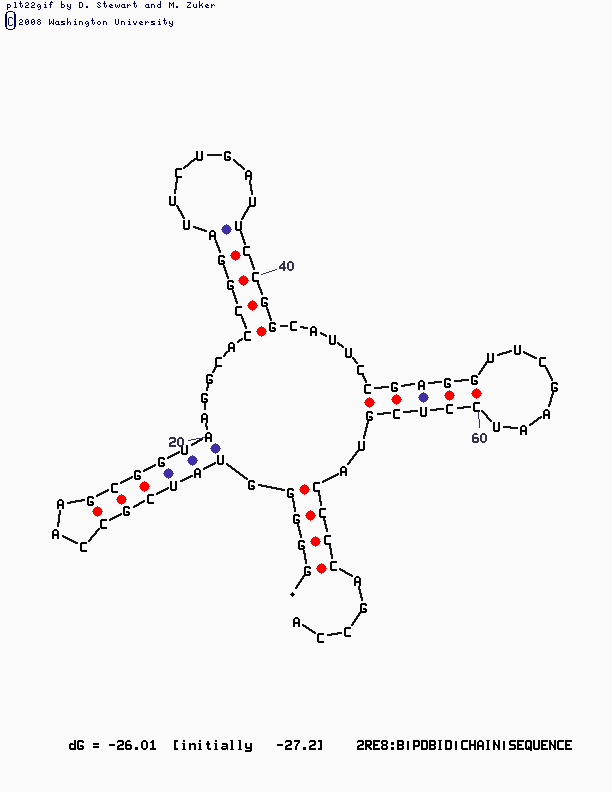
|
Вторичная структура РНК - структура, образуемая спаренными основаниями на
однонитевой молекуле РНК. Биологическая роль вторичной структуры:
структурная (РНК - рибосомная, тРНК), регуляция
, рибозимы, стабильность РНК. Предсказание вторичной структуры является сложной задачей. Для предсказания вторичной структуры используются энергетические параметры, а они определены не очень точно. Более того, в клетке бывают разные условия, и, соответственно, реализуются разные параметры. Находится единственная структура с минимальной энергией, в то время как обычно существует несколько структур с энергией, близкой к оптимальной. Только около 65-70% тРНК сворачиваются в правильную структуру. В данном случае программа Мфолд определила вторичную структуры довольно точно. Определены все пары,кроме неканонических.(Программа выдала этот результат при запуске с параметром субоптимальности 15). Программа einverted из-за еще менее совершенного алгоритма нашла только акцепторный стебель(при запуске с различными параметрами).Второй вариант который она выдает определяeт только 1-2 или не одного взаимодействия из каждой шпильки. Скорее всего эта программа не предназначена для предсказания подобных структур. |