7. Характеристики PDB-файла; биологические единицы
1. Технические характеристики PDB-записи 1DUV
- Разрешение - 1.70Å
REMARK 2 RESOLUTION. 1.70 ANGSTROMS.
- Число рефлексов - 119618
REMARK 3 NUMBER OF REFLECTIONS : 119618
- Полнота (процент рефлексов с разрешением ниже указанного) - 85,8%
REMARK 3 COMPLETENESS (WORKING+TEST) (%) : NULL REMARK 200 OVERALL. REMARK 200 COMPLETENESS FOR RANGE (%) : 85.8
- R-фактор - 0,192
REMARK 3 R VALUE (WORKING SET) : 0.192
- Свободный R-фактор - 0,221;
процент рефлексов, использованных для его вычисления - не приведён в записи:REMARK 3 FREE R VALUE : 0.221 REMARK 3 FREE R VALUE TEST SET SIZE (%) : NULL
Зато приведено число таких рефлексов:REMARK 3 FREE R VALUE TEST SET COUNT : 6000
Можем посчитать процент самостоятельно: 6000 / 119618 * 100% = 5,02% - Кристаллографическая группа и тип симметрии - P 21 21 2
CRYST1 86.680 134.230 109.300 90.00 90.00 90.00 P 21 21 2 12
Из этой же строки получены следующие данные: - Длины векторов a, b, c
(базисных периодов кристаллической решётки):
a = 86,680
b = 134,230
c = 109,300 - Углы между базисными периодами равны и составляют 90°, то есть базисные периоды ортогональны.
- Депонируемая (используемая в записи PDB) система координат совпадает с абсолютной, т.к. матрица перехода единичная:
ORIGX1 1.000000 0.000000 0.000000 0.00000 ORIGX2 0.000000 1.000000 0.000000 0.00000 ORIGX3 0.000000 0.000000 1.000000 0.00000
- По данным записи PDB биологическая единица совпадает с ассимметрической единицей, записанной в файле. Это указано в поле REMARK 350.
- Все атомы в структуре имеют коэффициент заполнения, равный единице.
2. Вычисление относительных координат атомов
Вычислим относительные координаты одного из СА-атомов исследуемого белка. Для этого воспользуемся матрицей перехода к относительным координатам:SCALE1 0.011537 0.000000 0.000000 0.00000 SCALE2 0.000000 0.007450 0.000000 0.00000 SCALE3 0.000000 0.000000 0.009149 0.00000Возьмём СА-атом серина-2 цепи G:
ATOM 2 CA SER G 1 19.118 15.243 52.461 1.00 70.26 CРассчитаем его относительные координаты:
19,118 * 0,011537 + 15,243 * 0,000000 + 52,461 * 0,000000 = 0,220564366
19,118 * 0,000000 + 15,243 * 0,007450 + 52,461 * 0,000000 = 0,113560350
19,118 * 0,000000 + 15,243 * 0,000000 + 52,461 * 0,009149 = 0,479965689
3. Биологическая единица записи 1PNR
C сервера PDBe получим файл с биологической единицей записи 1PNR. Сравним биологическую и асимметрическую единицы комплекса.Асимметрическая единица представляет собой одну белковую цепь, связанную с одной цепью ДНК:
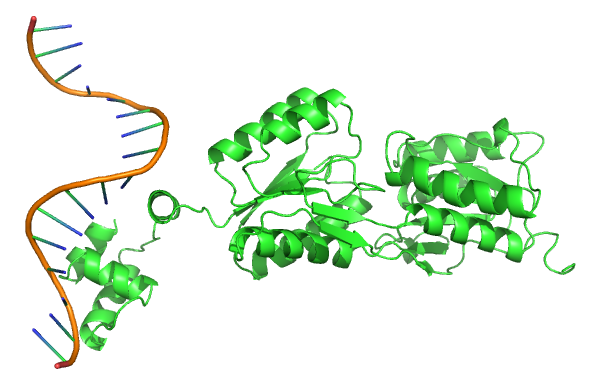
Биологическая единица, в свою очередь, является белковым димером, который взаимодействует с двуцепочечной ДНК (грубо говоря, биологическая единица является удвоенной асимметрической):
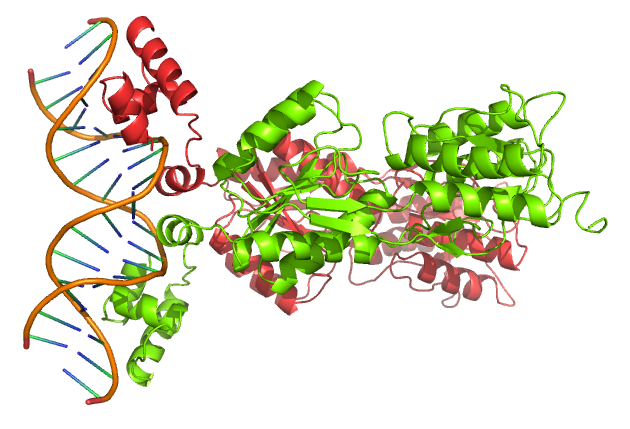
Анализируя структуру одной лишь асимметрической единицы, нельзя правильно рассмотреть контакты между двумя асимметрическими единицами в биологической. Кроме того, сложно также говорить о взаимодействии белка с ДНК, поскольку в природе у E.coli, которой принадлежит белок, ДНК двуцепочечна. Также, очевидно, взаимодействие двух полипептидных цепей влияет на связи всего белкового комплекса с ДНК.
Назад