2. Программы DSSP и HBplus
1. Изучение вторичной структуры белка 1DUV
PDB-файл 1DUV.pdb содержит информацию о трёх полипептидных цепях (G, H, I). Для определения вторичной структуры он был подан на вход программе DSSP:dsspcmbi 1DUV.pdb 1duv.dsspРезультат: 1duv.dssp.
Для каждой цепи DSSP нашёл 13 альфа-спиральных участков (блоки идущих подряд букв Н) и 9 бета-тяжей (блоки букв Е).
Сравним результаты работы DSSP с данными из PDB-файла на примере цепи G:
| α-спирали | β-тяжи | ||
| DSSP | PDB | DSSP | PDB |
| - | 10 - 14 | 46 - 51 | 46 - 51 |
| 16 - 34 | 15 - 35 | 72 - 76 | 72 - 76 |
| 57 - 68 | 56 - 69 | 102 - 106 | 102 - 106 |
| 89 - 96 | 88 - 97 | 124 - 126 | 124 - 126 |
| 110 - 120 | 109 - 121 | 157 - 161 | 157 - 161 |
| 134 - 147 | 133 - 148 | 182- 186 | 182 - 186 |
| - | 152 - 155 | 211 - 215 | 211 - 215 |
| 167 - 179 | 166 - 180 | 227 - 230 | 227 - 230 |
| - | 188 - 192 | 269 - 272 | 269 - 272 |
| 195 - 207 | 194 - 208 | ||
| 218 - 222 | 217 - 223 | ||
| 242 - 250 | 241 - 251 | ||
| - | 252 - 254 | ||
| 257 - 261 | 256 - 262 | ||
| 283 - 291 | 282 - 292 | ||
| 302 - 305 | 301 - 306 | ||
| 312 - 331 | 311 - 332 | ||
Как видно из таблицы, границы бета-тяжей, определённые DSSP, совпали с данными авторов PDB-файла.
Все предсказанные DSSP альфа-спиральные участки тоже совпали с указанными в PDB (совпадение не совсем точное, т.к. DSSP не включает в состав альфа-спирали граничные остатки). Участки 10-14, 152-155, 188-192 и 252-254, описанные в PDB как альфа-спирали, DSSP предсказал как 3/10-спирали.
2. Анализ торсионных углов φ по результатам работы DSSP
Остатки, имеющие положительное значение угла φ:| Цепь G | Цепь Н | Цепь I | |||||
| гистидин 5 | 57,8 | гистидин 5 | 60,3 | гистидин 5 | 59,4 | ||
| глицин 36 | 68,7 | глицин 36 | 73,6 | глицин 36 | 76,9 | ||
| глицин 44 | 87,3 | глицин 44 | 84,1 | глицин 44 | 84,4 | ||
| глицин 70 | 83,5 | глицин 70 | 83,2 | глицин 70 | 79,9 | ||
| глицин 84 | 75,0 | глицин 84 | 82,6 | глицин 84 | 96,0 | ||
| серин 121 | 72,2 | серин 121 | 69,6 | серин 121 | 70,4 | ||
| глицин 150 | 54,9 | глицин 150 | 63,5 | глицин 150 | 79,9 | ||
| глицин 180 | 87,1 | глицин 180 | 87,8 | глицин 180 | 76,2 | ||
| глицин 209 | 91,5 | глицин 209 | 88,5 | глицин 209 | 85,0 | ||
| глицин 224 | 82,4 | глицин 224 | 76,1 | глицин 224 | 74,9 | ||
| глицин 237 | 91,6 | глицин 237 | 97,2 | глицин 237 | 92,6 | ||
| глицин 264 | 71,9 | глицин 264 | 68,5 | глицин 264 | 76,7 | ||
| лейцин 274 | 72,3 | лейцин 274 | 71,3 | лейцин 274 | 73,3 | ||
| глицин 293 | 58,6 | глицин 293 | 56,2 | глицин 293 | 64,6 | ||
| глицин 296 | 79,2 | глицин 296 | 86,6 | ||||
| глицин 2 | 98,6 | ||||||
| лизин 333 | 57,2 | ||||||
Большинство перечисленных остатков отнесено DSSP к поворотам и изгибам (turn, bend).
Среди остатков с положительным значением φ преобладают глицины. Во всех трёх цепях представлен также Leu274, который описан в поле REMARK PDB-файла как остаток с неожиданными значениями торсионных углов.
Рассмотрим цепь I:
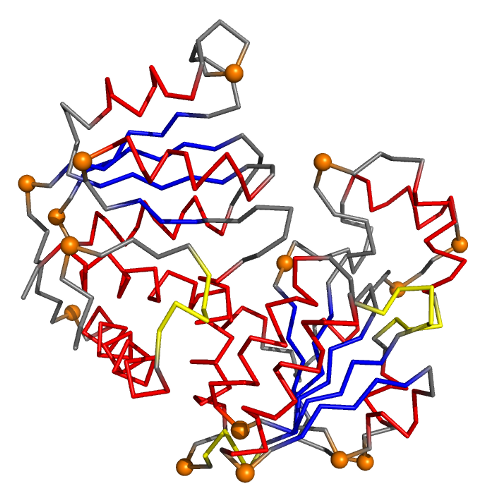
На изображении красным выделены предсказанные DSSP альфа-спирали, синим - бета-тяжи, оранжевым - остатки с положительным углом φ (шариками показаны СА-атомы). Жёлтым отмечены 310-спирали (описанные в PDB-файле как альфа-спирали).
3. Определение водородных связей программой HBplus
Для работы в HBplus необходимо оставить в PDF-файле одну цепь белка. Возьмём цепь G. Файл выдачи программы: 1DUVg.hb2. Всего было найдено 1682 водородных связи. Изучим их подробнее.а) Пример водородной связи, участвующей в стабилизации вторичной структуры:
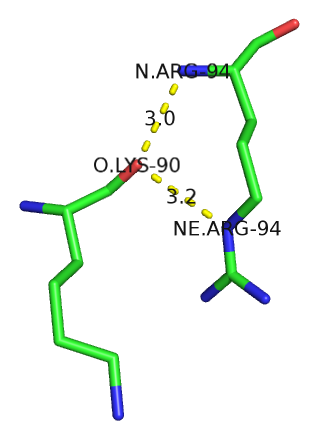
Между атомом кислорода лизина-90 и остатком аргинина-94 возможны даже два полярных контакта. Эти остатки участвуют в образовании альфа-спирали, находясь друг под другом в соседних витках (разница в нумерации составляет 4).
Для отображения водородных связей в PyMOL нужно нажать кнопку А в меню выделенного множества, затем find > polar contacts > within selection.
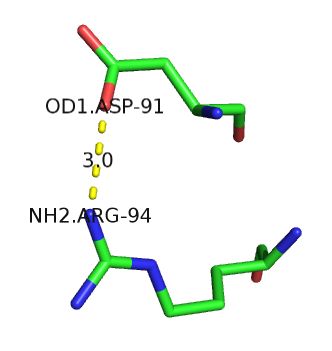
б) Пример водородной связи между боковыми цепями аминокислотных остатков:
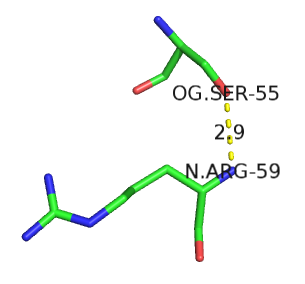
в) Пример водородной связи между боковой цепью одного остатка и остовным атомом другого:
Сюда также подходит пример из пункта а), поскольку там приведена водородная связь между остовным атомом кислорода лизина-90 и атомом азота боковой цепи (NE) аргинина-94.
г) Пример водородной связи между белком и лигандом (малой органической молекулой):
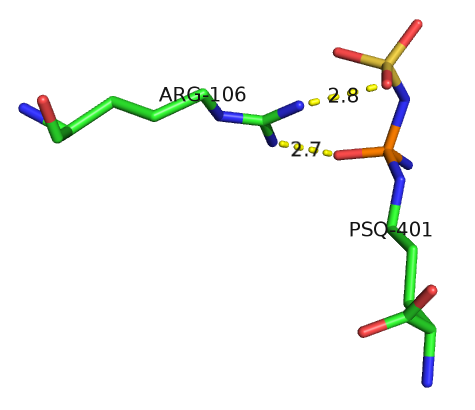
На изображении показаны две водородные связи между аргинином-106 и лигандом орнитином
(если точнее, то Nδ-(N'-сульфодимаинофосфинил)-L-орнитином, в PDB-записи он обозначен как PSQ).
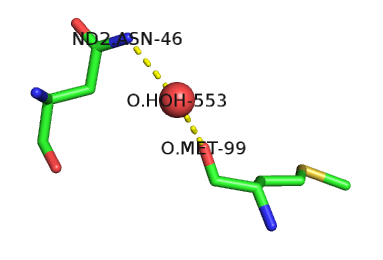
д) Пример водяного мостика:
Назад