10. Поверхность, гидрофобное взаимодействие, домены
1. Изучение поверхностей контактов димера пуриновых репрессоров 1PNR
Файл с биологической единицей пуринового репрессора: 1pnr_b.pdb.а) Изображение поверхности контакта мономера белка с симметричным мономером на фоне остовной (ribbon) модели мономера:
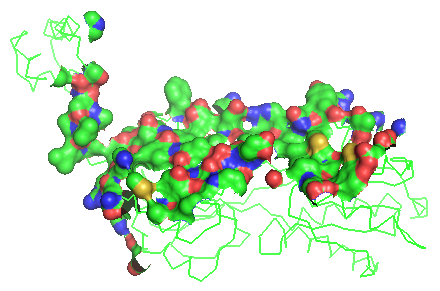
Поверхность контакта была построена с помощью следующей последовательности команд:
create dimer,chain a+d select contact, chain a & (chain d around 5) remove chain d show surface, contact show ribbon,chain a set surface_quality,2 |
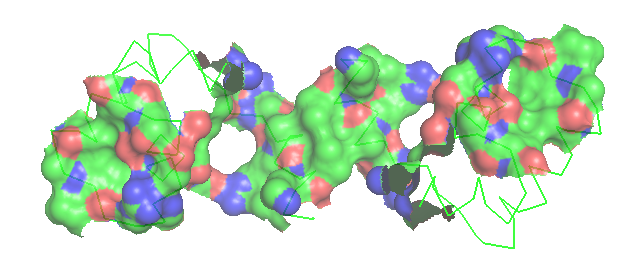
б) Изображение поверхности контакта димера белков с двойной спиралью ДНК на фоне остовной модели части белка, вовлечённой в контакт:
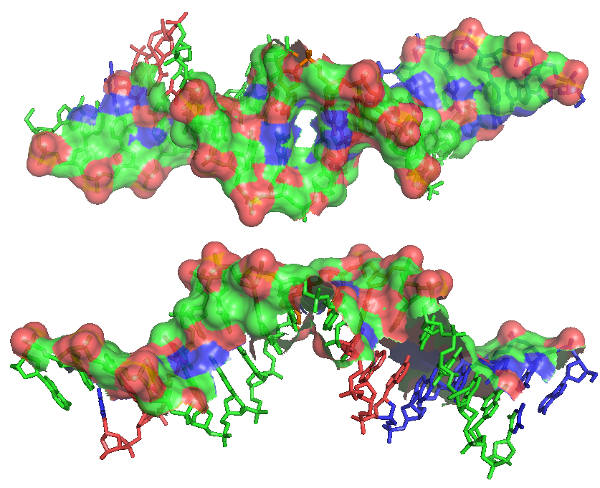
в) Изображение поверхности контакта ДНК с димером белков на фоне проволочной (sticks) модели двойной спирали:
2. Работа с сервисом PROTORP
Пользуясь сервисом PROTORP, найдем площадь контакта мономеров белка из упражнения 1. Для этого нужно загрузить файл с биологической единицей белка через поле Option 3. Нам нужен контакт между цепями A и D.Исходя из выдачи программы, площадь поверхности контакта равна 2576,97Ų. На гидрофобные взаимодействия (взаимодействия между неполярными атомами) приходится 43.59% площади, то есть 1123,3Ų.
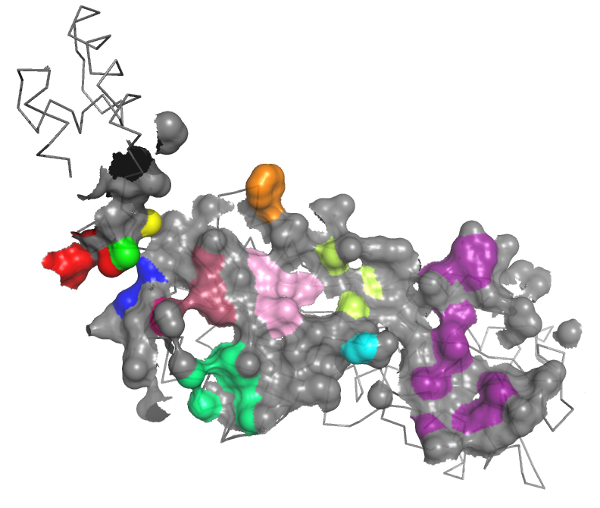
3. Работа с сервисом CluD
Пользуясь сервисом CluD, определим гидрофобные кластеры на интерфейсе мономеров исследуемого белка объёмом не менее 10 атомов.CluD выдаёт нам файл со списком атомов, которые образуют гидрофобные кластеры. Из этого файла был получен скрипт для PyMOL, который красит все атомы из списка в жёлтый цвет. В результате работы скрипта получилось изображение поверхности контакта между белковыми мономерами. Полученные в CluD кластеры размечены разными цветами:
4. Работа с сервисом pDomains
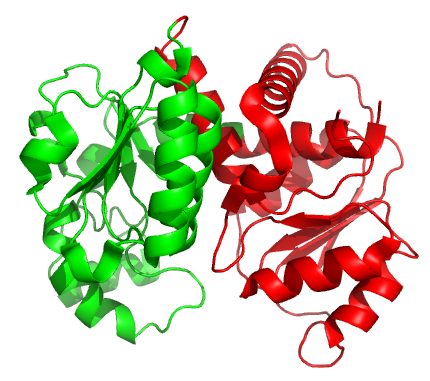
На сервисе pDomains определим доменную структуру:a) цепи G белка 1DUV.
Практически все методы выделили два домена (кроме DDomain, который обнаружил всего один). Если смотреть на пространственную структуру цепи, домены хорошо различимы:
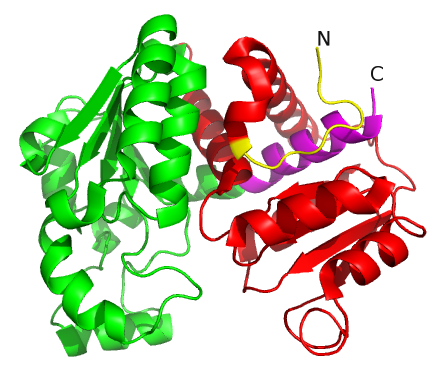
В этом случае нельзя выделить N-концевой и C-концевой домены, поскольку домен, окрашенный красным, содержит и N, и С-конец. Проиллюстрируем это:
Поэтому некоторые методы давали такой вариант разделения на домены:
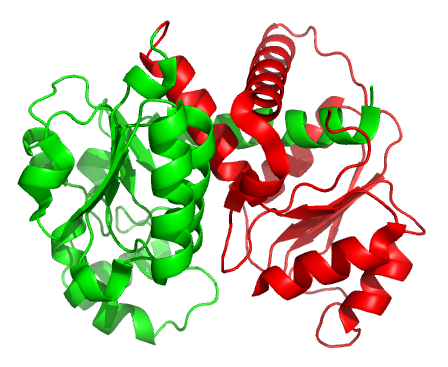
Как видно, С-концевая альфа-спираль цепи располагается внутри N-концевого домена. Возможно, поэтому метод Ddomain эту полипептидную цепь вообще на домены не разделил.
б) цепи белка из упр.1. Будем рассматривать цепь А PDB-файла 1PNR.
В этом случае вариантов доменов уже больше:
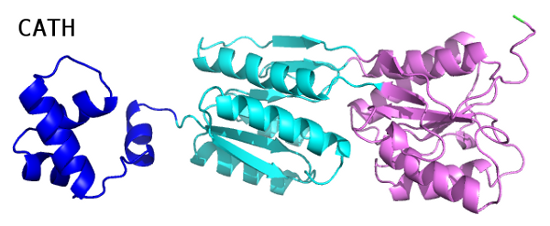
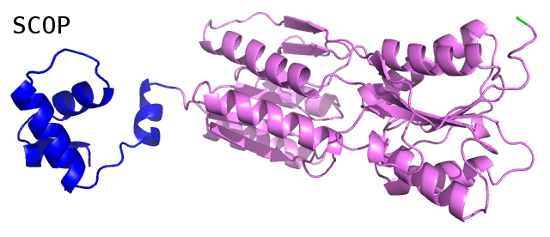
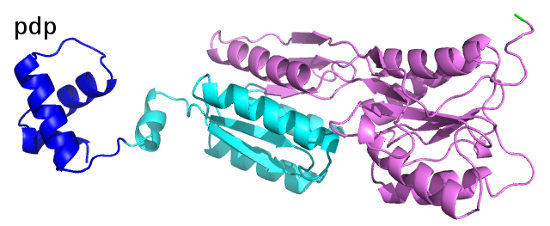
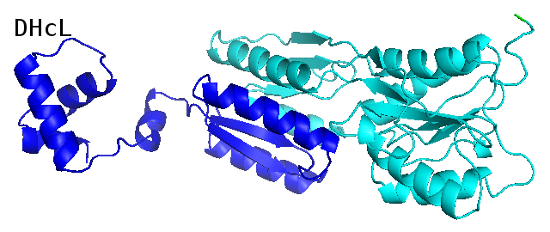
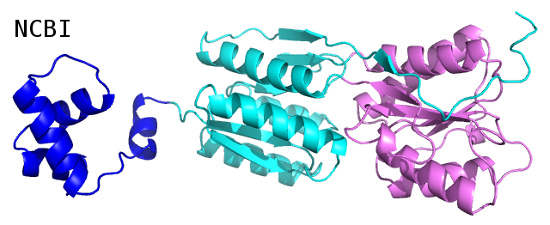
Наиболее чётко почти все методы выделили N-концевой домен (выделен тёмно-синим). Он является ДНК-связывающим:
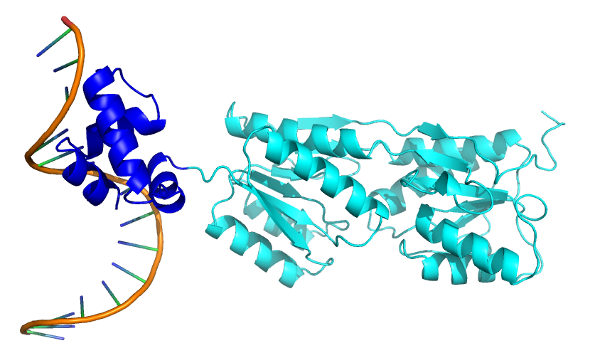
С остальной частью белка дело обстоит сложнее. Если разделить её на два пространственных домена, то не получится выделить непрерывные домены в белковой последовательности, поскольку, как и в случае с белком 1DUV, цепь уложена в петли, которые проходят через оба домена. Поэтому, на мой взгляд, наиболее правильным является деление всей цепи не на три, а на два домена (N и С-концевой), полученное методом SCOP.
Назад