1
Работа в командной строке Linux
При выполнении команды seqret sw:p04391 -auto в активной директории создается файл с названием otc1_ecoli.fasta, который представляет собой последовательность белка OTC1_ECOLI в fasta-формате. При выполнении команды more otc1_ecoli.fasta файл просматривается. При выполнении команды entret sw:p04391 -auto в активной директории создается файл otc1_ecoli.entret. При вводе команды more otc1_ecoli.entret он также просматривается. Это запись UniProt о моем белке.
Клавиши "стрелка вверх" и "стрелка вниз" вводят в командную строку команды, введенные ранее/позднее. Команда history выводит полный список всех ранее использованных пронумерованных команд. При вводе more S и последующем нажатии Tab автоматически вводится название файла Student_1.jpg, лежащего в директории. Нажатием Enter можно этот файл открыть.
2
Построить и сравнить оптимальные глобальные выравнивания 2-х последовательностей
needle otc1_ecoli.fasta otc_metmp.fasta alignment1.needle -auto
Этот файл содержит информацию о выравнивании (названия выравниваемых последовательностей, длина выравнивания, штрафы за гэпы, количество гэпов, вес выравнивания и т.д.), далее приведено само выравнивание. Штрафы за гэпы равны: gap_penalty (штраф за новый гэп) = 10.0, extend_penalty (штраф за новые пробелы в гэпе) = 0.5.
Затем создадим еще одно выравнивание, увеличив штрафы за гэпы в два раза (gap_penalty = 20.0, extend_penalty = 1.0). Сохраним полученные выравнивания в формате .msf, импортируемом в GeneDoc, и сравним их.
Полученные выравнивания оказались абсолютно одинаковыми:
Выравнивание 1 (штрафы за гэпы заданы по умолчанию):
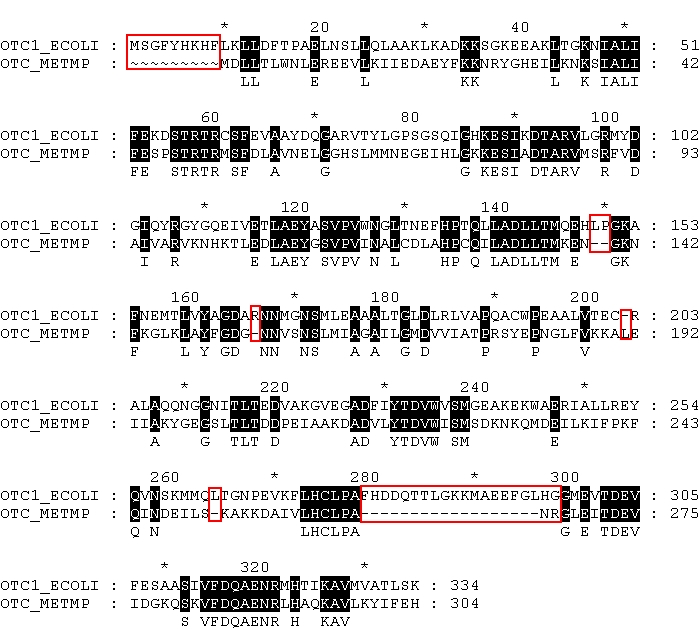
Выравнивание 2 (штрафы за гэпы увеличены вдвое):
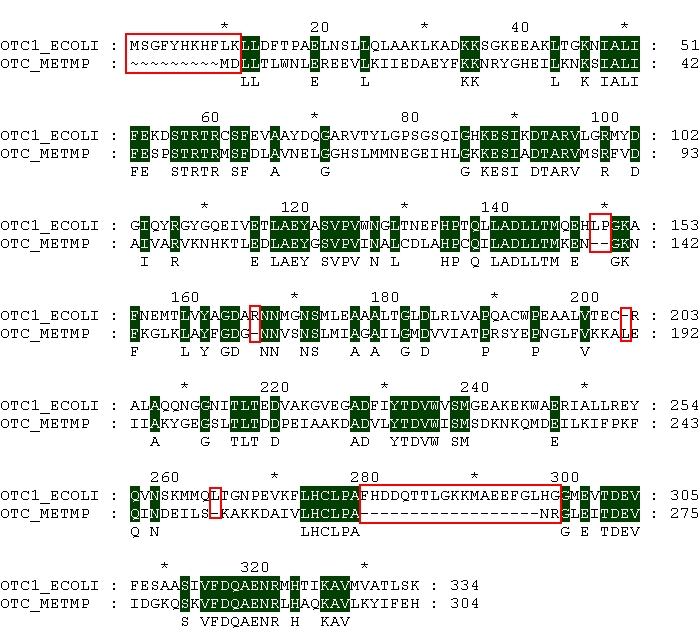
Видно, что в выравниваниях сопоставляются одни и те же участки последовательностей, гэпы находятся на аналогичных позициях (два больших гэпа занимают колонки 1-9 и 278-295, а также отдельные пробелы на одних и тех же позициях). На рисунках гэпы отмечены красным.
Tаким образом, в этих выравниваниях нет примеров того, что одной и той же позиции первой последовательности в разных глобальных выравниваниях сопоставлены разные позиции второй последовательности, и того, что в одном глобальном выравнивании какой-либо позиции первой последовательности сопоставлена некоторая позиция второй, а в другом выравнивании против той же позиции оказался пропуск.
Интересно, что при выравнивании первая последовательность (белок OTC1_ECOLI, рассматриваемый мной в течение семестра) сохранена практически без изменений (есть только один гэп длиной в один аминокислотный остаток), в то время как последовательность белка OTC_METMP имеет два больших разрыва и несколько маленьких. Это позволяет предположить, что в процессе эволюции при образовании рассматриваемых белков произошла делеция либо вставка.
3
Построить и сравнить оптимальные локальные выравнивания 2-х последовательностей
1)gap_penalty = 10.0, extend_penalty = 0.5 (заданы по умолчанию);
2)gap_penalty = 20.0, extend_penalty = 1.0 (увеличены вдвое);
3)gap_penalty = 5.0, extend_penalty = 0.25 (уменьшены вдвое).
Рассмотрим эти выравнивания, предварительно сохранив их в формате .msf:
Выравнивание 1 (штрафы заданы по умолчанию):
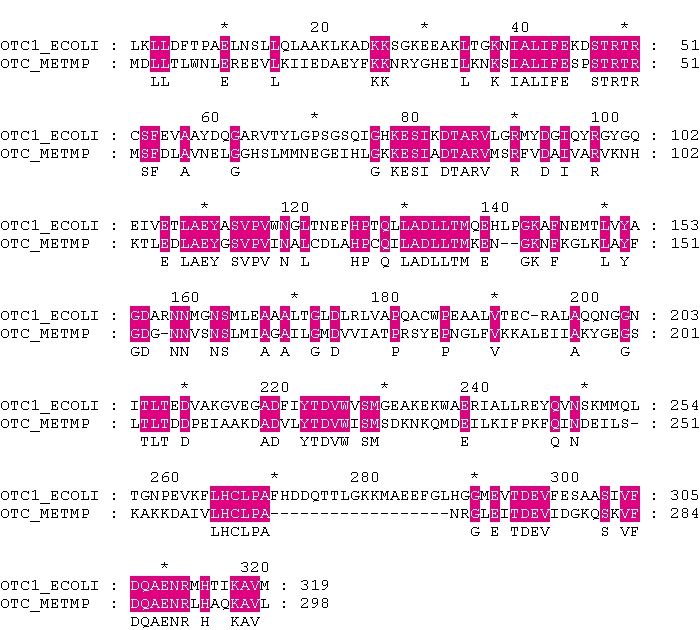
Выравнивание 2 (штрафы увеличены вдвое) снова оказалось таким же, как и первое, где штрафы были заданы по умолчанию. Зато выравнивание 3 (штрафы уменьшены вдвое) заметно отличается от двух первых:
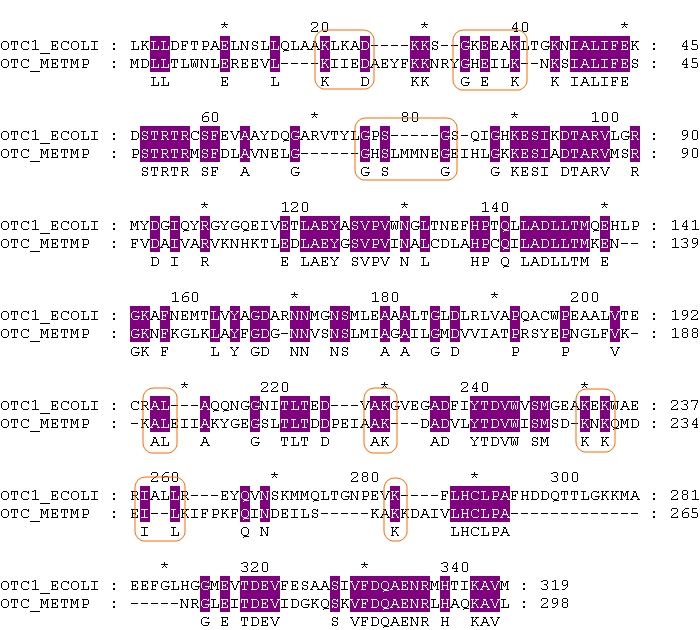
Сразу видно, что в этом выравнивании количество совпадающих аминокислотных остатков больше, чем в первом и втором (совпадения, отсутствующие в двух предыдущих выравниваниях, выделены на рисунке оранжевым). Увеличилось и число гэпов. Это произошло потому, что в первых двух выравниваниях из-за больших штрафов за гэпы многие аминокислотные остатки программа предпочитала не сопоставлять, так как прибавка за совпадение этих остатков не покрывала штраф за гэпы, образующиеся при их сопоставлении.
Приведем пример того, что одной и той же позиции первой последовательности в разных локальных выравниваниях сопоставлены разные позиции второй последовательности (сравниваем 1-е и 3-е выравнивания):
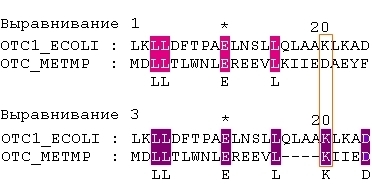
OTC1_ECOLI 10 LKLLDFTPAELNSLLQLAAKLKAD
:.||.....|...:|::....:..
OTC_METMP 1 MDLLTLWNLEREEVLKIIEDAEYF
OTC1_ECOLI 10 LKLLDFTPAELNSLLQLAAKLKAD
:.||.....|...:| |:..|
OTC_METMP 1 MDLLTLWNLEREEVL----KIIED
В первом выравнивании позиции 20 последовательности OTC1_ECOLI сопоставлена 20-я же позиция OTC_METMP, а во третьем - 16-я позиция этой последовательности.Теперь приведем пример того, что в одном локальном выравнивании какой-либо позиции первой последовательности сопоставлена некоторая позиция второй, а в другом выравнивании против той же позиции оказался пропуск:
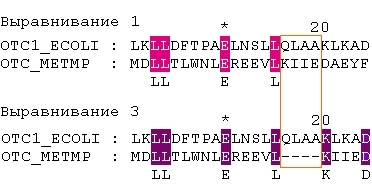
(текстовый вариант этого участка представлен выше)
В первом выравнивании сопоставлены 16-19-е позиции двух последовательностей, в третьем этим же позициям первой последовательности соответствуют пропуски второй.
Все оптимальные локальные выравнивания, построенные с использованием разных параметров, соответствуют одним и тем же фрагментам последовательностей. Кроме того, локальные выравнивания совпадают с соответствующими частями глобальных выравниваний (при равных штрафах за гэпы), так как вид выравнивания зависел именно от штрафов.
4*
Карта локального сходства заданных последовательностей
dotmatcher otc1_ecoli.fasta otc_metmp.fastа dothmatcher.ps
Полученный файл можно открыть с помощью какой-нибудь программы для просмотра файлов PostScript (я использовала PostScript Viewer). Затем сохраним изображение в формате .jpg
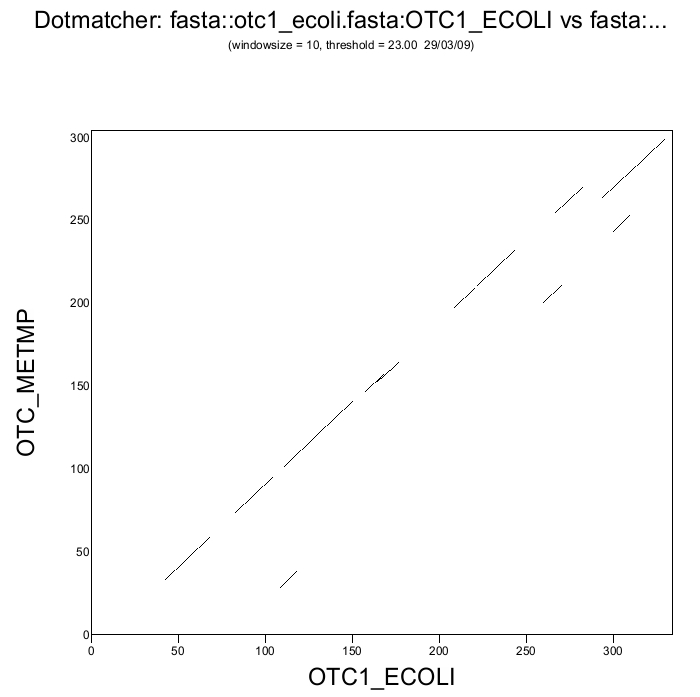
Белки довольно схожи (об этом говорит множество линий на диагонали карты, то есть имеются похожие участки на одних и тех же позициях). Заметим, что последовательности имеют разную длину, но их концы совпадают (на карте этот участок выбивается из общей линии схожих участков). Сдвиг этого участка относительно общей линии позволяет предположить, что в этом месте произошла делеция либо вставка аминокислотных остатков (позициям ~280-295 последовательности OTC1_ECOLI не соответствуют никакие остатки последовательности OTC_METMP). Это предположение уже высказывалось при рассмотрении глобальных выравниваний, и если мы посмотрим на изображение любого из них, то увидим, что как раз участку 278-295 последовательности OTC1_ECOLI соответствует гэп в OTC_METMP:
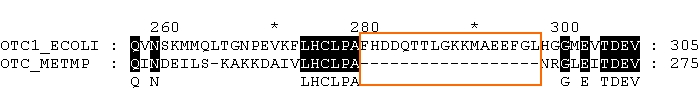
5*
Субоптимальные локальные выравнивания 2-х последовательностей
matcher otc1_ecoli.fasta otc_metmp.fastа suboptimal.matcher -alternatives 5
Просмотреть все 5 выравниваний можно в файле suboptimal.matcher
Во всех полученных выравниваниях фрагменты последовательностей не совпадают с выровненными фрагментами в оптимальном выравнивании. Возможно, это из-за разных значений штрафов за гэпы (в программе matcher по умолчанию установлены значения gap_penalty = 14, extend_penalty = 4). Так как значения штрафов велики, программа стремится уменьшить количество пропусков. Рассмотрим выравнивания, обладающие наибольшим весом:
1)Вес выравнивания 29, длина 23.
290 300
OTC1_E KKMAEEFGLHGGMEVTDEVFESA
:.: : . .. ::. :
OTC_ME KQMDEILKIFPKFQINDEILSKA
240 250 2)Вес выравнивания 27, длина 27.
270 280 290
OTC1_E KFLHCLPAFHDDQTTLGKKMAEEFGLH
. : : : . : : . ::
OTC_ME EILKIFPKFQINDEILSKAKKDAIVLH
240 250 260 Далее следуют выравнивания с весом, также равным 27. Среди них есть любопытное выравнивание, в котором сопоставлены очень далеко находящиеся друг от друга позиции двух последовательностей. Длина этого выравнивания равна 12:
220
OTC1_E EDVAKGVEGADF
:.: : .: :..
OTC_ME EEVLKIIEDAEY
20 Таким образом, программа matcher старается не "рвать" последовательности, она их двигает и ищет наиболее схожие их участки, причем неважно, в начале или конце последовательностей они находятся.
к проектам
на главную