Составление выборки аминокислотных последовательностей.
С помощью SRS в БД UniProt найдены аминокислотные последовательности:
- белки семейства MIP (PF00230) из таксонов Bacillales, Rhizobiales;
- все белки семейства MIP (PF00230) из царства архей (Archaea);
- как внешняя группа — Q65YQ3_CHICK, Q5W916_BOMMO, AQP3_HUMAN.
Все последовательности перенесены в один файл seq.txt
Получен также список организмов, чьи последовательности использовались в работе.
Он представлен в файле taxon_names.txt
Построение филогенетического дерева
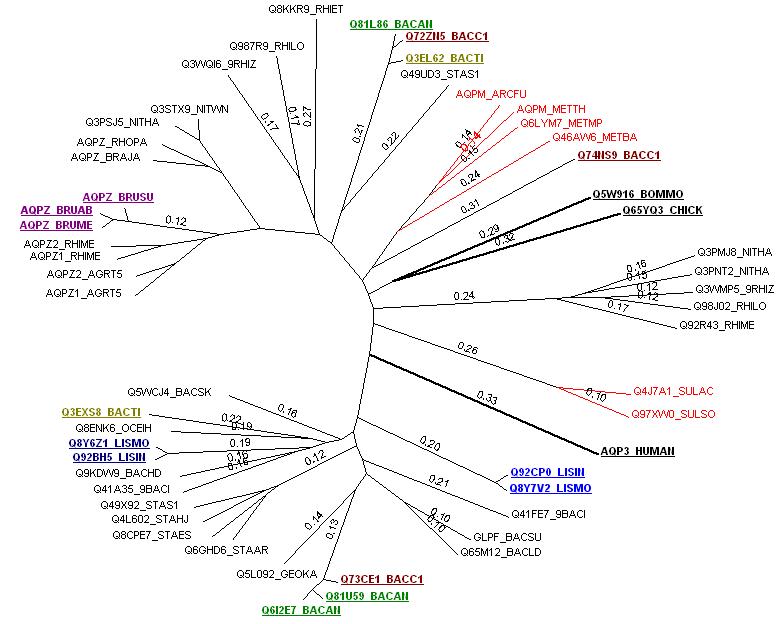
Построено множественное выравнивание (программа ClustalW). По данному множественному выравнивания построено
дерево (программа Proml пакета Phylip). Визуализация произведена с помощью программы GeneMaster.
На дереве раскрашены в красный цвет ветви, и выделены красным шрифтом названия соответствующие
белкам архей. Толстые ветви, подчеркнутые и полужирные названия соответствуют белкам
внешней группы — Q65YQ3_CHICK, Q5W916_BOMMO, AQP3_HUMAN. Другие обозначения см. ниже.
файл gdt
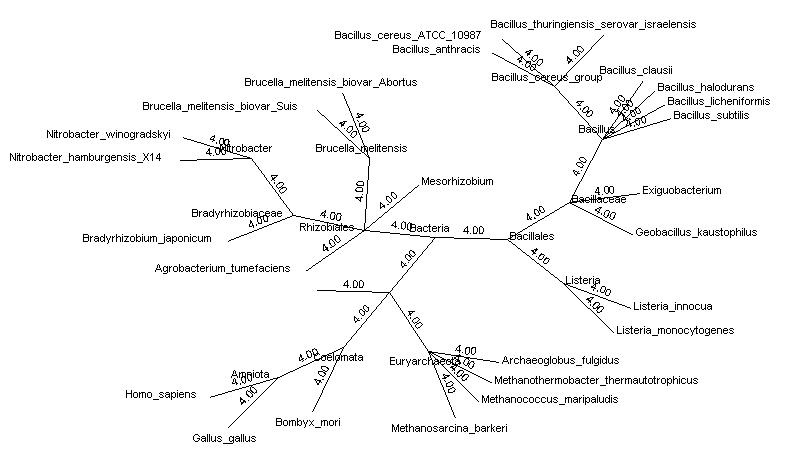
Построение таксономического дерева
С помощью сервера NCBI было получено таксономическое дерево. В дереве представлены организмы,
чьи аминокислотные последовательности использовались.
По скобочной формуле с помощью программы GeneMaster дерево было визуализировано.
файл gdt
Анализ дерева.
Рассмотрим сначала паралоги. То есть гомологичные аминокислотные последовательности
из одного организма; в основном появление которых связано с дупликацией гена у предкового вида и
дальнейшей дивергентной эволюцией. Яркими представителями паралогов в данной выборке
являются белки Q6I2E7_BACAN, Q81L86_BACAN, Q81U59_BACAN. На дереве выделены темно-зеленым
цветом. Видно, что данные паралоги близки друг к другу не одинаково. Последовательности
Q6I2E7_BACAN и Q81U59_BACAN ближе друг к другу, чем к третьей последовательности
из этого же организма - Q81L86_BACAN. Но все же их можно всех считать паралогами.
Аналогично, обстоят дела с тремя последовательностями
из одного организма - Q72ZN5_BACC1, Q73CE1_BACC1, Q74NS9_BACC1 (названия выделены
малиновым цветом). В отличии от предыдущего случая последовательности достаточно
эволюционно удалены друг от друга. Сложно сказать какая последовательность,
к какой ближе. Интересно, что один из белков ближе к белкам из архей, чем к
любому из бактериальных белков.
Последовательностями Q3EL62_BACTI, Q3EXS8_BACTI так же являются паралогами (названия
написаны оливковым цветом). Судя по расстоянию это не очень близкие паралоги.
На право называться паралогами также претендуют следующие пары (тройки)
последовательностей:
AQPZ1_AGRT5, AQPZ2_AGRT5
Q41A35_9BACI, Q41FE7_9BACI
Q3WMP5_9RHIZ, Q3WQI6_9RHIZ
Q3PMJ8_NITHA, Q3PNT2_NITHA, Q3PSJ5_NITHA
Q987R9_RHILO, Q98J02_RHILO
AQPZ1_RHIME, AQPZ2_RHIME, Q92R43_RHIME
Q49UD3_STAS1, Q49X92_STAS1 + см. ниже.
Рассмотрим теперь возможные ортологи. То есть гомологичные последовательности из
разных организмов, обладающие общей предковай последовательностью. Как правило,
могут образовывать подсемейства в семействе. На дереве выделено несколько пар
возможных ортологов. Выделенная сиреневым цветом тройка белков AQPZ_BRUAB,
AQPZ_BRUME, AQPZ_BRUSU из разных организмов обладает не плохой схожестью (не малую роль в этом
играет близость организмов - все принадлежат одному роду BRUCELLA)и в праве может
считаться ортологами. Выделенная темно-синим, пара последовательностей Q92BH5_LISIN,
Q8Y6Z1_LISMO так же может быть парой ортологов. Аналогично, и пара Q92CP0_LISIN, Q8Y7V2_LISMO
(светло-синий шрифт). Можно заметить, что пары Q92BH5_LISIN-Q92CP0_LISIN и
Q8Y7V2_LISMO-Q8Y7V2_LISMO являются паралогами.
В целом все последовательности семейства можно разбить на подсемейства. Каждое подсемейство
образовано своей группой паралогов.
- Архейные белки можно выделить в отдельную группу. Странно, но сюда же относится
один белок Q74NS9_BACC1.
© Тихонов Максим, 2006
|


