- Краткое описание структуры в файле 1PP7.pdb
В файле приведены координаты атомов следующих молекул:
-
1. 39 kDa initiator binding protein (из Trichomonas vaginalis) - 1 молекула
2. Ferredoxin инициаторный элемент(искусственно синтезирована) - 1 молекула (2 цепи)
3. Ионы цинка - 7 ионов
4. Вода - 65 молекул
F 5' [15] CAAGTGAAGTAAC [3] 3' | |||||| | | E 3' [25] GGTTACTTCACTT [37] 5'
где 3 и 37 - номера первого и последнего нуклеотида.
- Функции белка, структура которого представлена в файле 1PP7.pdb
В соответствующем документе UniProt, можно найти описание функций
(и особенностей) белка:
Код доступа ("Accession number") Q95VR4 Идентификатор записи в БД Q95VR4_TRIVA Название (краткое описание) белка 39 kDa initiator binding protein
EC=6.1.1.-;Длина: 341 AA Организм: Trichomonas vaginalis Идентификаторы записей PDB 1PP7; 1PP8; 1Q87; 1Q88; 1Q89 Примечания: Статус белка в базе: Unreviewed - Исследование структуры ДНК
С помощью программ find_pair и analyze
я смогла определить тип формы ДНК: правозакрученная B-ДНК. А с помощью Excel я определила средние значения торсионных углов
для внутренних нуклеотидов (для всех, кроме краевых) и номер самого "кривого" нуклеотида со значениями торсионных углов, наиболее отклоняющимися от средних.
- С34, цепь Е (файл с рассчетами).
- Исследование природы ДНК-белковых контактов
1. Скрипт my_dna.def
2. Таблица. Контакты разного типа в комплексе 1PP7.pdbКонтакты атомов белка с Полярные Неполярные Всего остатками 2'-дезоксирибозы 2 22 24 остатками фосфорной кислоты 4 11 15 остатками азотистых оснований со стороны большой бороздки 1 - 1 остатками азотистых оснований со стороны малой бороздки - 5 5
3. Можно заметить большое количество неполярных контактов белка с сахарофосфатным остовом. Это можно объяснить тем, что остовные атомы располагаются на поверхности молекулы ДНК.
Меньше всего контактов с остатками азотистых оснований со стороны большой бороздки, так как они находятся глубоко внутри молекулы и являются довольно труднодоступными.
- Получение популярной схемы ДНК-белковых контактов с помощью nucplot
Для начала переводим файл .pdb в старый формат:
remediator --old 1PP7.pdb > 1PP7_old.pdb
Затем создаём отдельную папку для выходных файлов программы nucplot и из неё запускаем программу:nucplot ../1PP7_old.pdb
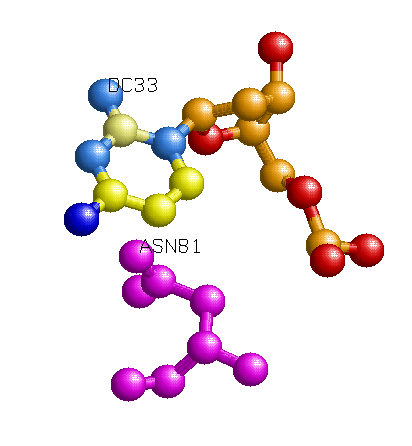
- Возможный распознающий контакт.
Я думаю, что одним из наиболее вероятных распознающих контактов является контакт С33 и Asn81, так как это один из немногих прямых контактов между остатком азотистого основания и аминокислотным остатком.
- Характеристика ДНК-связывающего домена Q95VR4_TRIVA
Доменная структуру белка из исследуемого комплекса была определена с помощью инструментов Pfam:
Transcription-initiator DNA-binding domain IBD

<<Обратно на третий семестр
<<Обратно на главную страницу
©Лелекова Мария,2009