Реконструкция и сравнение деревьев
Расстояния между последовательностями.
-
� Пользуясь таксономическим сервисом NCBI: http://www.ncbi.nlm.nih.gov/taxonomy/ ,
определите, к каким таксонам относятся отобранные в предыдущем задании бактерии.
Название Taxonomy Bradyrhizobium japonicum Bacteria; Proteobacteria; Alphaproteobacteria; Rhizobiales; Bradyrhizobiaceae; Bradyrhizobium Agrobacterium tumifaciens Bacteria; Proteobacteria; Alphaproteobacteria; Rhizobiales; Rhizobiaceae; Rhizobium/Agrobacterium group; Agrobacterium Rhizobium etli Bacteria; Proteobacteria; Alphaproteobacteria; Rhizobiales; Rhizobiaceae; Rhizobium/Agrobacterium group; Rhizobium Rhodobacter sphaeroides Bacteria; Proteobacteria; Alphaproteobacteria; Rhodobacterales; Rhodobacteraceae; Rhodobacter Burkholderia cenocepacia Bacteria; Proteobacteria; Betaproteobacteria; Burkholderiales; Burkholderiaceae; Burkholderia; Burkholderia cepacia complex Ralstonia pickettii Bacteria; Proteobacteria; Betaproteobacteria; Burkholderiales; Burkholderiaceae; Ralstonia Neisseria meningitidis Bacteria; Proteobacteria; Betaproteobacteria; Neisseriales; Neisseriaceae; Neisseria
Теперь наложим таксономию данных бактерий на филогенетическое дерево из предыдущего задания:
�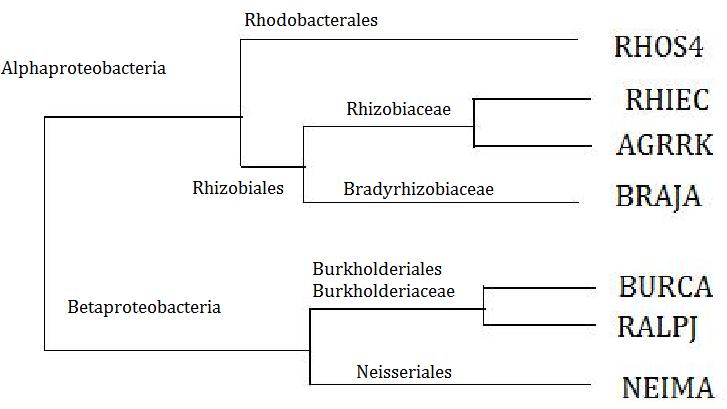
Отсюда видим, что привиденные бактерии делятся на две большие таксономические группы (класса): Alphaproteobacteria и Betaproteobacteria. В свою очередь класс Alphaproteobacteria делится на два порядка: Rhizobiales и Rhodobacterales. К последнему относится семейство Rhodobacteraceae, род Rhodobacter, вид Rhodobacter sphaeroides.
К порядку Rhizobiales относятся две из предложенных бактерий: семейство Bradyrhizobiaceae, род Bradyrhizobium, вид Bradyrhizobium japonicum; семейство Rhizobiaceae, два рода: род Agrobacterium, вид Agrobacterium tumifaciens, род Rhizobium, вид Rhizobium etli.
Класс Betaproteobacteria включает в себя два порядка: Burkholderiales и Neisseriales. Последний включает в себя: семейство Neisseriaceae, род Neisseria, вид Neisseria meningitidis. Порядок Burkholderiales включает в себя семейство Burkholderiaceae и два рода: род Ralstonia, вид Ralstonia pickettii и род Burkholderia, вид Burkholderia cenocepacia.
Подытожим:
Данное филогенетическое дерево полностью описывает таксономию выбранных бактерий. - Белки для дальнейшей реконструкции филогенетического дерева
Получим из Swiss-Prot последовавтельности белков с функцией энолазы из данных бактерий.
Для этого воспользуемся командой seqret:
seqret sw:ENO_ABC
(ABC - мнемоника бактерии)
Теперь создадим выравнивание эиих последовательностей программой muscle. Вот, чо получилось: eno.fasta
Импортируем файл в GeneDoc и найдем диагностические позиции выравнивания. - Реконструкция дерева программой fprotpars по белку ENO
� Проведем реконструкцию дерева программой fprotpars. Для этого воспользуемся
программой fprotpars. На вход даем файл с выравненными последовательностями
и получаем на выходе два файла:
eno.fprotpars и
eno.treefile. Вот, что мы видим:
((ENO_RHOS4:0.17016,(ENO_BRAJA:0.14518,(ENO_RHIEC:0.04929, ENO_AGRRK:0.04929):0.09589):0.02498):0.05774,(ENO_NEIMA:0.17095, (ENO_BURCA:0.04851,ENO_RALPJ:0.04851):0.12244):0.05695);
+--ENO_RALPJ +--6 +--------5 +--ENO_BURCA ! ! ! +-----ENO_NEIMA +--4 ! ! +--ENO_AGRRK ! ! +--3 1 +--------2 +--ENO_RHIEC ! ! ! +-----ENO_BRAJA ! +-----------------ENO_RHOS4Программа выдала нам неукорененное "бережливое" дерево без длин ветвей. В данном случае мы получили одно дерево (хотя их может быить и больше, "одинаково бережливых"). Сравнивая его с предыдущим, можно заметить, что они одинаковые.
�
- Эволюционные расстояния
Оценим эволюционные расстояния между последовательностями.
Для этого воспользуемся программой fprotdist.
На вход даем файл с выровненными последовательностями, а на выходе
получаем файл eno.fprotdist.
В итоге мы види матрицу расстояний:
7 ENO_RHOS4 0.000000 0.377800 0.310072 0.333097 0.423113 0.460577 0.444920 ENO_BRAJA 0.377800 0.000000 0.295330 0.285409 0.490374 0.478838 0.440661 ENO_RHIEC 0.310072 0.295330 0.000000 0.098582 0.469455 0.453309 0.435659 ENO_AGRRK 0.333097 0.285409 0.098582 0.000000 0.483030 0.456606 0.433120 ENO_NEIMA 0.423113 0.490374 0.469455 0.483030 0.000000 0.365469 0.318335 ENO_BURCA 0.460577 0.478838 0.453309 0.456606 0.365469 0.000000 0.097015 ENO_RALPJ 0.444920 0.440661 0.435659 0.433120 0.318335 0.097015 0.000000� Теперь попытаемся определить насколько расстояния отклоняются от ультраметричности:
в качестве примера рассмотрим расстояния: d(RHIEC,AGRRK), d(RHOS4,RHIEC), d(RHOS4,AGRRK). Отметим, что матрица ультраметрична, если d(RHOS4,RHIEC)=d(RHOS4,AGRRK). Итак, из матрицы мы видим, что d(RHIEC,AGRRK) < d(RHOS4,AGRRK) (0.098582 < 0.333097) и d(RHIEC,AGRRK) < d(RHOS4,RHIEC) (0.098582 < 0.310072). Однако d(RHOS4,AGRRK)=0.333097, а d(RHOS4,RHIEC)=0.310072. Различие во втором знаке, азначит, матрица не так уж ультраметрична.
Рассмотрим и другой пример: d(BURCA,RALPJ), d(BRAJA,RALPJ), d(BRAJA,BURCA). Условие ультраметричности: d(BRAJA,RALPJ) = d(BRAJA,BURCA). Итак, из матрицы видно, что d(BURCA,RALPJ) < d(BRAJA,RALPJ) (0.097015 < 0.444920) и d(BURCA,RALPJ) < d(BRAJA,BURCA) (0.097015 < 0.478838). Однако d(BRAJA,BURCA)=0.478838), а d(BRAJA,RALPJ)=0.444920. Снова различие во втором знаке. Гипотеза подтвердилась.
Теперь попробуем определить насколько расстояния отклоняются от аддитивности:
� В качестве примера возмем четыре последовательности: BURCA, RALPJ, AGRRK, RHIEC.
Для начала сформулируем свойство аддитивности:
d(BURCA,AGRRK) + d(RALPJ,RHIEC) = d(BURCA,RHIEC) + d(RALPJ,AGRRK);
d(BURCA,RALPJ) + d(AGRRK,RHIEC) < d(BURCA,AGRRK) + d(RALPJ,RHIEC),
d(BURCA,RALPJ) + d(AGRRK,RHIEC) < d(BURCA,RHIEC) + d(RALPJ,AGRRK).
Итак:?
0.456606 + 0.435659 = 0.453309 + 0.433120 <=> 0.892265 &ne 0.886429;
0.097015 + 0.098582 < 0.456606 + 0.435659 <=> 0.195597 < 0.892265;
0.097015 + 0.098582 < 0.453309 + 0.433120 <=> 0.195597 < 886429;
� Как мы видим, последние два условия выполняются, однако первое нет. Тем не менее, разница незначительная (0.005836), поэтому можно сделать вывод, что данная матрица аддитивна. - Реконструкция дерева программой fneighbor по белку ENO
� Получим две реконструкции дерева программой fneighbor, используя два алгоритма: UPGMA и Neighbor-Joining. На вход даем файл с
матрицей расстояний, а на выходе получим файлы:
enon.fneighbor и enou.fneighbor.
� Заметим:
алгоритм UPGMA выдаёт укоренённое дерево с длинами ветвей;
алгоритм Neighbor-Joining выдаёт неукоренённое дерево с длинами ветвей.
Реконструкция дерева алгоритмом UPGMA:+----------ENO_RHOS4 +--4 ! ! +--------ENO_BRAJA ! +-3 ! ! +--ENO_RHIEC --6 +-----2 ! +--ENO_AGRRK ! ! +----------ENO_NEIMA +--5 ! +--ENO_BURCA +-------1 +--ENO_RALPJЭто дерево абсолютно такое же, как и два предыдущих.
Реконструкция дерева алгоритмом Neighbor-Joining:+--------ENO_BRAJA +--4 ! ! +--ENO_RHIEC ! +---3 ! +---ENO_AGRRK ! ! +----------ENO_NEIMA 5-----2 ! ! +---ENO_BURCA ! +------1 ! +-ENO_RALPJ ! +---------ENO_RHOS4Это дерево похоже на предыдущие, если укоренить его в позиции между "5" и "2".
© Азнаурян 2008 marina-91@list.ru