Филогенетические деревья
Отобранные бактерии
| Название | Мнемоника | Таксоны, к которым относятся отобранные бактерии |
| Bacillus anthracis | BACAN | Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus cereus group |
| Bacillus subtilis | BACSU | Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Bacillus; Bacillus subtilis group |
| Clostridium botulinum | CLOB1 | Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Clostridium tetani | CLOTE | Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiaceae; Clostridium |
| Enterococcus faecalis | ENTFA | Bacteria; Firmicutes; Bacilli; Lactobacillales; Enterococcaceae; Enterococcus |
| Finegoldia magna | FINM2 | Bacteria; Firmicutes; Clostridia; Clostridiales; Clostridiales incertae sedis; Clostridiales Family XI. Incertae Sedis; Finegoldia |
| Geobacillus kaustophilus | GEOKA | Bacteria; Firmicutes; Bacilli; Bacillales; Bacillaceae; Geobacillus |
| Lactobacillus acidophilus | LACAC | Bacteria; Firmicutes; Bacilli; Lactobacillales; Lactobacillaceae; Lactobacillus |
Скобочная формула дерева
(((LACAC,ENTFA),((BACAN,BACSU),GEOKA)),(FINM2,(CLOTE,CLOB1)))Изображение дерева
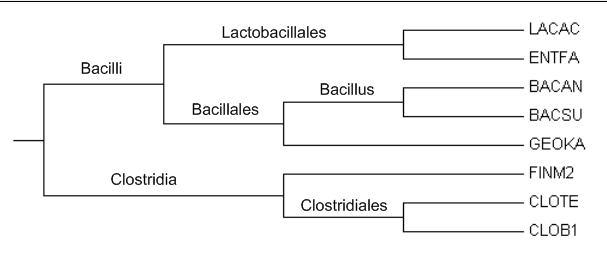
Ветви дерева
Дерево содержит следующие нетривиальные ветви:1) {LACAC,ENTFA} против {FINM2,CLOTE,CLOB1,GEOKA,BACAN,BACSU}
2) {LACAC,ENTFA,BACAN,BACSU,GEOKA} против {FINM2,CLOTE,CLOB1}
3) {CLOTE,CLOB1} против {FINM2,LACAC,ENTFA,GEOKA,BACAN,BACSU}
4) {BACAN,BACSU} против {FINM2,LACAC,ENTFA,GEOKA,CLOTE,CLOB1}
5) {BACAN,BACSU,GEOKA} против {LACAC,ENTFA,FINM2,CLOTE,CLOB1}
Выделяющиеся ветви
Bacillales - BACAN, BACSU, GEOKA
Clostridia - CLOB1, CLOTE, FINM2
Lactobacillales - ENTFA, LACAC
Bacillus - BACAN, BACSU
Clostridiaceae - CLOB1, CLOTE
Bacilli - ENTFA, LACAC, BACAN, BACSU, GEOKA
Реконструкция филогенетического дерева
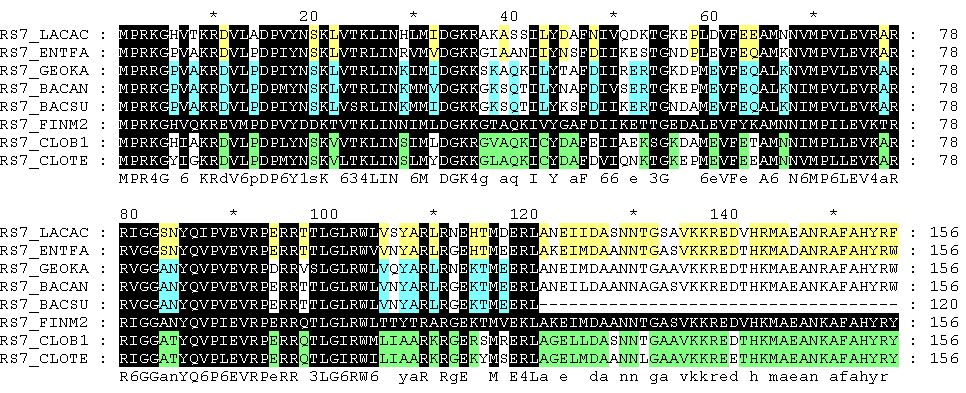
Для реконструкции филогенетического дерева отобранных бактерий был выбран рибосомный белок S7 (мнемоника RS7).Последовательности белка RS12 всех выбранных бактерий были получены из Swiss-Prot.
Последовательности были выравнены при помощи muscle.
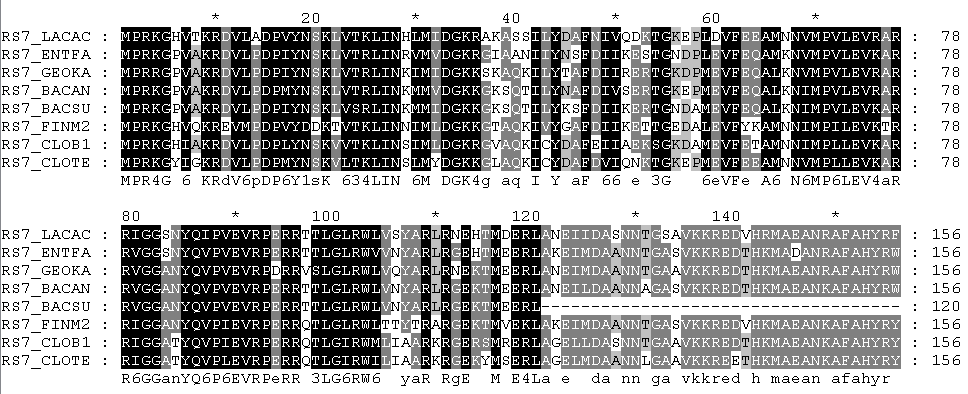
Были "подкрашены" позиции, консервативные внутри таксона:
Реконструкция дерева программой fprotpars
Программа нашла одно наиболее "бережливое" дерево.Для запуска программы fprotpars была использована следующая команда:
fprotpars -sequence rs7_aligned.fasta -outfile rs7.fprotpars
Скобочная формула: ((((CLOTE,CLOB1),FINM2),(GEOKA,(BACAN,(BACSU,ENTFA)))),LACAC)
изображение:
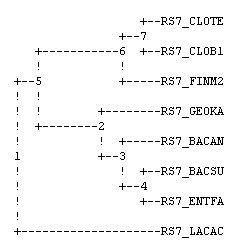
Это неукорененное дерево. Топология данного дерева немного отличается от правильного. Кластер ((CLOTE,CLOB1),FINM2) остаётся неизменным, однако ENTFA, принадлежащая с LACAC к Lactobacillales, в данном дереве становится близкой к BACSU (Bacillus subtilis group). Из-за чего первоначальный кластер ((LACAC,ENTFA),((BACAN,BACSU),GEOKA)) изменяется. Всего 2 нетривиальные ветви совпадают, остальные же противоречат правильным.
Оценка эволюционных расстояний между последовательностями программой fprotdist
Для запуска программы fprotpars была использована следующая команда:fprotdist -sequence rs7_aligned.fasta -outfile rs7.fprotdist
Выдача программы: rs7.fprotdist
Матрица расстояний
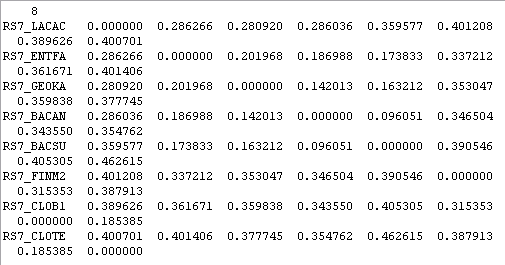
Утверждение, равносильное аксиоме ультраметричности: "из трех расстояний между тремя объектами два всегда равны между собой и не меньше третьего".
Оценим, насколько расстояния отклоняются от ультраметричности:
Рассмотрим LACAC, BACSU и FINM2 (все из далеких групп):
d(LACAC,BACSU) = 0.359577
d(LACAC,FINM2) = 0.401208
d(BACSU,FINM2) = 0.390546
Наиболее близкие расстояния (0.401208 и 0.390546) больше третьего (0.359577). Эта тройка удовлетворяет аксиоме ультраметричности.
Рассмотрим GEOKA, BACAN и CLOB1 (все из далеких групп):
d(GEOKA,BACAN) = 0.142013
d(GEOKA,CLOB1) = 0.359838
d(BACAN,CLOB1) = 0.343550
Наиболее близкие расстояния (0.359838 и 0.343550) также больше третьего (0.142013). Эта тройка тоже будет удовлетворять аксиоме ультраметричности.
Аддитивность: если есть 4 последовательности: A, B, C, D, - то из трех сумм d(A,B) + d(C,D); d(A,C) + d(B,D); d(A,D) + d(B,C) две равны между собой и больше третьей.
Оценим, насколько расстояния отклоняются от аддитивности:
Рассмотрим LACAC, BACSU, BACAN, CLOB1:
d(LACAC,BACSU) + d(BACAN,CLOB1) = 0.359577+0.343550=0.703127
d(LACAC,BACAN) + d(BACSU,CLOB1) = 0.286036+0.405305=0.691341
d(LACAC,CLOB1) + d(BACSU,BACAN) = 0.389626+0.096051=0.485677
Эти последовательности удовлетворяют аддитивности.
Реконструкции дерева программой fneighbor, с использованием алгоритмов UPGMA и Neighbor-Joining
С помощью программы fneighbor по матрице расстояний были постороены два дерева, с использованием алгоритмов UPGMA и Neighbor-Joining.Команды для запуска:
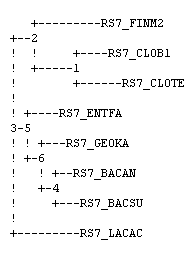
Neighbor-Joining:
fneighbor -datafile rs7.fprotdist -outfile rs7.Neighbor-Joining.fneighbor -outtreefile rs7.Neighbor-Joining.fneighbor.tree
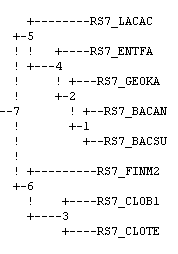
UPGMA:
fneighbor -datafile rs7.fprotdist -outfile rs7.UPGMA.fneighbor -outtreefile rs7.UPGMA.fneighbor.tree -treetype u
Результаты: UPGMA tree, UPGMA outfile, Neighbor-Joining tree, Neighbor-Joining outfile.
Neighbor-Joining:
UPGMA:
TreeTop
Для создания запроса выравнивание было переведено в другой формат.Результат работы программы (в графическом виде):
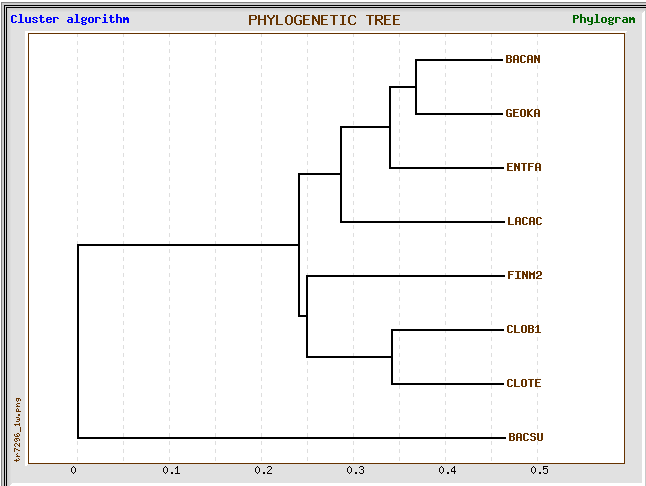
Топология этого дерева не совпадает с топологией дерева, выданного fprotpars.
Матрица расстояний:
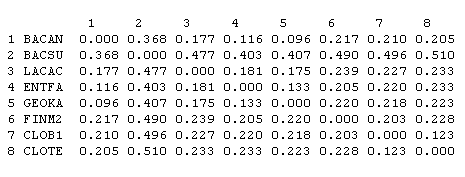
По-моему, данный формат матрицы расстояний более удобен, чем формат выдачи fprotdist.
Приводится не так много значащих цифр и матрица изначально является "таблицей" (строки и столбцы пронумерованы).
Во всех реконструкциях встретились ветви
{LACAC,ENTFA,BACAN,BACSU,GEOKA} против {FINM2,CLOTE,CLOB1}
{CLOTE,CLOB1} против {FINM2,LACAC,ENTFA,GEOKA,BACAN,BACSU}
© Dzama Margarita, 2011