| Онтология GO (название словаря) | Количество разных ассоциированных терминов GO | Функция белка (краткое описание, близкое к тексту определения термина(ов) GO |
|
|---|---|---|---|
| Где? | К сожалению, ничего нет | ||
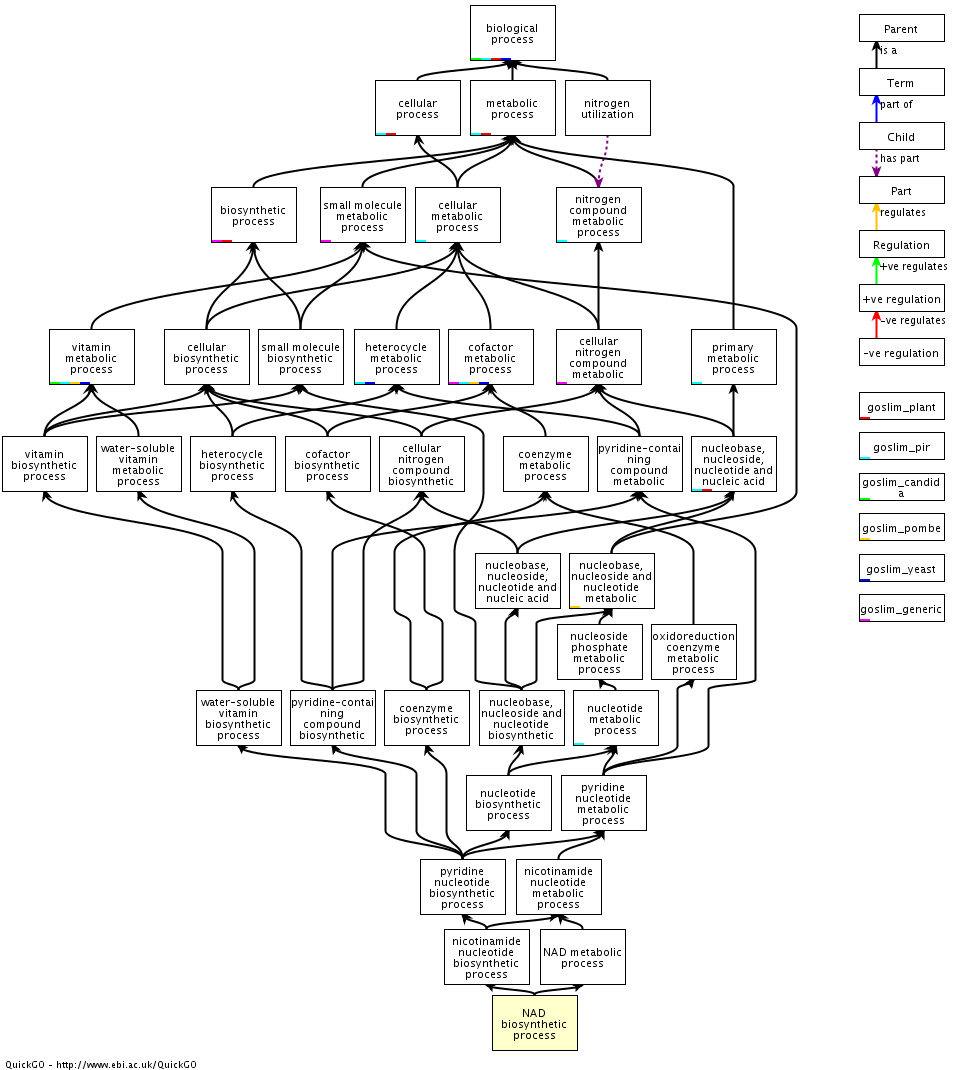
| Зачем, для чего? | 3 | Biological process (биологический процесс) | Ответная реакция на стресс (response to stress)
NAD биосинтетический процесс Споруляция, приводящая к формированию клеточной споры |
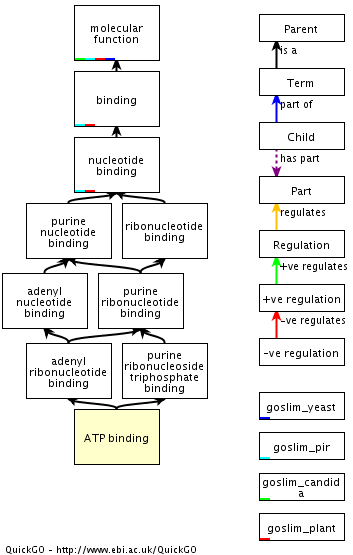
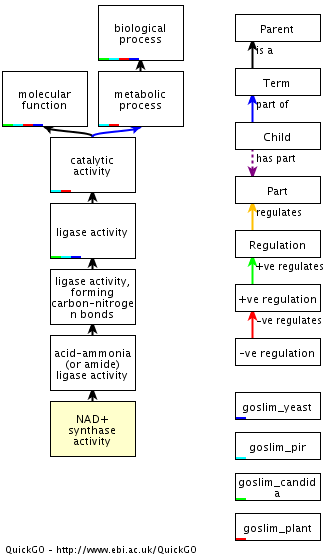
| Молекулярный механизм? | 7 | Molecular function (молекулярная функция) | Связывание с нуклеотидами
НАД+синтазная (глутамин-гидрализующая) активность Связывание с АТФ Лигазная активность |
| Специфичность? | К сожалению, ничего нет |
| GO ID выбранного термина | Список синонимов | Список ближайших родительских терминов GO с указанием типа связи | Список ближайших дочерних терминов GO с указанием типа связи |
|---|---|---|---|
| GO:0009435 | reduced nicotinamide adenine dinucleotide biosynthetic process
reduced nicotinamide adenine dinucleotide biosynthesis reduced NAD biosynthetic process reduced NAD biosynthesis oxidized nicotinamide adenine dinucleotide biosynthetic process oxidized nicotinamide adenine dinucleotide biosynthesis nicotinamide adenine dinucleotide biosynthesis NADH biosynthetic process NADH biosynthesis NAD synthesis NAD formation NAD biosynthesis NAD anabolism NAD (reduced) biosynthetic process NAD (reduced) biosynthesis NAD (oxidized) biosynthetic process nicotinamide adenine dinucleotide biosynthetic process oxidized NAD biosynthesis oxidized NAD biosynthetic process NAD (oxidized) biosynthesis |
is_a (inferred): biological_process (GO:0008150) cellular nitrogen compound metabolic process (GO:0034641) primary metabolic process (GO:0044238) cellular process (GO:0009987) metabolic process (GO:0008152) nucleobase, nucleoside, nucleotide and nucleic acid metabolic process (GO:0006139) biosynthetic process (GO:0009058) cellular metabolic process (GO:0044237) nitrogen compound metabolic process (GO:0006807) nucleobase, nucleoside and nucleotide metabolic process (GO:0055086) small molecule metabolic process (GO:0044281) cellular biosynthetic process (GO:0044249) cofactor metabolic process (GO:0051186) heterocycle metabolic process (GO:0046483) nucleobase, nucleoside, nucleotide and nucleic acid biosynthetic process (GO:0034654) nucleoside phosphate metabolic process (GO:0006753) small molecule biosynthetic process (GO:0044283) vitamin metabolic process (GO:0006766) cellular nitrogen compound biosynthetic process (GO:0044271) coenzyme metabolic process (GO:0006732) cofactor biosynthetic process (GO:0051188) heterocycle biosynthetic process (GO:0018130) nucleobase, nucleoside and nucleotide biosynthetic process (GO:0034404) nucleotide metabolic process (GO:0009117) oxidoreduction coenzyme metabolic process (GO:0006733) pyridine-containing compound metabolic process (GO:0072524) vitamin biosynthetic process (GO:0009110) water-soluble vitamin metabolic process (GO:0006767) coenzyme biosynthetic process (GO:0009108) nucleotide biosynthetic process (GO:0009165) pyridine nucleotide metabolic process (GO:0019362) pyridine-containing compound biosynthetic process (GO:0072525) water-soluble vitamin biosynthetic process (GO:0042364) nicotinamide nucleotide metabolic process (GO:0046496) pyridine nucleotide biosynthetic process (GO:0019363) is_a: NAD metabolic process (GO:0019674) nicotinamide nucleotide biosynthetic process (GO:0019359) |
is_a: NAD biosynthetic process NAD biosynthetic process NAD biosynthetic process |
| GO:0005524 | Нет | is_a (inferred): molecular_function (GO:0003674) binding (GO:0005488) nucleotide binding (GO:0000166) purine nucleotide binding (GO:0017076) ribonucleotide binding (GO:0032553) adenyl nucleotide binding (GO:0030554) purine ribonucleotide binding (GO:0032555) is_a: adenyl ribonucleotide binding (GO:0032559) purine ribonucleoside triphosphate binding (GO:0035639) |
Нет |
| GO:0008795 | deamido-NAD+:ammonia ligase (AMP-forming) diphosphopyridine nucleotide synthetase activity NAD synthase (AMP-forming) NAD synthase activity NAD synthetase activity NAD(+) synthetase activity NAD+ synthetase activity nicotinamide adenine dinucleotide synthetase activity |
is_a (inferred) molecular_function (GO:0003674) catalytic activity (GO:0003824) ligase activity (GO:0016874) ligase activity, forming carbon-nitrogen bonds (GO:0016879) is_a: acid-ammonia (or amide) ligase activity (GO:0016880) |
Нет |
Латинское название - Oryza L. Английское название - Rice Русское название - Рис
На сайте NCBI в БД Taxonomy определили для
Oryza L.
ранг таксона: род идентификатор (NCBI_TaxID): 4527.
Изучим в SRS поле ProteinExistence БД UniProt:
1: evidence at protein level (Экспериментальные данные о существовании белка) 2: evidence at transcript level (Экспериментальные данные о существовании транскрипта, существование белка строго не доказано) 3: inferred from homology (Гипотетический белок, предсказан по гомологии) 4: predicted (Иные предсказанные гипотетические белки) 5: uncertain (Сомнительное существование белка)
| Количество в UniProt | UniProt, % | |
|---|---|---|
| Существование белка доказано экспериментально | 385 | 0,26 |
| Известны только соответствующие транскрипты | 16065 | 10,97 |
| Гипотетический белок, предсказан по гомологии | 9596 | 6,56 |
| Иные предсказанные гипотетические белки | 120394 | 82,21 |
| Количество в SwissProt | SwissProt, % | |
|---|---|---|
| Существование белка доказано экспериментально | 275 | 9,2 |
| Известны только соответствующие транскрипты | 2001 | 66,4 |
| Гипотетический белок, предсказан по гомологии | 724 | 24,0 |
| Иные предсказанные гипотетические белки | 13 | 0,4 |
Коды экспериментального доказательства функции: # EXP: Inferred from Experiment # IDA: Inferred from Direct Assay # IPI: Inferred from Physical Interaction # IMP: Inferred from Mutant Phenotype # IGI: Inferred from Genetic Interaction # IEP: Inferred from Expression Pattern Запрос: Taxonomy: Oryza ProteinExistence 1: evidence at protein level DBxref_ (*GO*)&(*P:*)&(*F:*)&(*C:*)&(EXP:|IDA:|IPI:|IMP:|IGI:|IEP:) Если в запрос включить транскрипты, результат остается прежним. ((([uniprot-Taxonomy:Oryza*] & ((((([uniprot-ProteinExistence:1:*] & [uniprot-ProteinExistence:evidence*]) & [uniprot-ProteinExistence:at*]) & [uniprot-ProteinExistence:protein*]) & [uniprot-ProteinExistence:level*]) | [uniprot-ProteinExistence:1: evidence at protein level*])) & (((([uniprot-DBxref_:*GO*] & [uniprot-DBxref_:*P:*]) & [uniprot-DBxref_:*F:*]) & [uniprot-DBxref_:*C:*]) & ((((([uniprot-DBxref_:EXP:*] | [uniprot-DBxref_:IDA:*]) | [uniprot-DBxref_:IPI:*]) | [uniprot-DBxref_:IMP:*]) | [uniprot-DBxref_:IGI:*]) | [uniprot-DBxref_:IEP:*])))
В результате получено 30 записей
Такое небольшое количество аннотированых по трем онтологиям белков с подтвержденной экспериментально функцией(< 8% ), вероятно, связано с недостаточной изученностью белков и временными затратами на экспериментальные подтверждения.
GO: 0019856(pyrimidine base biosynthetic process) Ontology: Biological processОсуществили поиск в SRS:
запрос: ([uniprot-DBxref_:GO:0019856*] & [uniprot-Taxonomy:Oryza*])В результате найдено 3 белка. Белки проаннотированы по словарям: Function, Biological Process.
GO:0004151; F:dihydroorotase activity; IEA*:InterPro. GO:0046872; F:metal ion binding; IEA:UniProtKB-KW. GO:0019856; P:pyrimidine base biosynthetic process; IEA:InterPro.Найденные белки относятся к семейству дегидротаз, для Q0JJD1_ORYSJ известен транскрипт, A2WV09_ORYSI и Q94J38_ORYSJ - гипотетические белки по гомологии.