Профиль гидрофобности был построен с помощью программы pepwindow,
эта программа была запущена со значением data параметра graph. Размер окна составлял
19 аминокислотных остатков (-length 19).
Данные, полученные с использованием этой программы, были обработаны в
Excel; был построен график профиля гидрофобности. Этот график находится в файле profile.xls.
Используя данный график, было произведено предсказание трансмембранных сегментов; это
предсказание было сопоставлено с предсказанием программы TMHMM, а также с данными ресурса OPM о топологии
прототипа заданного белка — AQP4_RAT (оказалось, что прототип имеет то же количество
аминокислотных остатков, что и белок AQP4_BOVIN, и выравнивание этих белков не содержит гэпы,
поэтому аминокислотные координаты по обоим белкам совпадает). Последовательности этих белков были
найдены в базе данных Swiss-Prot по ID. Вес выравнивания, полученного с помощью программы
needle при параметрах по умолчалию, — 1589, процент идентичности последовательностей — 94.1.
Координаты трансмембранных участков в аминокислотной последовательности по данным трёх источников
также приведены в файле profile.xls.
При построении топологии вручную изначально длина спирали принималась равной
20 аминокислотным остаткам, её центр соответствовал пику на профиле гидрофобности, но и ширина
участка с довольно высоким средним значением гидрофобности должна была приблизительно равняться 20 остаткам.
Определённые таким образом координаты немного редактировались на концах (выбрасывались заряженные
остатки на концах спиралей или к спиралям добавлялись близлежащие гидрофобные остатки — что встречалось чаще).
Помимо этого, в соответствии с правилом фон Хейне, гласящем, что немембранные участки белка,
лежащие в цитоплазме, содержат больше положительно заряженных остатков (K и R), чем те, что лежат на внешней стороне мембраны,
были предсказаны внешние и внутренние петли. Это же предсказание было сделано с помощью программы TMHMM, а
также после анализа координат трансмембранных сегментов, содержащихся в базе данных OPM. Результаты работы
представлены в виде трёх новых последовательностей, присоединённых к выравниванию белков AQP4_BOVIN и AQP4_RAT;
их названия — Manual, TMHMM и OPM (в порядке перечисления). Трансмембранные спирали обозначены буквами H,
участки белка, находящиеся на внутренней стороне мембраны — знаком +, а участки с внешней стороны —
знаком –.
Выравнивания находятся в файле marking.msf.
Копия страницы, выданной прграммой TMHMM находится здесь. Помимо
приведённых координат предполагаемых трансмембранных, внутренних и наружных участков белка, программа
выдаёт график, на котором показана зависимость вероятности вхождения аминокислоты в один из
трёх видов участков белка от её номера в последовательности.
| |
По профилю гидрофобности |
TMHMM |
| Всего предсказано остатков в спиралях(N) |
123 |
138 |
| Из них |
|
|
|
| |
А. По данным ОРМ находятся внутри мембраны |
109 |
119 |
| |
В. Из них — "торчащие" из мембраны остатки на концах
спиралей (по данным PDB-структуры) |
1 |
4 |
| |
С. Вообще не имеют никакого отношения к трансмембранным
спиралям |
14 |
19 |
| D. Число непредсказанных остатков, которые по данным ОРМ
находятся в мембране. |
33 |
28 |
| Точность предсказания, A/N |
0,886 |
0,862 |
| Сверхпредсказание, C/N |
0,114 |
0,138 |
| Недопредсказание, D/(L-N), где L-длина
последовательности (323 а.о.) |
0,165 |
0,151 |
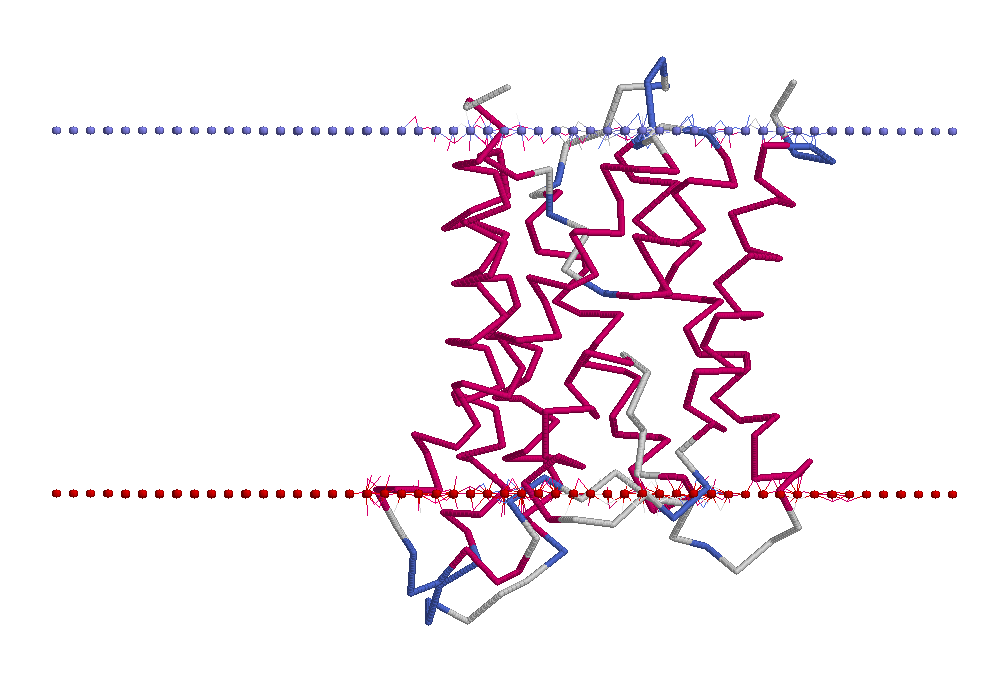
При определении остатков, "торчащих" из мембраны на концах спиралей был использован PDB-файл
структуры белка-прототипа — 2d57.pdb. Используя RasMol, была выделена одна из цепей
тетрамера, содержащегося в файле, и раскрашена по структуре. Кроме того, в PDB-структуре содержатся
dummy-атомы, изображающие границу мембраны (наружная — красная, внутренняя — синяя).
Номера, остатков, покрашенных в красный цвет (входящих в спирали) и торчащих из мембраны:
33, 57, 58, 59, 68, 69, 206, 224. Хотя по данным, указанным на сайте OPM, эти остатки не
входят в спирали, в пункте B рассматривались именно они.
Ниже представлен текст скрипта который был
написан для определения номеров остатков, а справа помещено изображение им порождаемое.
load 2d57.pdb
background white
wireframe off
backbone 70
color structure
restrict *A or hetero
select hetero
cpk 100
color cpk
|
|
|
Согласно таблице, оба метода (определение трансмембранных участков
вручную и с помощью программы TMHMM) дают погрешность в 10 – 15%. Отчасти это связано с тем,
что идентифицировать 2 α-спирали, состоящие из 10 остатков, не удалось при применении обоих
методов. Эти спирали в прототипе, скорее всего, пронизывают лишь один слой мембраны, чем и
объясняется их небольшая длина. Вполне логично, что программа pepwindow, строившая профиль
при длине рамки 19, не выдала широкого пика на этом участке. Что касается программы TMHMM, то все
спирали, предсказанные ей, имели длину в 23 аминокислотных остатка (при параметрах по умолчанию),
что указывает на её неприменимость к таким α-спиралям небольшого размера.
|
Из структуры белка, содержащейся в PDB-файле, видно, что продолжения спиралей,
состоящих из 10 аминокислотных остатков и пронизывающих один липидный слой мембраны, выходят
в канал, образованный мономером белка. Изображение, полученное из PDB-файла находится справа.
|

|

