- Краткое описание структуры в файле 1EXD.pdb
В данном файле приведены координаты атомов двух молекул:
молекулы аптамера глютаминовой тРНК и
молекулы белка-синтетазы глютаминовой тРНК
из организма Escherichia coli.
Для исследования была выбрана цепь B, представляющая собой глютаминовую тРНК со следующей последовательностью:
[902] 5' - GGGGUAUCGCCAA GCGGUAAGGCACCGGAUUCUGAUU CCGGAGGUCGAGGUUCGAAUCCUC GUACCCCAGCCA - 3' [976],
где 902 и 976 - номера первого и последнего нуклеотида.
В последовательности на 3'-конце имеется триплет CCA, к которому собственно и присоединяется аминокислота. Кроме того, в pdb-файле приведены координаты его атомов. - Исследование вторичной структуры
С помощью программы find_pair пакета 3DNA были определены возможные
водородные связи между азотистыми основаниями
(файл rna.fp).
В соответствии с полученными данными:
акцепторный стебель состоит из участка 902-907 и комплементарного ему участка 966-971,
Т-стебель состоит из участков 949-953 и 961-965,
D-стебель - из участков 910-912 и 923-925
и антикодоновый стебель - из участков 937-944 и 926-933,
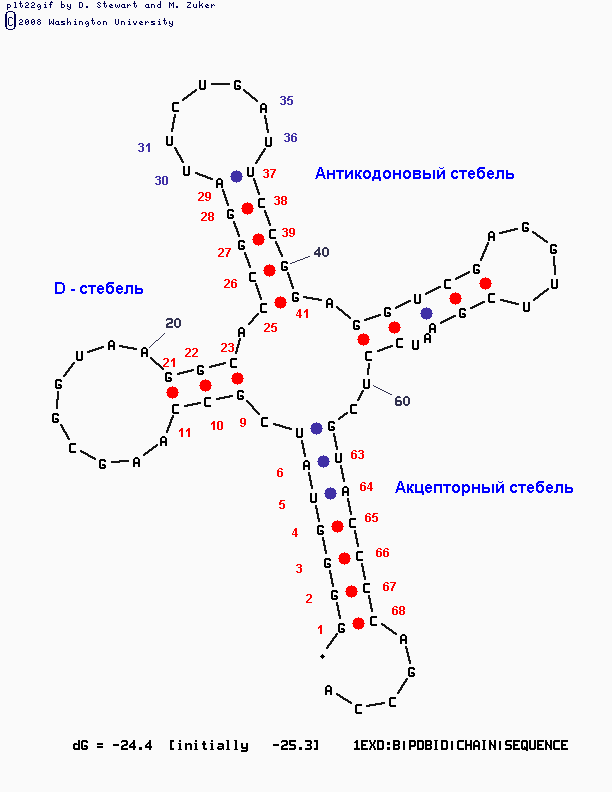
причем водородные связи на комплементарных участках образуются антипараллельно.Ниже на рисунке представлено изображение остова молекулы исследуемой тРНК, полученное в RasMol при помощи соответствующего скрипта.
В остове акцепторный стебель выделен красным, Т-стебель - зеленым, D-стебель - синим, антикодоновый - оранжевым цветами.Рис.1. Вторичная структура молекулы аптамера глютаминовой тРНК из организма Escherichia coli Скрипт для получения изображения 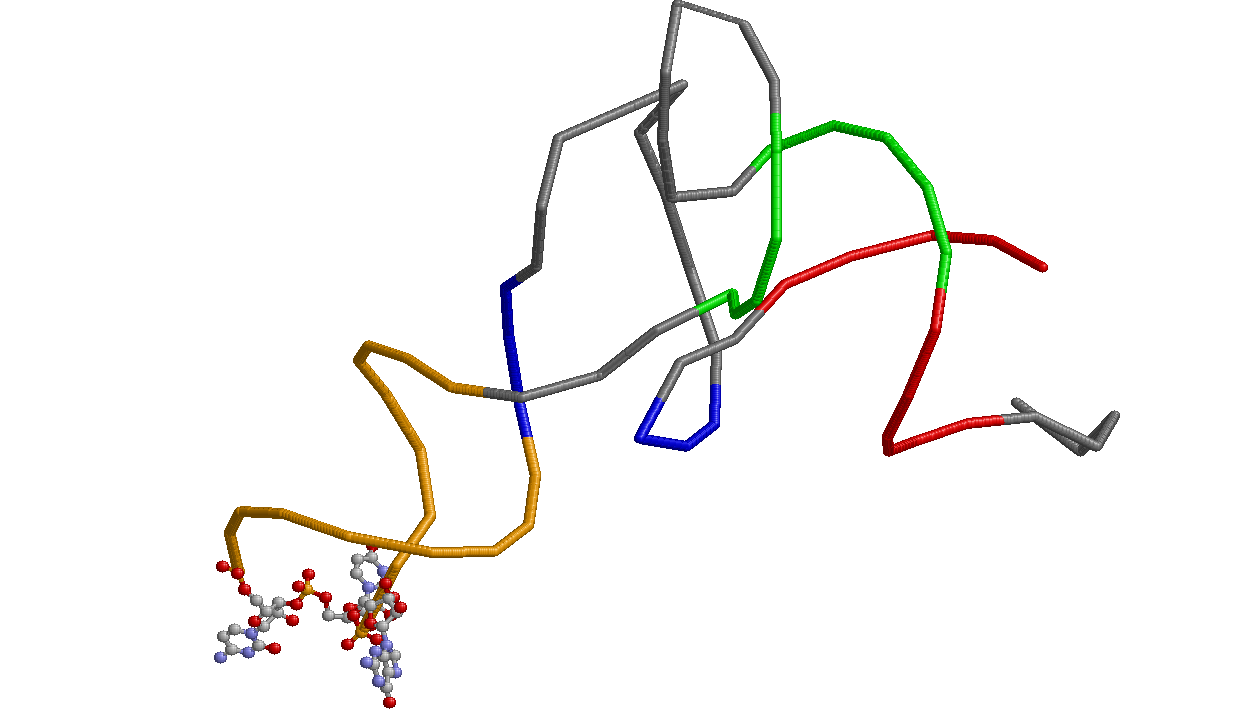
define helix1 902-907,966-971 define helix3 937-944, 926-933 define helix4 949-953, 961-965 define helix2 910-912, 923-925 background white select all wireframe off backbone 100 select helix1 color red select helix2 color blue select helix3 color orange select helix4 color green select not (helix1, helix2, helix3, helix4) color grey
На Рис.1 в антикодоновой петле в шарнирной модели показаны нуклеотиды, составляющие антикодон. Он образован основаниями с номерами 934-936 и представляет собой последовательность CUG. Последовательность ДНК, комплементарная такому антикодону, т.е. кодон, будет иметь вид CAG. И именно эта последовательность является одной из двух кодирующих последовательностей для аминокислоты глутамин.Структуру стеблевых дуплексов поддерживают 19 канонических и 9 неканонических пар оснований.
Например, есть неканоническая пара А-А, образованная нуклеотидами 944 и 926, и выглядит она следующим образом (изображение, полученное при помощи программы RasMol):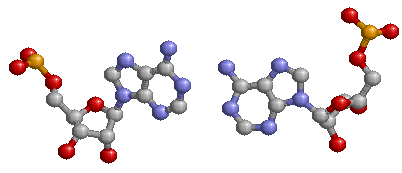
Также можно отметить отсутствие вариабельной петли в структуре тРНК и полное отсутствие модифицированных оснований, отсутствие остатка тимидина в Т-петле, отсутствие дигидроуридинов в D-петле - Исследование третичной структуры
1. В файле stacking.pdb программа analiyze выдала предполагаемые
стекинг-взаимодействия между парами оснований, связанных водородными связями.
Всего было найдено 27 возможных стекинг-взаимодействий для 28 пар оснований.
Из них я выбрала для изучения взаимодействие между парами
A_907-U_966 и C_949-G_965, идущее под номером 6.
Это стекинг-взаимодействие между основаниями акцепторного стебля и Т-стебля
изображено ниже на картинке, полученной при помощи команды stack2img.
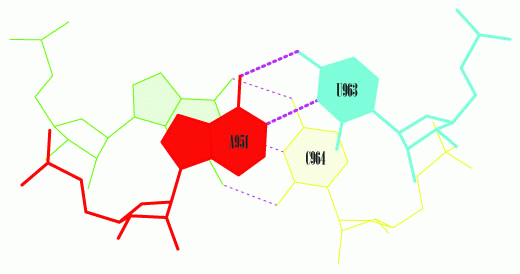
По данным, полученным программой analyze (файл rna.out), суммарная площадь перекрывания для рассматриваемых мной двух пар равна 9,51 квадратных ангстрем. В сравнении с аналогичной величиной для других стекинг-взаимодействий, это значение кажется мне довольно большим, а вероятность реального существования такого стекинг-взаимодействия - высокой.
2. В файле rna.fp есть информация о двух дополнительных водородных связях между основаниями D- и Т-петель.
Одна из них образуется между G 918 и U 955, пара оснований G и U является неканонической. 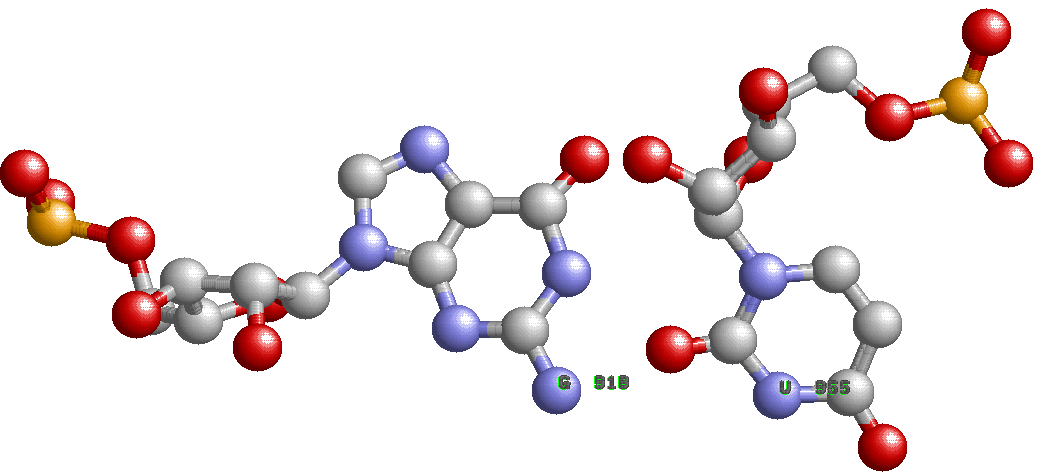
Вторая водородная связь образуется между G 919 и C 956, пара G и C является канонической. 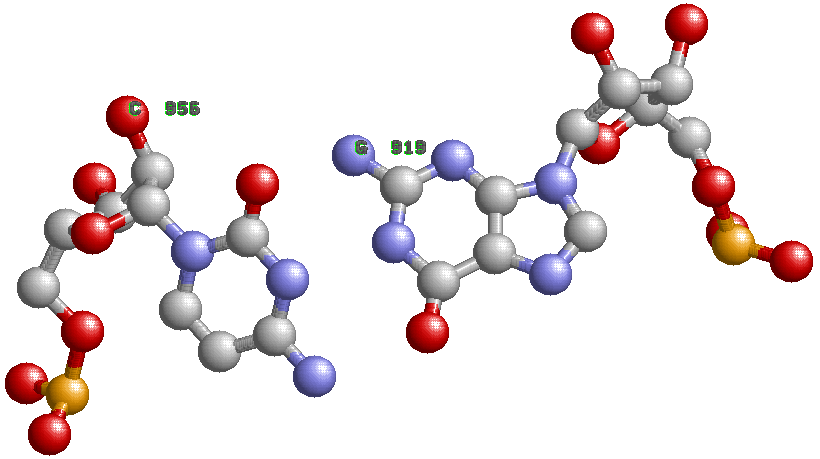
- Предсказание вторичной структуры тРНК
Реальная и предсказанная вторичная структура тРНК из файла 1EXD.pdb
Участок структуры
Позиции в структуре
(по результатам find_pair)Результаты предсказания
с помощью einvertedРезультаты предсказания
по алгоритму ЗукераАкцепторный стебель 5' 902-907 3'
5' 966-971 3'
Всего 6 парпредсказаны все 6 пар предсказаны все 6 пар и одна лишняя D-стебель 5' 910-912 3'
5' 923-925 3'
Всего 3 парыне предсказано ни одной пары предсказаны все 3 пары T-стебель 5' 949-953 3'
5' 961-965 3'
Всего 5 парне предсказано ни одной пары предсказаны все 5 пар (но в файле, отличающемся от взятого мной за оптимальный) Антикодоновый стебель 5' 937-944 3'
5' 926-933 3'
Всего 8 парне предсказано ни одной пары предсказаны 5 пар Общее число канонических пар нуклеотидов 19 6 19 в оптимальной структуре Предсказание с помощью einverted
Мы пытались предсказать вторичную структуру тРНК при помощи программы einverted - в итоге угадали всего 6 пар из акцепторного стебля
Требовалось подобрать параметры, при которых будет найдено как можно больше реальных пар, образующих водородные связи. В действительности, данная программа, насколько я поняла, предназначена для поиска палиндромов в ДНК. Она также может работать и с РНК, при этом только заменяет основания: урацил на тимин. Но ее алгоритм для нашей задачи кажется мне не слишком удобным. Подбирая различные параметры, я пыталась добиться хоть какого-то положительного результата. Но каждый раз оказывалось, что программа "складывает" последовательность РНК почти пополам при поиске канонических пар. Причем при таком "складывании" всегда получалось, что "перегиб" проходит по зоне антикодонового стебля, т.е. таким образом программа пытается искать водородные связи между основаниями, между которыми их заведомо быть не может. В общем, за разумное число попыток мне не удалось найти антикодоновый стебель при помощи данной программы.
Но зато практически всегда находился акцепторный стебель: его основания находятся на конце (или почти на конце) нуклеотидной последовательности РНК. Поэтому такой алгоритм вполне позволяет находить акцепторный стебель с достаточной точностью.
Что же касается D- и T- стеблей, их, как мне кажется, маловероятно найти такой программой, т.к для из поиска РНК потребовалось бы "складывать" по "оси", значительно удаленной от середины последовательности. А для программы, ищущей палиндромы, это, наверное, не очень приемлемо.Предсказание с помощью mfold
С программой mfold было интереснее работать, т.к, во-первых, более наглядно, а во-вторых, программа выдает больше интересных вариантов вторичной структуры тРНК, чем einverted.
Сначала программа была запущена со входным параметром Р, равным 0. Этот параметр показывает, на сколько процентов выдаваемое предсказание структуры может отличаться по своей вычисленной энергии от оптимального.
В первый раз mfold предложил 3 возможных структуры, которые хоть и далеки от реальной, но все же имеют с ней много общего. Эти же 3 структуры программа, естественно, предлагала и при всех остальных параметрах.
При Р=5 выдача mfold не изменилась, 3 соответствующих структуры описаны нижы:Наименее удачная структура, но одна из немногих, где верно предсказан T-стебель.
(на всех изображениях - выдачах программы mfold красными цифрами указаны номера нуклеодитов верно предсказанных пар)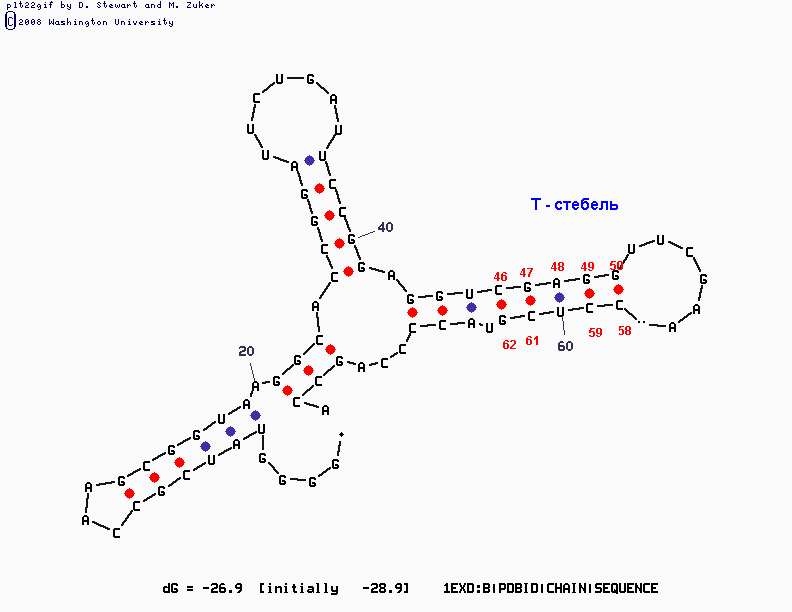
Также верно предсказан Т-стебель, но помимо него программа обнаружила акцепторный. 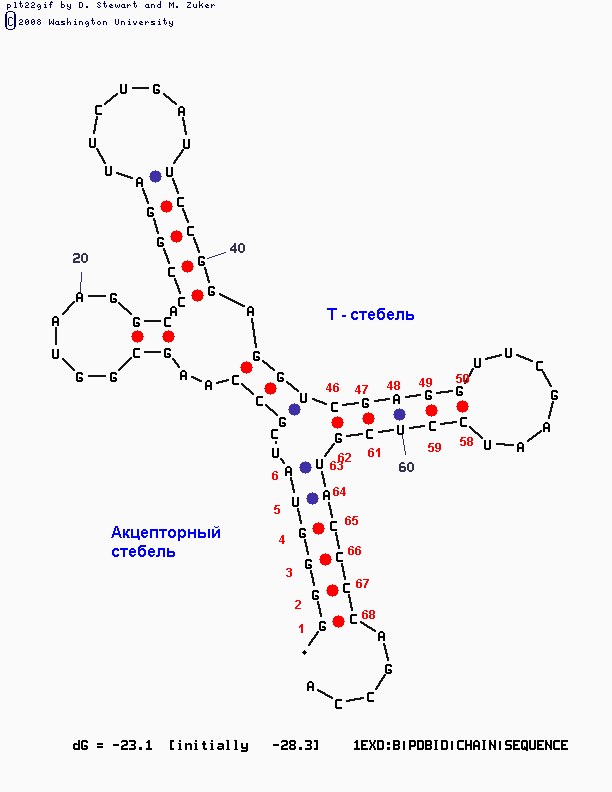
Программа нашла D- и акцепторный стебли.
Очевидно, с параметром Р=0 mfold выдал не особо удачные варианты вторичной структуры тРНК.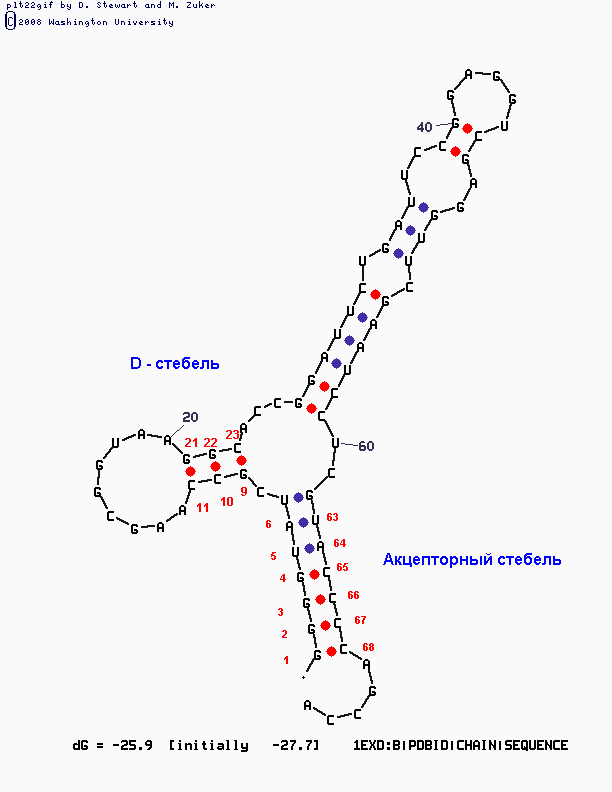
При Р=15 программа выдала уже 9 различных структур, из которых одна показалась мне наиболее удачной:
Полностью верно предсказан D-стебель, в акцепторный mfold "вписал" лишнюю пару, а в антикодоновом пропустил 3, две из которых, вообще-то говоря, являются неканоническими. Но в целом данная структура является, на мой взгляд, наиболее близкой к реальной, ее я и взяла за оптимальную, выданную mfold.