EC=1.1.1.44
1. Oxidoreductases (оксидоредуктазы)
1.1. Acting on the CH-OH group of donors... (Действие на СН-ОН группу доноров...)
1.1.1. ...with NAD+ or NADP+ as acceptor (С NAD+ или NADP+ в качестве акцептора)
1.1.1.4. phosphogluconate dehydrogenase (decarboxylating)(фосфоглутарат дегидрогеназа (декарбоксилирующая).
Уравнение катализируемой реакции:
6-phospho-D-gluconate + NADP+ = D-ribulose 5-phosphate + CO2 + NADPH
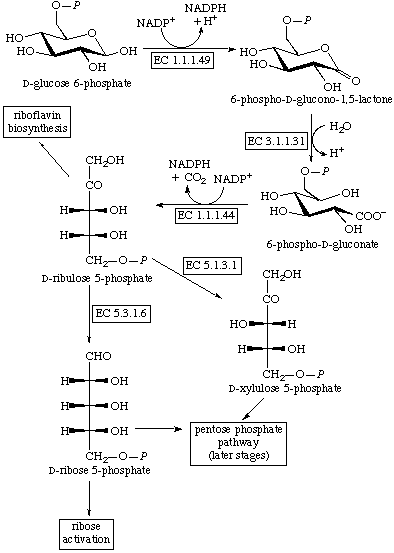
Название локуса гена: b2029
Метаболические пути:
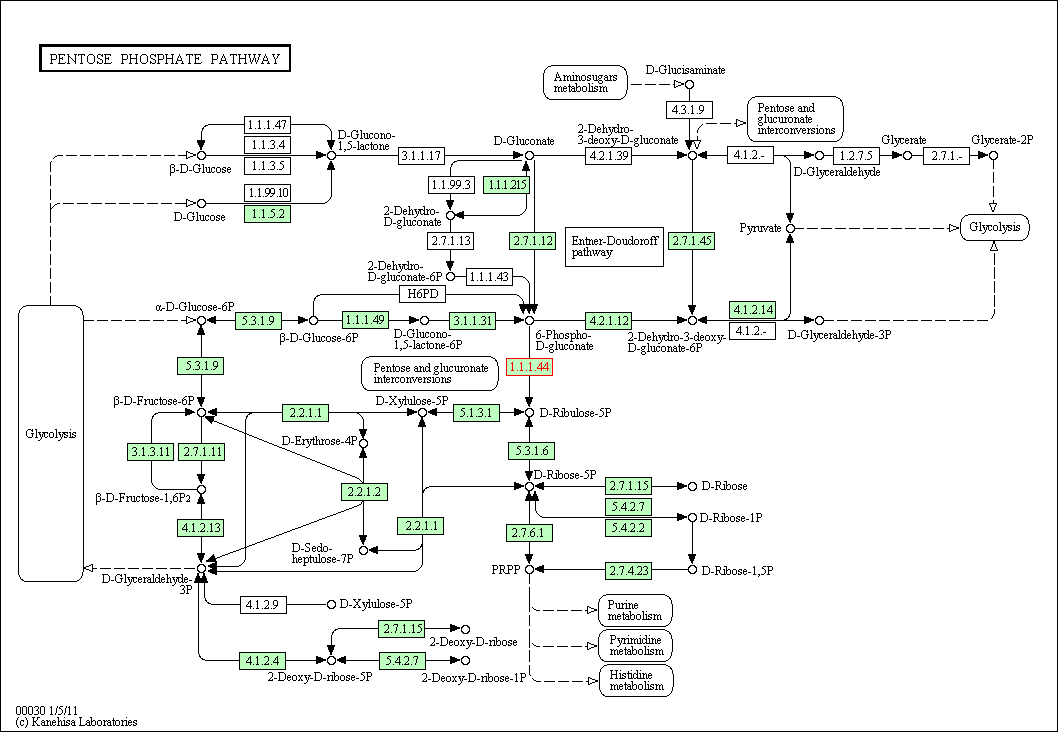
eco00030 - Pentose phosphate pathway (Пентозный фосфатный путь).
eco00480 - Glutathione metabolism (Метаболизм глутатиона).
eco01100 - Metabolic pathways (Метаболические пути).
eco01110 - Biosynthesis of secondary metabolites (Биосинтез вторичных метаболитов).
eco01120 - Microbial metabolism in diverse environments (Метаболизм микроорганизмов в различных условиях).
Пентозный фосфатный путь:
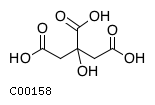
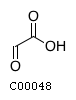
| Цитрат | Глиоксилат |
| Citrate; Citric acid; 2-Hydroxy-1,2,3-propanetricarboxylic acid; 2-Hydroxytricarballylic acid | Glyoxylate; Glyoxalate; Glyoxylic acid |
| C00158 | C00048 |
 |  |
Выбранная цепочка ферментативных реакций:
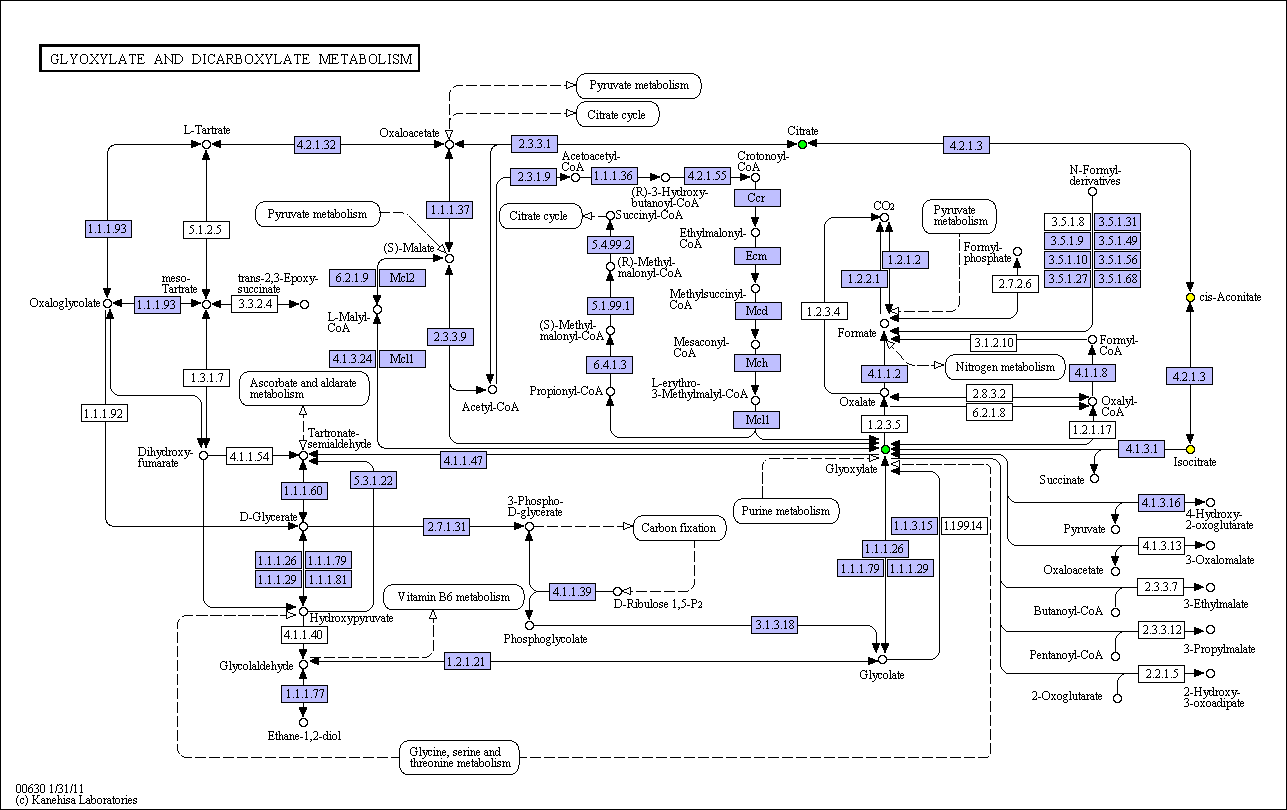
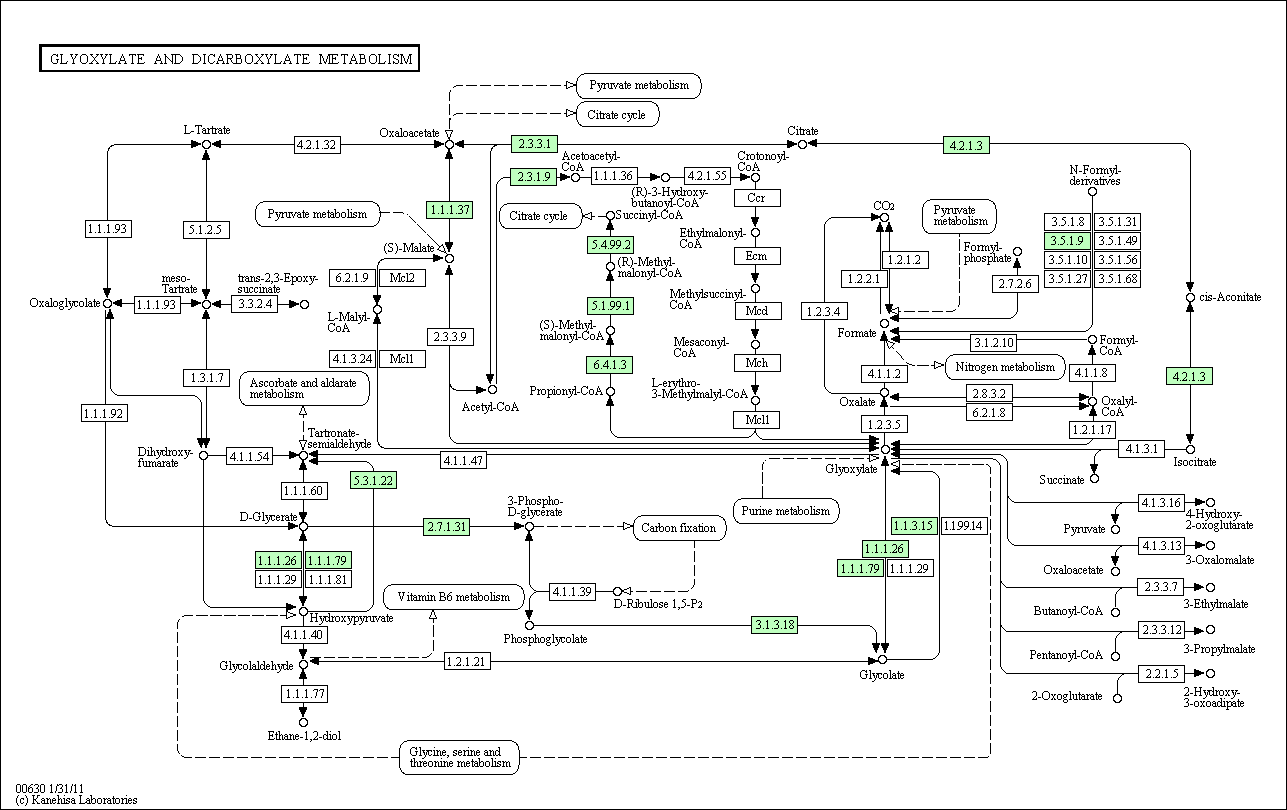
путь: ko00630 (Glyoxylate and dicarboxylate metabolism)- Метаболизм глиоксилата и дикарбоксилата,
цепочка: Citrate <--> Glyoxylate.
интермедиаты: cis-Aconitate (цис-аконитат) С00417, Isocitrate (изоцитрат) С00311.
| Организм | Возможна ли цепочка реакций (да/нет/неизвестно) |
Обоснование |
| Escherichia coli K-12 MG1655 | Да | Присутствуют все ферменты, необходимые для осуществления цепочки реакций |
| Archaeoglobus fulgidus | Да | Присутствуют все ферменты, необходимые для осуществления цепочки реакций |
| Arabidopsis thaliana (thale cress) | Да | Присутствуют все ферменты, необходимые для осуществления цепочки реакций |
| Homo sapiens | Да | Присутствуют все ферменты, необходимые для осуществления цепочки реакций |
Указанный путь для homo sapiens:
Фермент 1.11.1.6 в разных организмах:
Код фермента: 1.11.1.6.
Опция использования маски (Use wildcards) снимается, чтобы находились ферменты именно с заданным EC, не с похожими ЕС.
Запрос: ([swissprot-ECNumber:1.11.1.6] & ([swissprot-ID:*_human] | [swissprot-ID:*_ARCFU]))
В каждом организме найдено по одному белку: CATA_HUMAN (P04040) и KATG_ARCFU (O28050).
Доменная организация найденных белков (по PFAM):
CATA_HUMAN (P04040):

KATG_ARCFU (O28050):
В этих ферментах нет одинаковых доменов.
Доменная организация найденных белков (по PFAM):
Ортолог CATA_HUMAN в эукариотах:
Ген: hsa:847
Ортолог:pon:100171588
Организм: Pongo abelii (Sumatran orangutan)
Процент идентичности: 98,5%
Перекрытие последовательностей: 527
Ортолог KATG_ARCFU в эукариотах:
Ген: afu:AF2233
Ортолог:Ferp_2153
Организм: Ferroglobus placidus
Процент идентичности: 82,3%
Перекрытие последовательностей: 745
В одинаковых верментах далеких организмов (человека и археи Archaeoglobus fulgidus) не найдено одинаковых доменов. Однако судя по проценту идентичности этих ферментов и их ортологов из другого таксона, эти белки схожи.