Докинг низкомолекулярных лигандов в структуру белка
NAG
SMILES NAG
Команды:
obgen nag.smi > nag.mol
babel -imol nag.mol -opdb nag.pdb
С помощью скрипта созданы файлы pdbqt для белка
и
лиганда.
Создан файл vina.cfg.
Первый докинг.
Получены файлы:
nag_prot.pdbqt - положения лиганда.
и
nag_prot.log. - энергии моделей.
Три лучших энергии:
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -5.0 0.000 0.000
2 -4.8 3.523 5.281
3 -4.8 4.304 6.329
Все состояния лиганда:
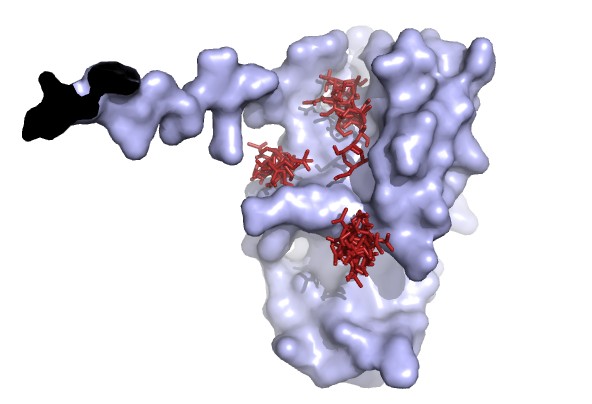
Докинг с учетом подвижности боковых радикалов.
Подвижная часть: Asp59, Asn113, Arg115.
Команды:
python /usr/share/pyshared/AutoDockTools/Utilities24/prepare_flexreceptor4.py -r seq5.pdbqt -s ASP59_ASN113_ARG115
vina --config vina.cfg --receptor 5_rigid.pdbqt --flex 5_flex.pdbqt --ligand nag.pdbqt --log nag_prot_flex.log --out nag_5_flex.pdbqt
Полученные файлы:
nag_prot_flex.log.
nag_prot_flex.pdbqt
Три лучших энергии:
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -5.0 0.000 0.000
2 -4.8 3.186 4.663
3 -4.7 1.532 3.869
Докинг с подвижными лигандами считался дольше, чем обычный, что логично.
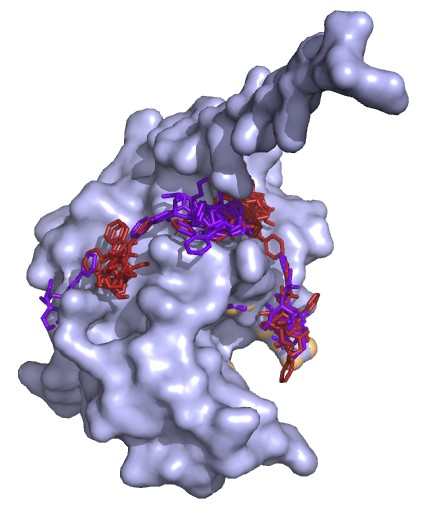
Полученные структуры:
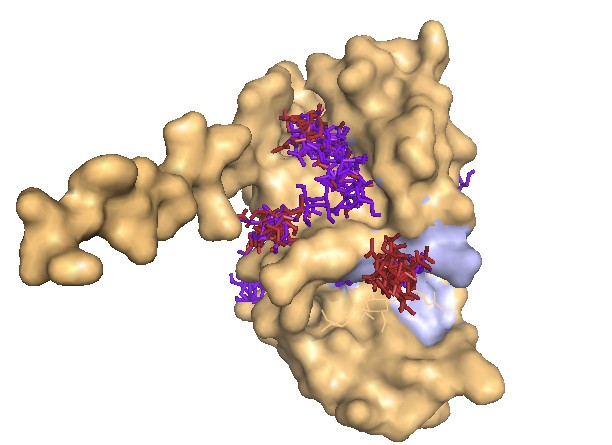
В обычном докинге (сиреневый белок, красный лиганд) центров расположения лиганда меньше, чем в докинге с подвижными
радикалами (оранжевый белок, фиолетовый лиганд).
В обычном докинге большая часть положений лиганда сосредоточена в активном центре, тогда как во втором докинге все положения лиганда
в основном разбросаны в других участках белка. Таким образом, подвижный докинг является более гибким, возможно, с его помощью можно
проследить путь лиганда к активному центру.
Докинг с другими лигандами.
Получены лиганды, в которых СH3C(=O)NH группа замещена на
-OH nag_oh.smi
-NH2 nag_nh2.smi
-H nag_h.smi
-Ph nag_ph.smi
Для докинга воспользовалась скриптом:
export PATH=${PATH}:/home/preps/golovin/progs/bin
for i in nag_h nag_oh nag_nh2 nag_ph ;do
obgen ${i}.smi > ${i}.mol
babel -imol ${i}.mol -opdb ${i}.pdb
/home/preps/golovin/progs/bin/prepare_ligand4.py -l ${i}.pdb -o ${i}.pdbqt
vina --config vina.cfg --receptor 5.pdbqt --ligand ${i}.pdbqt --out ${i}_5.pdbqt --log ${i}_5.log
vina --config vina.cfg --receptor 5_rigid.pdbqt --flex 5_flex.pdbqt --ligand ${i}.pdbqt --out ${i}_5_fr.pdbqt --log ${i}_5_fr.log
done
Файлы можно посмотреть здесь
Данные для:
-OH
Неподвижный докинг
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -5.4 0.000 0.000
2 -4.8 4.467 6.170
3 -4.8 4.262 7.165
Подвижный докинг
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -5.3 0.000 0.000
2 -4.8 3.495 4.770
3 -4.8 3.221 5.378
PyMol:
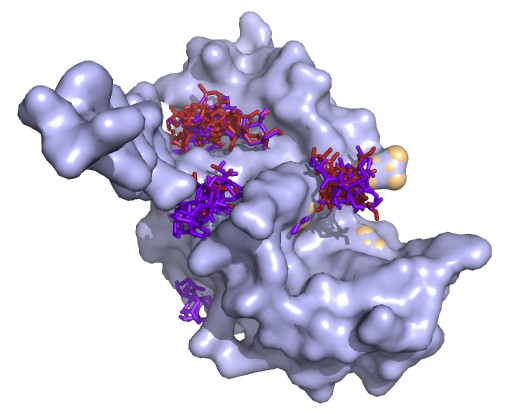
-NH2
Неподвижный докинг
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -5.8 0.000 0.000
2 -5.7 2.333 6.877
3 -5.3 2.697 6.594
Подвижный докинг
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -5.6 0.000 0.000
2 -5.3 2.013 3.009
3 -4.8 8.433 10.679
PyMol:
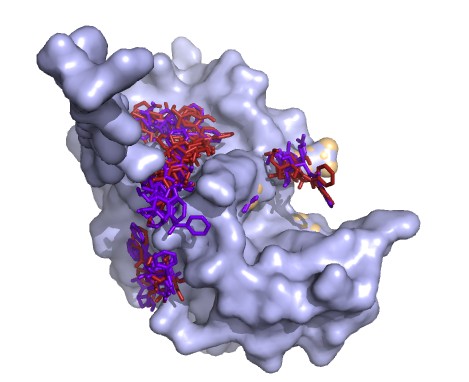
-Ph
Неподвижный докинг
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -5.8 0.000 0.000
2 -5.7 2.344 6.859
3 -5.4 2.622 6.720
Подвижный докинг
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -4.7 0.000 0.000
2 -4.5 2.584 4.080
3 -4.4 2.794 6.106
PyMol:
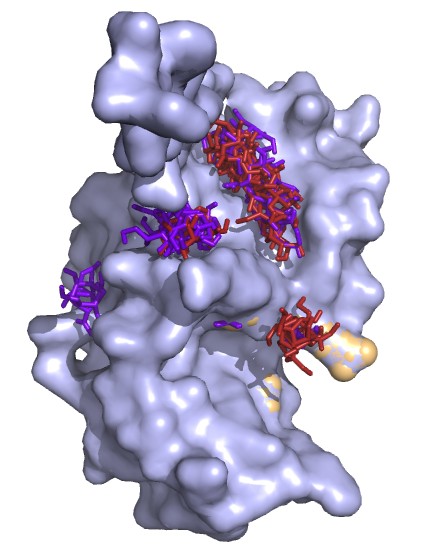
-H
Неподвижный докинг
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -4.4 0.000 0.000
2 -4.3 3.485 4.478
3 -4.2 9.046 10.688
Подвижный докинг
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -4.4 0.000 0.000
2 -4.2 4.014 5.208
3 -4.2 4.625 7.031
PyMol:
Самая высокая афинность у лиганда с фенильной группой.
© Karavaeva Julia 2009





