В качестве белка-прототипа был задан белок из кишечной палочки, описание его свойств найдем на сайте EcoCyc.
Белок RbsR_Ecoli представляет собой репрессор рибозного оперона.
Имя гена - rbsR.
Длина - 330 а.о.
Молекулярный вес белка: 36.612 kD
pI: 5.32
Реакция : D-ribose + RbsR = RbsR-ribose
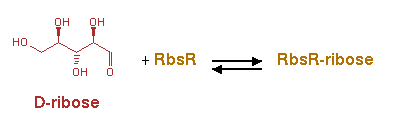
Функции в GO :
| Biological Process: | GO:0006350 - транскрипция; GO:0006355 - регуляция транскрипции; GO:0016052 - карбогидратный каталитический процесс |
| Molecular Function: | GO:0005515 - связывание с белком GO:0003677 - связывание с ДНК GO:0003700 - транскрипционный фактор активности GO:0016564 - транскрипционный репрессор активности |
| Cellular Component: | GO:0005622 - внутриклеточный GO:0005737 - цитоплазма |
Транскрипция этого оперона начинается, когда существует недостаток глюкозы и когда физиологический индуктор, D-рибоза, связывается с геном - репрессором RbsR. Когда D-рибоза связывается с RbsR, белок становится инактивированным, потому что уменьшается его аффиность к связыванию.
- Создание хорошего множественного выравнивания доменов группы белков frur.
Доменная структура белка rbsR_Ecoli была найдена в БД PFAM.
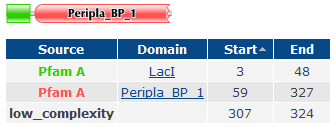
Эффектор-связывающий домен - Peripla_BP_1. Выравнивание эффекторсвязывающих доменов возьмем для него из этой же БД. Скачаем выравнивание всех доменов этого типа в формате FASTA.
Затем получим скрипт для того, чтобы из полного выравнивания получить выравнивание нужных доменов, которые заданы в файле frur.chmod +x scr.scr ./scr.scr
Файл с выравниванием сохраним в рабочей директории с названием PF00532_frur.fasta, где PF00532-идентификатор домена, frur - имя группы специфичности. - Создание единого множественного выравнивания эффекторных доменов всех групп специфичности.
Выравнивание заданных для исследования доменов импортируем в Genedoc и объявим его последовательности новой группой (кнопка меню "Groups=>Edit sequence groups"). Назовем группу. Зададим также цвет для маркировки последовательностей этой группы.
Затем последовательно будем добавлять по одному выравниванию доменов с разной специфичностью (кнопка "S"), каждый раз объявляя новую группу.
Получим раскраску по группам (меню "Groups"), наиболее наглядно иллюстрирующую различия между группами. Сохраним полученное выравнивание. Изображение раскрашенного выравнивания тоже сохраним.Группы окрашены:
galrs - темно-синий gntr - серо-зеленый ptxs - рыжий purr - бурый scrr - тсветло-голубой thur_rafr - серый trer - серо-голубой laci - розовый mali - фиолетовый rbsr - желтый frur - зеленый
В позиции 5 этих выравниваний наблюдается практически полностью консервативный участок, кроме thur_rafr и trer в их последовательностях встречаются гепы на этих участках. Так же практически полностью совпадает позиция 85, 112, 145. Если внимательно рассмотреть rbsr выравнивание, то заметно, что большинство консервативных участков совпадает с аналогичными консервативными участками в других группах.
- Создание лого-изображения полного выравнивания эффекторных доменов и выравнивания доменов группы специфичности rbsr.
Используем ресурс WebLogo. Получили лого-изображение всех эффекторных доменов, а так же лого-изображение для группы rbsr
- Добавление веса в выравнивание.
Выполняем с помощью pwf из пакета PFTOOLs:
pfw -m PF00532_rbsr.fasta > out.weighted.fasta
- Построение профиля.
Выполняем с помощью команды:
pfmake -m out.weighted.fasta /usr/share/pftools23/blosum45.cmp > myprofile.prf
Затем нормируем профиль относительно случайной базы, выполняем следующим образом:
autoscale -m myprofile.prf > myprofile.scaled.prf
Нормированный профиль так же сохраняем. - Поиск по профилю в выборке последовательностей
Выполняем с помощью команды:
pfsearch -C 10.0 -f myprofile.scaled.prf Marinomonas.fasta > marim.search
Получаем файл, в котором указан номер белка и его длина.
Получилось, что подходит только 1 белок:
> 1851 MSSTIKDVALRAGVSTTTVSHVLNKTRFVAKTTQERVFQAAEELNYAPSAVARSLKVKNTKSLGMLVTTT LNPFFAELVNEVEKCCYREGYNLVLCNTDGELEKTNSYLRMLTQKRVDGILVMCSAYDDSLFSSLIGQRN LPMVVMDWGPSDDYVDRIQDNSVKGAHLAIQHLIEQGHKRIAYIGGPLEKLPAKQRLDGFMEAMEQANLP VQGDWVIESDFEFEGGKLGMRQLLSCHNLPSAVFVANDAMAMGAMSEAQLSGVKIPQQLSIIGYDNCMYS AYFSPPLTTINQPKERLAELAISTMIERIENPRQEGKMIMLEPNLVVRSSVAAVSAE
Выравнила с помощью muscle данный белок с последовательностями из выравнивания сделанного ранее.

Далее, мною была найдена структура белка-предшественника RBSR_ECOLI c рибозой. Затем получила аминокислоты, которые взаимодействуют с белком на расстоянии 3.5А:
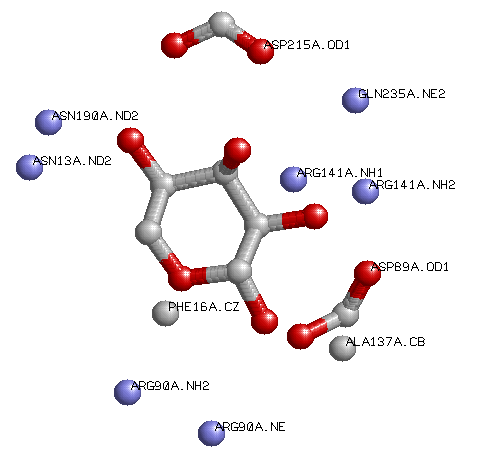
Получила список аминокислот:
ASN 13 N PHE 16 F ASP 89 D ARG 90 R ALA 137 A ARG 141 R ASN 190 N ASP 215 D GLN 235 Q
После этого смотрим, чтобы эти аминокислоты были консервативны в найденных позициях.

На основании полученных данных можно сделать вывод о том, что данный белок связывается с рибозой, то есть выполняет такую же функцию как и белок-предшественник.
©