Для этого используем базу данных PDBSum ; с страницы структуры 1LI5 переходим по гиперссылке "PROCHECK" (слева).
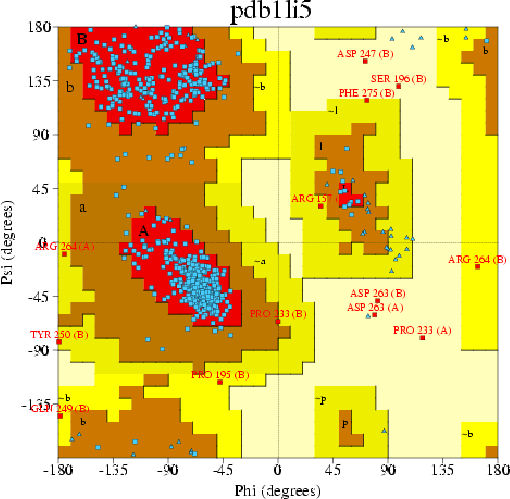
Посмотрим карту Рамачандрана для данной структуры:

Как видно на изображении, число остатков (отличных от Gly и Pro), попавших в запрещенные области на карте, равно 4. Это соответсвует 0.6% от всех остатков. В препочитаемые области попало 607 остатков (91.3%). Если рассмотреть G-фактор, то можно сделать вывод о том, что структура является довольно хорошей.
На сервере EDS на странице структуры 1LI5 выберем гиперссылку "Significant regions". На этой странице мы можем узнать количество остатков каждой цепи белка со значением RSR, отклонившимся от своего среднего значения больше чем на 2 сигмы :
разрешение для данной структуры: 2.20Å;<RSR>+2.00*sigma: 2.00-2.20Å
| Цепь | Число остатков с высоким значеним Z-score |
| A | 10 |
| B | 31 |
Как видим, число таких остатков во второй цепи больше, несмотря на то, что они практически идентичны.
Посмотрим на один из таких остатков, пусть это будет триптофан 184 из цепи B.
Для него значения:
пространственный R-фактор (<RSR>) = 0.215;
коэффициент корреляции для электронной плотности ("real-space correlation coefficient") = 0.905;
Z-score = 2.535714;
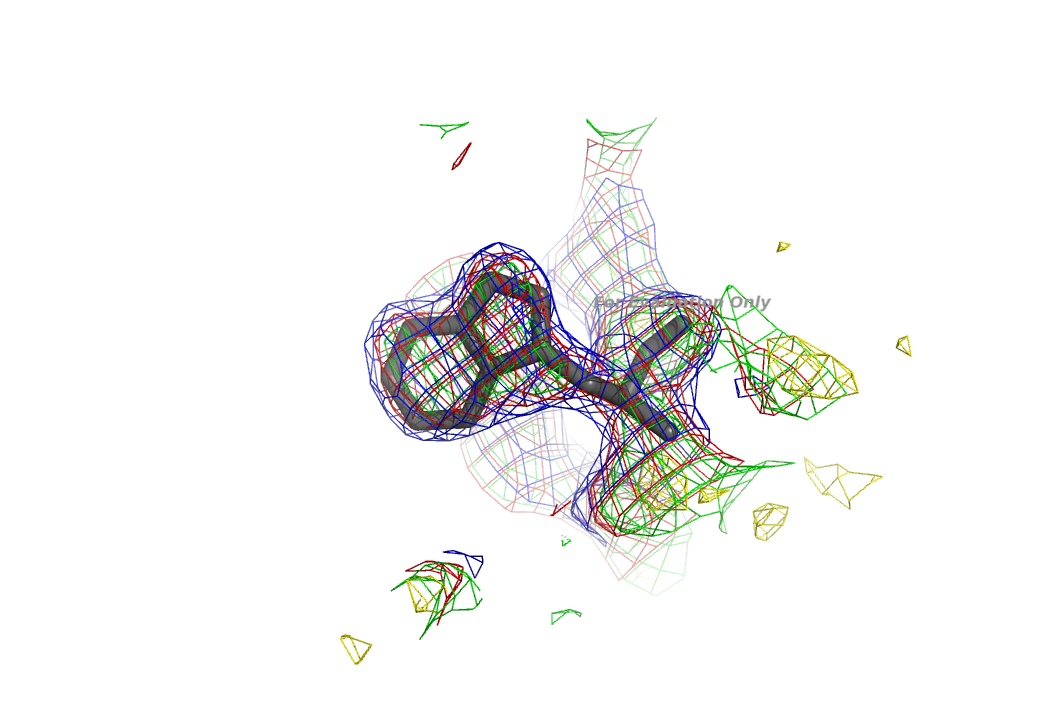
Электронная плотность на уровне 0.5 - синего цвета, 1.0 - красного цвета; 1.5 - зеленого цвета; 3.0 - желтого цвета.
Рассмотрим, как выглядит плотность на одном уровне 0.5:
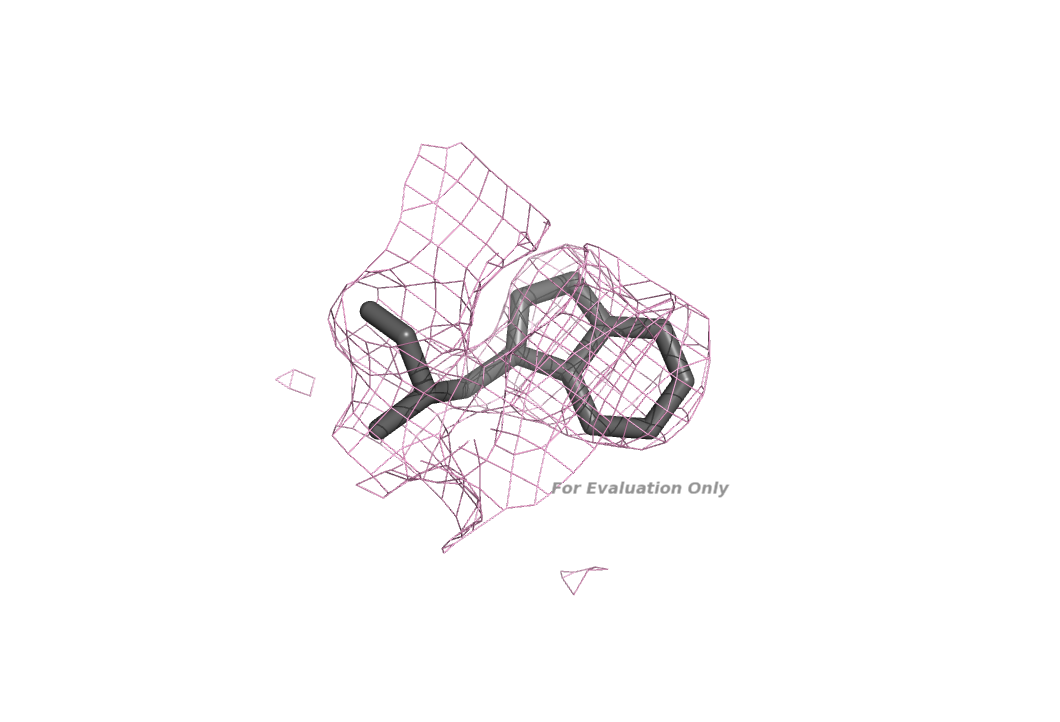
Как видно из изображения, вписать этот остаток в электронную плотность можно и другим способом. Так же можно сказать, что поскольку для этого остатка данные RSR > 0.1; Z-score > 2, то данные эксперементальной электронной плотности довольно хорошо соответствуют реальной.
Со страницы структуры 1LI5 в EDS перейдем по гиперссылке "PDB_REDO". Изучим таблички "R-values" и "WHAT_CHECK validation". В таблице рассмотрим различия в показателях у оптимизированной ("Fully optimised") структуры и неоптимизированной:
| Показатели качества | Неоптимизированная структура | Полностью оптимизированная структура |
| R | 0.2450 | 0.2087 |
| R-free | 0.2830 | 0.2454 |
| σR-free | 0.0028 | 0.0024 |
| R-free Z-score | 9.07 | 5.71 |
| 1st generation packing quality | NA | -0.140 |
| 2nd generation packing quality | -2.294 | -0.576 |
| Ramachandran plot appearance | -1.926 | -0.069 |
| Chi-1/Chi-2 rotamer normality | -1.551 | 0.297 |
| Backbone conformation | -0.197 | -0.082 |
| Bond length RMS Z-score | 0.476 | 0.496 |
| Bond angle RMS Z-score | 0.827 | 0.696 |
| Total number of bumps | 176 | 90 |
| Unsatisfied H-bond donors/acceptors | 50 | 40 |
Как видим, после оптимизации следующие параметры уменьшились: значение R-value; R-free; σR-free; R-free Z-score;валентные углы; количество атомов с недостоверно коротким межатомным расстоянием; число атомов, способных образовывать водородные связи, но их не образующих.
В целом, оптимизация повысила качество структуры белка записи 1LI5.
Получим оптимизированную структуру нашего белка, для этого скачаем ее с страницы графа "Fully optimised structure". Попробуем выяснить, чем она отличается от исходной, для этого сравним лучше ли остаток Trp 184 из цепи B вписывается в электронную плотность. Посмотрим на его изображение теперь:
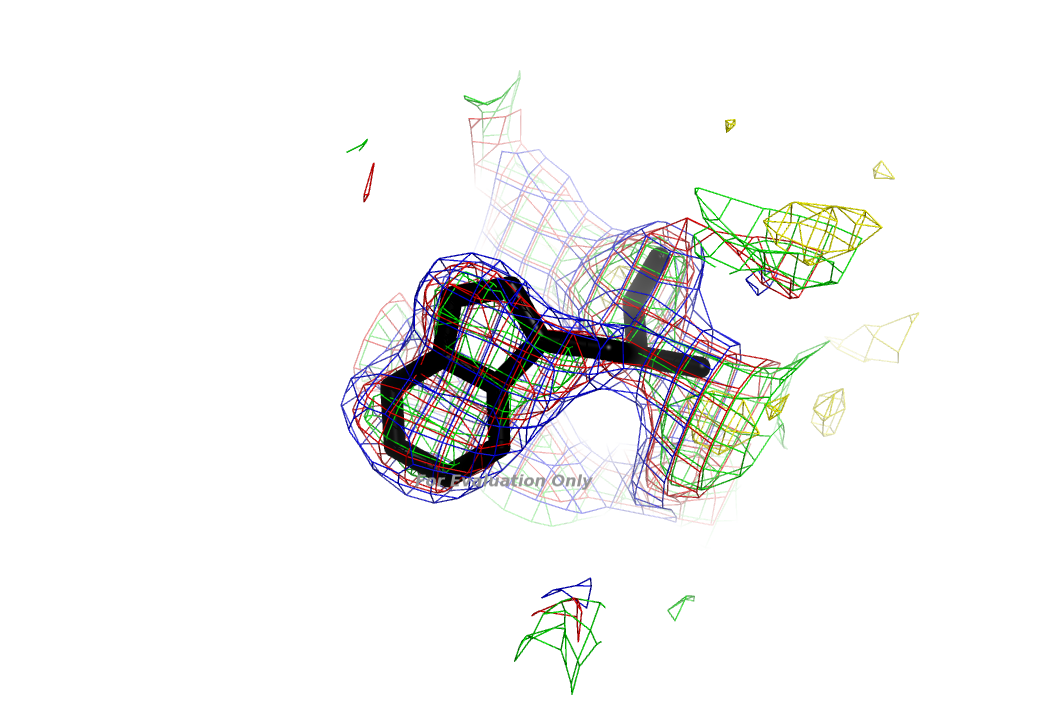
Электронная плотность на уровне 0.5 - синего цвета, 1.0 - красного цвета; 1.5 - зеленого цвета; 3.0 - желтого цвета.
Если рассмотреть только плотность на уровне 0.5, то видим:
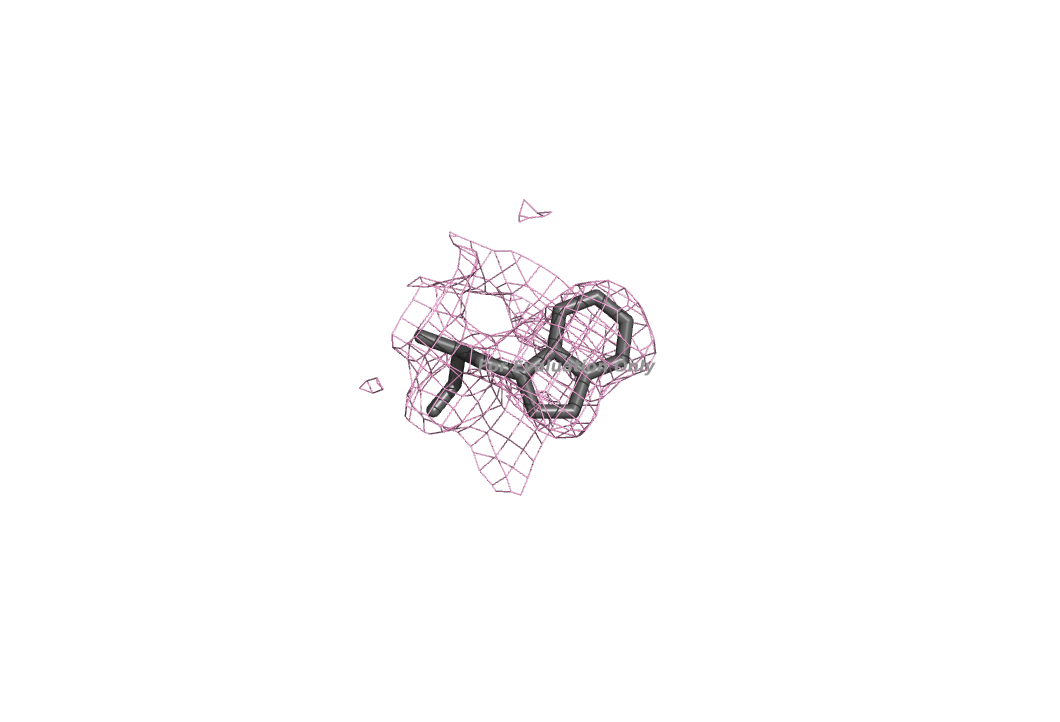
Остаток так же хорошо вписывается в полученную электронную плотность, даже чуть-чуть лучше. Посмотрим отличия в расположениях этого остатка в двух моделях:
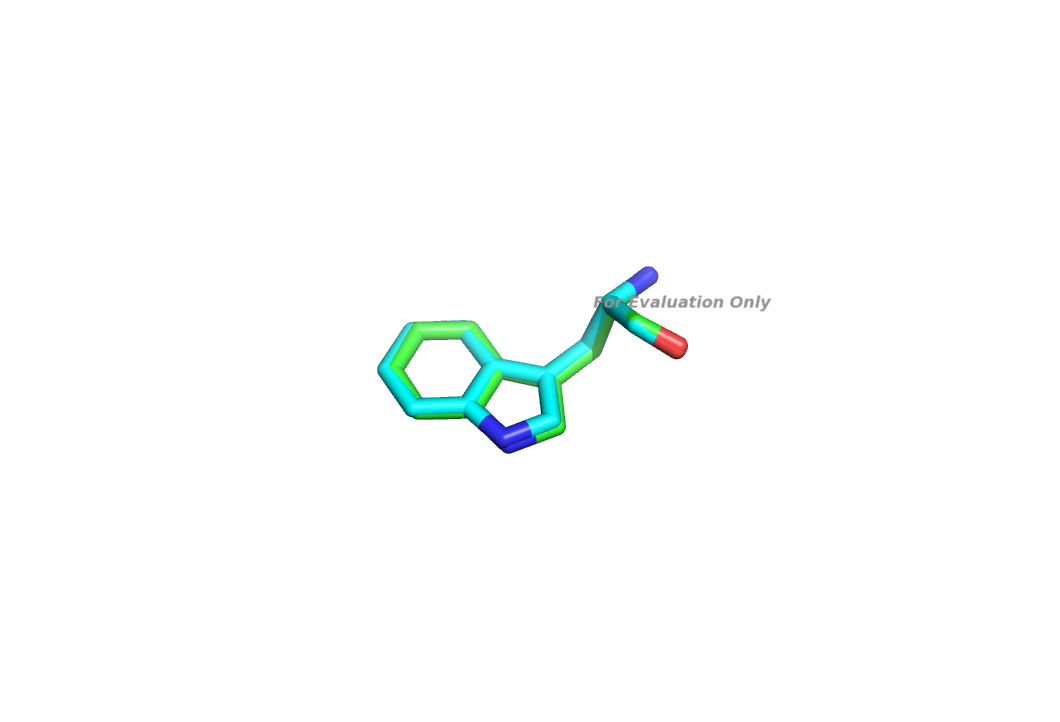
Остатки накладываются друг на друга, заметны лишь небольшие изменения в координатах атомов.
Исследовав эту выдачу можно узнать:
есть ли кристаллографическая симметрия между молекулами молекулярную массу всех цепей полимера объем элементарной ячейки есть ли стерические нарушения в структуре молекулы (доп. внутрицепочечные ковалентные связи, перекрывания атомов и т.д.) вмешиваются ли доп.атомы в образование водородных связей нет ли больших молекул (сахара,липиды), образующих водородные связи с атомами белка есть ли неполные атомы и атомы с нулевой полнотой карта Рамачандрана для каждой цепи вторичная структура цепей по данным DSSP нарушения в последовательностях белка (округления координат, искуственные цепи, недостающие атомы, С-концевой азот и тд ) температурный B-фактор и информация о нем нарушения в названиях атомов, остатков, цепей нарушения в геометрии структуры нарушения в торсионных углах атомы с коротким межатомным расстоянием упаковка, доступность и пронизывание вода и водородные связи ну и, конечно, краткое финальное саммари по всей выдаче
О теперь посмотрим,что именно было в такой выдаче для моего белка 1LI5:
Нет кристаллографической симметрии между молекулами - нет экстра кристаллографической симметрии между различными молекулами Коэффициент Метьюса очень низкий - плотность структуры белка в кубических Ангстремах на Дaлтон Нормальные значения лежат между 1.5 (плотно упакованой) и 4.0 (свободно упакованной, много пространства для растворителя).Факт, что у 1LI5 это значение ниже чем 1.5 возможно вызван неверно оцененной ценностью Z на карте CRYST1. Молекулярный вес всех полимерных цепей: 86221.008
V отдельной ячейки = 2048971.1
Сложность ячейки: 16 Коэффициент Метьюса для атомов Vm = 1.485 Все атомы расположены достаточно далеко от осей симметрии Нет странностей в взаимодействиях внутренних цепей - нет ковалентных связей между разными цепями Нет повторений в названиях лигандов Либо структура не содержит дополнительные атомы, либо они не были перепутанны, либо ошибки не замечены Во всех случаях использовался основной дополнительный атом - нет необходимости делать исправления в атомах Не обнаруженно остатков в лигандах Топология Лигандов хорошая - все лиганды включены в оптимизацию с водородными связями Нет прикрепленных групп, вмешивающихся в структуру с водородными связями Нет атомов с высоким занятием в специальных положениях У всех остатков полная и одинаковая основа Нет обнаруженных атомов с нулевым занятием Карты Рамачандрана:
| Цепь А | Цепь В |
|
|
Не обнаруженно округленных координат Нет искусственных посторонних цепей Недостающие атомы в входном файле: 10 ARG ( 10-) A - CG 10 ARG ( 10-) A - CD 10 ARG ( 10-) A - NE 10 ARG ( 10-) A - CZ 10 ARG ( 10-) A - NH1 10 ARG ( 10-) A - NH2 16 LYS ( 16-) A - CG 16 LYS ( 16-) A - CD 16 LYS ( 16-) A - CE 16 LYS ( 16-) A - NZ 60 LYS ( 60-) A - CG 60 LYS ( 60-) A - CD 60 LYS ( 60-) A - CE 60 LYS ( 60-) A - NZ 73 LYS ( 73-) A - CG 73 LYS ( 73-) A - CD 73 LYS ( 73-) A - CE 73 LYS ( 73-) A - NZ 76 LYS ( 76-) A - CG 76 LYS ( 76-) A - CD 76 LYS ( 76-) A - CE 76 LYS ( 76-) A - NZ 98 LYS ( 98-) A - CG 98 LYS ( 98-) A - CD 98 LYS ( 98-) A - CE Нет С-коневого азота Не хватает покрова на некоторых С-концевых атомах Веса проверенны хорошо Все занятия, вцелом, составляют 1, кроме недостающих атомов Проблема в среднем В-факторе - он слишком большой 32.943. В норме от 10 до 20. Номенклатура нормальныя для валина, треонина, изолейцина, лейцина, аргинина, фосфатных групп, тяжелых атомов и имен цепей Проблемы с названиями со следующими тирозинами, фенилаланинами, аспарагиновой килотой, глутаминовой кислотой: 59 TYR ( 59-) A - 234 TYR ( 250-) A - 268 TYR ( 284-) A - 282 TYR ( 298-) A - 288 TYR ( 304-) A - 304 TYR ( 320-) A - 445 TYR ( 59-) B - 608 TYR ( 250-) B - 644 TYR ( 291-) B - 673 TYR ( 320-) B - 47 PHE ( 47-) A - 144 PHE ( 144-) A - 165 PHE ( 181-) A - 258 PHE ( 274-) A - 259 PHE ( 275-) A - 322 PHE ( 338-) A - 334 PHE ( 350-) A - 433 PHE ( 47-) B - 519 PHE ( 144-) B - 691 PHE ( 338-) B - 703 PHE ( 350-) B - 713 PHE ( 360-) B - 48 ASP ( 48-) A - 71 ASP ( 71-) A - 90 ASP ( 90-) A - 99 ASP ( 99-) A - 311 ASP ( 327-) A - 434 ASP ( 48-) B - 455 ASP ( 69-) B - 465 ASP ( 90-) B - 638 ASP ( 285-) B - 680 ASP ( 327-) B - 13 GLU ( 13-) A - 22 GLU ( 22-) A - 95 GLU ( 95-) A - 271 GLU ( 287-) A - 320 GLU ( 336-) A - 379 GLU ( 395-) A - 408 GLU ( 22-) B - 640 GLU ( 287-) B - 689 GLU ( 336-) B - 707 GLU ( 354-) B - Анамольные длины связей: 231 ASP ( 247-) A - N -C 1.24 -4.2 248 ARG ( 264-) A - CA C 1.61 4.1 250 LYS ( 266-) A - N -C 1.21 -5.7 532 ARG ( 157-) B - CA C 1.63 5.0 605 ASP ( 247-) B - N CA 1.35 -5.6 606 GLY ( 248-) B - N CA 1.37 -5.1 607 GLN ( 249-) B - N CA 1.65 10.2 607 GLN ( 249-) B - CA C 1.65 5.8 608 TYR ( 250-) B - N CA 1.61 8.2 608 TYR ( 250-) B - N -C 1.45 6.3 Нормальная изменяемость длин связей Аномальные углы связей: 31 THR ( 31-) A - N CA C 88.59 -8.1 32 VAL ( 32-) A - N CA CB 100.84 -5.7 71 ASP ( 71-) A - N CA C 88.24 -8.2 72 ASP ( 72-) A - N CA C 124.86 4.9 72 ASP ( 72-) A - N CA CB 103.04 -4.4 197 ASN ( 213-) A - CA CB CG 116.95 4.3 206 ASP ( 222-) A - N CA C 123.30 4.3 208 HIS ( 224-) A - CG ND1 CE1 109.89 4.3 217 PRO ( 233-) A - -C N CA 165.75 8.6 217 PRO ( 233-) A - -C N CD 65.39 -14.5 217 PRO ( 233-) A - CA CB CG 95.88 -4.5 217 PRO ( 233-) A - CD N CA 102.14 -7.0 231 ASP ( 247-) A - -C N CA 109.57 -6.7 233 GLN ( 249-) A - -CA -C N 106.11 -4.9 233 GLN ( 249-) A - CG CD NE2 126.32 6.6 233 GLN ( 249-) A - NE2 CD OE1 113.42 -9.2 234 TYR ( 250-) A - -CA -C N 105.74 -5.2 248 ARG ( 264-) A - -C N CA 112.90 -4.9 249 GLU ( 265-) A - -C N CA 148.00 14.6 249 GLU ( 265-) A - N CA CB 122.48 7.0 446 LYS ( 60-) B - N CA C 98.42 -4.6 531 SER ( 156-) B - -C N CA 113.09 -4.8 531 SER ( 156-) B - CA CB OG 119.69 4.3 532 ARG ( 157-) B - -CA -C N 126.07 4.9 532 ARG ( 157-) B - -C N CA 114.36 -4.1 533 GLN ( 158-) B - -C N CA 129.39 4.3 571 ASN ( 213-) B - -C N CA 113.50 -4.6 571 ASN ( 213-) B - CA CB CG 117.44 4.8 582 HIS ( 224-) B - CG ND1 CE1 109.72 4.1 591 PRO ( 233-) B - -C N CA 179.71 11.4 591 PRO ( 233-) B - -C N CD 76.11 -11.9 591 PRO ( 233-) B - CD N CA 103.70 -5.9 604 HIS ( 246-) B - -C N CA 111.36 -5.7 604 HIS ( 246-) B - N CA C 130.60 6.9 604 HIS ( 246-) B - CA CB CG 109.11 -4.7 605 ASP ( 247-) B - -C N CA 111.66 -5.6 606 GLY ( 248-) B - -O -C N 131.11 5.1 606 GLY ( 248-) B - -CA -C N 103.22 -6.5 606 GLY ( 248-) B - -C N CA 128.82 4.8 607 GLN ( 249-) B - CG CD NE2 126.48 6.7 607 GLN ( 249-) B - NE2 CD OE1 112.93 -9.7 624 LYS ( 266-) B - N CA C 124.54 4.8 625 MET ( 267-) B - -CA -C N 107.02 -4.6 627 PHE ( 274-) B - -C N CA 114.49 -4.0 Нормальная изменяемость углов связей Замечены хиральные отклонения Есть неправильные торсионные углы: 217 PRO ( 233-) A - -3.1 591 PRO ( 233-) B - -3.1 608 TYR ( 250-) B - -2.9 70 ILE ( 70-) A - -2.7 218 HIS ( 234-) A - -2.5 592 HIS ( 234-) B - -2.5 30 ILE ( 30-) A - -2.4 424 ILE ( 38-) B - -2.4 581 ILE ( 223-) B - -2.3 82 GLY ( 82-) A - -2.2 234 TYR ( 250-) A - -2.2 283 ARG ( 299-) A - -2.2 607 GLN ( 249-) B - -2.2 553 PRO ( 195-) B - -2.2 85 PHE ( 85-) A - -2.1 703 PHE ( 350-) B - -2.1 313 THR ( 329-) A - -2.0 255 LEU ( 271-) A - -2.0 Есть аномальные ротатомеры: 34 ASP ( 34-) A - 0.38 241 SER ( 257-) A - 0.39 Есть аномально короткие межатомные расстояния: 216 PHE ( 232-) A - O <--> 217 PRO ( 233-) A - CD 0.81 1.59 INTRA B3 216 PHE ( 232-) A - C <--> 217 PRO ( 233-) A - CD 0.79 1.51 INTRA B2 228 CYS ( 244-) A - SG <--> 758 HOH (1101 ) A - O 0.67 2.33 INTRA 590 PHE ( 232-) B - C <--> 591 PRO ( 233-) B - CD 0.56 1.74 INTRA B2 230 HIS ( 246-) A - CD2 <--> 758 HOH (1099 ) A - O 0.55 2.25 INTRA 590 PHE ( 232-) B - O <--> 591 PRO ( 233-) B - CD 0.38 2.02 INTRA B3 593 HIS ( 235-) B - CD2 <--> 614 HIS ( 256-) B - NE2 0.38 2.72 INTRA 26 TYR ( 26-) A - OH <--> 193 CYS ( 209-) A - SG 0.37 2.63 INTRA 512 ALA ( 137-) B - O <--> 515 GLY ( 140-) B - N 0.36 2.34 INTRA BF 28 CYS ( 28-) A - SG <--> 219 HIS ( 235-) A - CE1 0.35 3.05 INTRA 219 HIS ( 235-) A - CD2 <--> 240 HIS ( 256-) A - CE1 0.32 2.88 INTRA 604 HIS ( 246-) B - O <--> 605 ASP ( 247-) B - CB 0.32 2.28 INTRA BF 554 SER ( 196-) B - O <--> 557 GLY ( 199-) B - N 0.27 2.43 INTRA BF 581 ILE ( 223-) B - CD1 <--> 613 MET ( 255-) B - SD 0.27 3.13 INTRA 252 SER ( 268-) A - N <--> 257 ASN ( 273-) A - OD1 0.27 2.43 INTRA 72 ASP ( 72-) A - OD1 <--> 186 ARG ( 202-) A - NH2 0.25 2.45 INTRA BF 520 ASP ( 145-) B - O <--> 523 THR ( 148-) B - OG1 0.25 2.15 INTRA BF 523 THR ( 148-) B - O <--> 525 PRO ( 150-) B - CD 0.24 2.56 INTRA BF 470 GLU ( 95-) B - CD <--> 631 ARG ( 278-) B - NH2 0.23 2.87 INTRA BF 620 VAL ( 262-) B - N <--> 623 GLU ( 265-) B - O 0.23 2.47 INTRA BF 508 HIS ( 133-) B - O <--> 520 ASP ( 145-) B - N 0.23 2.47 INTRA BF 30 ILE ( 30-) A - CD1 <--> 96 MET ( 96-) A - SD 0.23 3.17 INTRA BF 101 ASP ( 101-) A - OD1 <--> 107 ARG ( 107-) A - NH1 0.23 2.47 INTRA BF 624 LYS ( 266-) B - CB <--> 625 MET ( 267-) B - N 0.21 2.49 INTRA BF 153 GLY ( 153-) A - CA <--> 220 GLU ( 236-) A - CD 0.21 2.99 INTRA Распределение внутренних и наружных остатков нормальное Низкий Z-score для некоторых остатков: 16 LYS ( 16-) A - -3.35 402 LYS ( 16-) B - -3.33 416 ILE ( 30-) B - -3.28 677 ARG ( 324-) B - -3.13 308 ARG ( 324-) A - -3.12 10 ARG ( 10-) A - -3.01 537 MET ( 179-) B - -2.84 622 ARG ( 264-) B - -2.79 419 TYR ( 33-) B - -2.69 624 LYS ( 266-) B - -2.65 506 LYS ( 131-) B - -2.64 488 ARG ( 113-) B - -2.57 521 VAL ( 146-) B - -2.54 253 LYS ( 269-) A - -2.50 Водные контакты нормальные Вода не нуждается в передвижениях Есть одна вода без водороных связей: 758 HOH (1100 ) A - O Вода не описанa для данных позиций: 30 ILE ( 30-) A - N 33 TYR ( 33-) A - N 37 HIS ( 37-) A - N 65 ARG ( 65-) A - NH1 69 ASP ( 69-) A - N 70 ILE ( 70-) A - N 71 ASP ( 71-) A - N 86 VAL ( 86-) A - N 115 THR ( 115-) A - N 192 GLU ( 208-) A - N 193 CYS ( 209-) A - N 194 SER ( 210-) A - N 194 SER ( 210-) A - OG 197 ASN ( 213-) A - ND2 206 ASP ( 222-) A - N 280 GLY ( 296-) A - N 289 SER ( 305-) A - N 319 GLY ( 335-) A - N 423 HIS ( 37-) B - N 425 GLY ( 39-) B - N 428 ARG ( 42-) B - NH2 451 ARG ( 65-) B - NH1 456 ILE ( 70-) B - N 464 VAL ( 89-) B - N 482 ARG ( 107-) B - N 516 ASP ( 141-) B - N 538 ASP ( 180-) B - N 556 TRP ( 198-) B - N 564 HIS ( 206-) B - N 565 ILE ( 207-) B - N 566 GLU ( 208-) B - N 567 CYS ( 209-) B - N 568 SER ( 210-) B - N 571 ASN ( 213-) B - ND2 573 LYS ( 215-) B - N 600 SER ( 242-) B - OG 614 HIS ( 256-) B - NE2 631 ARG ( 278-) B - N 649 GLY ( 296-) B - N 658 SER ( 305-) B - N 688 GLY ( 335-) B - N 69 ASP ( 69-) A - OD1 200 GLN ( 216-) A - OE1 208 HIS ( 224-) A - ND1 219 HIS ( 235-) A - NE2 240 HIS ( 256-) A - NE2 508 HIS ( 133-) B - ND1 582 HIS ( 224-) B - ND1 593 HIS ( 235-) B - NE2 599 GLN ( 241-) B - OE1