Так же используем International Union of Biochemistry and Molecular Biology.
Было обнаружено, что EC=2.7.1.23.
Теперь внимательно изучим каждый пункт данного кода:
2.7.1.23 - группа трансфераз ( Transferase ). Это ферменты, осуществляющие перенос групп, например, метильной группы или гликозил группы, от одного соединения (как правило, рассматривается в качестве донора) к другому соединению (как правило, рассматривается в качестве акцептора). Классификация основана на схеме "донор-акцептор-переносимая группа". Принятые имена, как правило, формируются как "акцептор - группа переноса" или "донор - группа переноса". Во многих случаях, доноры это кофактор (кофермент), несущий группу для переноса . Аминотрансферазы представляют собой особый случай (подкласс ЕС 2,6).
2.7.1.23 - группа трансфераз, осуществляющих передачу фосфор-и азотсодержащих групп (Transferring phosphorus-containing groups). Это довольно большая группа ферментов, которые осуществляют не только передачи фосфата, но и дифосфата, нуклеотидных остатков и др. Фосфотрансферазы подразделяется в соответствии с акцепторной группы.
2.7.1.23 - группа фосфотрансфераз с гидроксильной группой в качестве акцептора (Phosphotransferases with an Alcohol Group as Acceptor).
2.7.1.23 - NAD+-киназы (NAD+ kinase) или ATP:NAD+ 2'-фосфотрансферазы (ATP:NAD+ 2'-phosphotransferase) .
Реакция, катализируемая этим ферментом: ATP + NAD+ = ADP + NADP+
Графическое изображение катализируемой реакции:
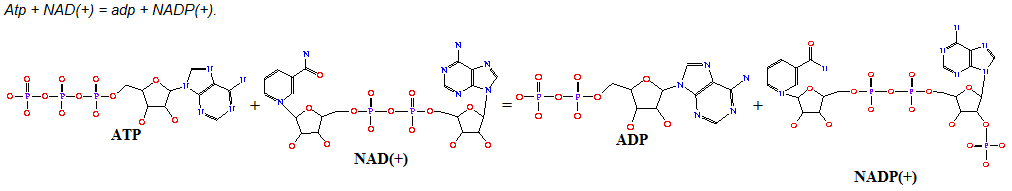
В документе UNIPROT с описанием заданного белка (была открыта в прошлом задании) найдем имя локуса его гена, в данном случае это b2615. Теперь открыв главную страничку БД KEGG, произведем поиск гена по названию его локуса ( запрос есо:b2615).
В открывшемся документе найдем описание метаболических путей, ассоциированных с изучаемым геном:
1. eco00760 Метаболизм никотина и никотинамида (Nicotinate and nicotinamide metabolism)
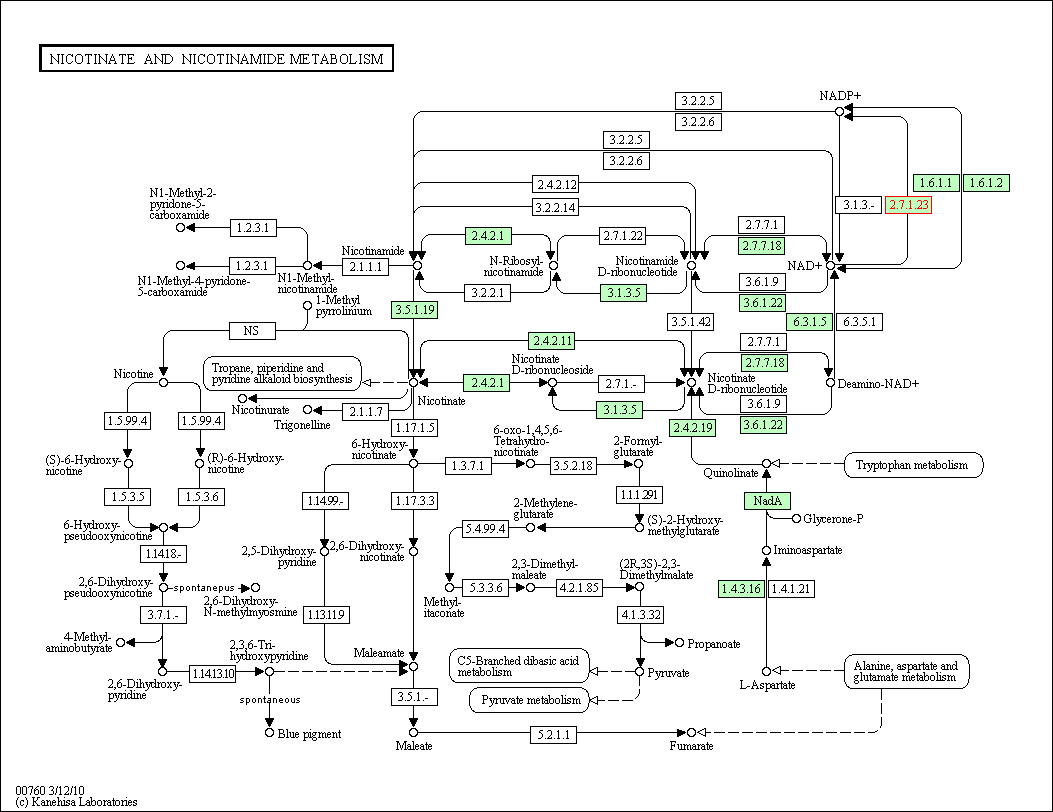
Работа фермента отмечена красным.
Скачать карту "Метаболизм никотина и никотинамида"
2. eco01100 Метаболические пути (Metabolic pathways)Откроем страницу оглавления KEGG и найдем ссылку на базу данных химических соединений (KEGG LIGAND), затем проведем поиск по каждому из названий веществ.
(поиск проводим по английским названиям!)
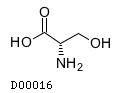
1. L-серин (L-Serine):
идентификатор - C00065 (код для kegg drug: D00016)
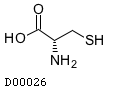
2. L-цистеин ( L-Cysteine ):
идентификатор - C00097 (код для kegg drug: D00026)
В поле запроса вводим идентификаторы соединений, для которых проводим поиск; так же указываем, что хотим раскрасить первое соединение красным, а второе - зеленым. После ввода запроса на экране появляется список карт. Используем ту, для которой указаны два идентификатора. Убедимся, что оба вещества раскращены и выберем кратчайший путь! Если подвести курсор к любому интермедиату на карте, высветится код данного соединения.
Путь: Метаболизм серы (восстановление и фиксация).
Вещества: L-цистеин (C00097) и L-серин (C00065).
Цепочка: L-серина -> L-цистеин
Промежуточные соединения: О-Ацетил-L-серин (С00979).
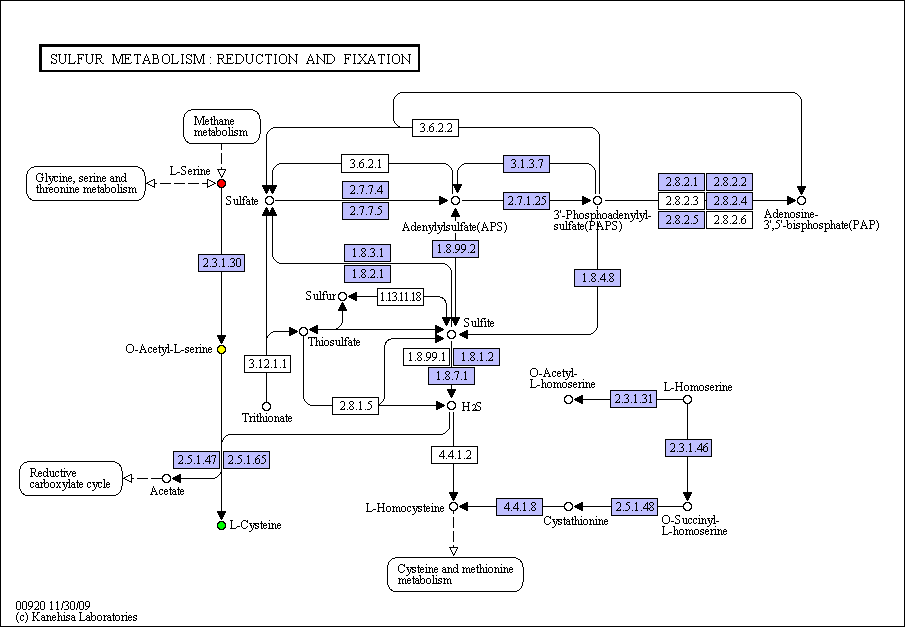
L-цистеин окрашен зеленым. L-серин окрашен красным. Промежуточное вещество - О-Ацетил-L-серин окрашен желтым.
Мною была выбрана данная карта, т.к. она отражает самый кратчайший путь перехода L- серина в L- цистеин.
Скачать карту "Метаболизм серы (восстановление и фиксация)".
Определим, возможна ли выбранная в задании 4 цепочка ферментативных реакций у организмов, перечисленных в таблице ниже.
Изменяем организм в поле Reference map.
| Организм | Возможна ли цепочка реакций (да/нет/неизвестно) |
Обоснование |
| Escherichia coli K-12 MG1655 | присутствуют ферменты, необходимые для осуществления цепочки реакций |
|
| Archaeoglobus fulgidus | нет ферментов, необходимых для осуществления цепочки реакций |
|
| Arabidopsis thaliana (thale cress) | присутствуют ферменты, необходимые для осуществления цепочки реакций (кроме 2.5.1.65 - выполняет аналогичную функцию с 2.5.1.47, поэтому возможен этот процесс) |
|
| Homo sapiens | нет ферментов, необходимых для осуществления цепочки реакций |
По полученным данным можно сделать вывод о том, что этот метаболизм очень важен для жизни организмов, но с процессом эволюции он проходит в большей степени через другие ферментативные реакции.
- Найдем с помощью SRS ферменты с ЕС кодом 4.2.1.24 у человека и археи Archaeoglobus fulgidus.
Для того, чтобы провести поиск необходимо снять опцию маски ( Use wildcards ), чтобы при поиске EC белка находились лишь ферменты с EC=4.2.1.24, и не находились ферменты с похожими EC (например, 4.2.1.240 и тд). Для упрощения запроса воспользуемся тем, что для белков приняты короткие имена. ID белков человека заканчиваются на _HUMAN, белки археи Archaeoglobus fulgidus - на _ARCFU. Для отсева идентичных последовательностей воспользуемся ccылкой UNIREF100. В результате, запрос для поиска в БД UniProt выглядит так:([uniprot-ECNumber:4.2.1.24] & ([uniprot-ID:*_HUMAN] | [uniprot-ID:*_ARCFU]))
Получили 4 белка, все относятся к классу дегидрогеназ дельта-амино-левулиновой кислоты. 1 из них относится к археям - HEM2_ARCFU (O28305), а 3 найдены у человека (причем все они представляют один белок, в последовательности это одни участки только с немного различающимися длинами) - выбираем, несмотря на это, белок HEM2_HUMAN (P13716), потому что последовательности остальных входят в последвательность первого(B7Z3I9_HUMAN; Q6ZMU0_HUMAN).
- Сравнение доменной организации (по PFAM) белков HEM2_HUMAN, B7Z3I9_HUMAN, Q6ZMU0_HUMAN и HEM2_ARCFU.
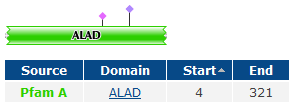
Для этого используем режим SW_InterProMatches (для просмотра находок). - Определение % совпадения последовательностей гомологичных доменов (ALAD) из археи (HEM2_ARCFU) и человека (HEM2_HUMAN).
- Нахождение лучшего ортолога для белков HEM2_ARCFU и HEM2_HUMAN.
Выполняем поиск ортологов с помощью инструментов KEGG.Найдем документ KEGG с описанием гена нужного белка по схеме, описанной в упр.2. На найденной страничке выбираем кнопку "Ortholog", а в открывшемся окне - опцию Best-best (bidirectional best hit),указываем нужную группу организмов и выбираем кнопку "GO".
Для белка HEM2_HUMAN название гена ALAD; для HEM2_ARCFU - hemB. Соответсвующие id в KEGG hsa:210 и afu:AF1974.
Ген ортолог для HEM2_HUMAN среди архей:
Ген: mka:MK0198 Организм: Methanopyrus kandleri Процент идентичности: 0.469 Перекрытие последовательностей: 309
Ген ортолог для HEM2_ARCFU среди эукариот:
Ген: pti:PHATRDRAFT_50723 Организм: Phaeodactylum tricornutum Процент идентичности: 0.489 Перекрытие последовательностей: 321
B7Z3I9_HUMAN - домен хоть и выделен, но не отмечен.
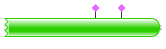
Q6ZMU0_HUMAN - менее похож на белок из Archaeoglobus fulgidus (немного длиннее и у него есть участок low_complexity (низкого сродства)).
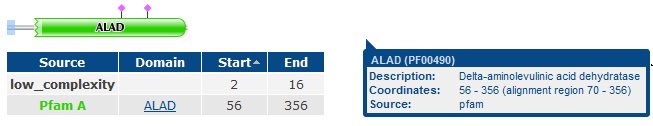
HEM2_HUMAN (P13716)
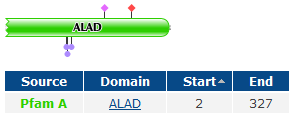
HEM2_ARCFU (O28305)
Воспользуемся программой needle для создания выравнивания. Получили процент сходства 61.4%.

Белок в организме человека соответсвует белку в археи с относительно большим процентом идентичности, белки выполняют одинаковые функции в столь разных и далеких организмах. Аналогичные выводы можно сделать, во время рассмотрения белка HEM2_ARCFU и его ортолога.