Чтобы провести данный вид определения для белка 1LI5, воспользуемся программой DSSP.
Возможно запустить данную программу на kodomo:
dsspcmbi 1LI5.pdb 1LI5.dssp
НО мы скачаем уже готовый результат по FTP с ftp://ftp.cmbi.ru.nl//pub/molbio/data/dssp/
Получаем файл с описанием вторичной структуры. Импортировав этот файл в Exel, посчитаем количество элементов в каждой из цепей (в моем белке это А и В). Одним элементом вторичной структуры будем считать блок идущих подряд букв H (альфа-спираль) или E (бета-тяж).
Поместим данные в таблицу: в цепи А - 13 альфа-спиралей и 12 бета-тяжей, в цепи B - 12 альфа-спиралей и 12 бета-тяжей.
Теперь изучим поля HELIX и SHEET в pdb-файле и сравнис два описания вторичной структуры (вычисленное DSSP и приведённое авторами записи).
| Номер цепи \спирали | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 | 13 | 14 | 15 | 16 | 17 |
| A (по данным dssp) | 38-57 | 72-80 | 85-102 | - | 118-130 | - | 207-217 | - | 234-245 | - | 277-281 | 286-294 | 306-323 | 336-347 | 352-372 | 374-388 | 398-401 |
| A (по описанию в pdb) | 37-58 | - | 84-103 | 113-116 | 117-131 | 146-149 | 206-218 | 227-231 | 233-246 | 268-271 | 276-282 | 285-295 | 305-324 | 335-348 | 351-373 | 373-389 | 397-402 |
| Различия | ~ | не найден в pdb | ~ | не найден dssp | ~ | не найден dssp | ~ | не найден dssp | ~ | не найден dssp | ~ | ~ | ~ | ~ | ~ | ~ | ~ |
| B (по данным dssp) | 38-56 | 87-102 | - | 118-130 | - | 207-216 | - | 234-246 | 277-280 | 286-294 | 306-322 | 336-347 | 352-372 | 374-390 | 398-401 | ||
| B (по описанию в pdb) | 37-57 | 86-103 | 113-116 | 117-131 | 146-149 | 206-217 | 227-231 | 233-247 | 276-281 | 285-295 | 305-324 | 335-348 | 351-373 | 373-391 | 397-402 | ||
| Различия | ~ | ~ | не найден dssp | ~ | не найден dssp | ~ | не найден dssp | ~ | ~ | ~ | ~ | ~ | ~ | ~ | ~ |
| Номер цепи \спирали | 1 | 2 | 3 | 4 | 5 | 6 | 7 | 8 | 9 | 10 | 11 | 12 |
| A (по данным dssp) | 3-6 | 11-14 | 23-27 | 60-65 | 134-136 | 142-144 | 181-186 | 201-203 | 221-225 | 251-254 | 260-262 | 302-304 |
| A (по описанию в pdb) | 3-5 | 12-14 | 22-27 | 60-65 | 134-136 | 142-144 | 181-186 | 201-203 | 221-225 | 251-254 | 260-262 | 302-304 |
| Различия | ~ | ~ | ~ | |||||||||
| B (по данным dssp) | 3-6 | 11-14 | 22-27 | 60-65 | 134-136 | 142-144 | 181-186 | 201-203 | 221-225 | 251-254 | 260-262 | 302-304 |
| B (по описанию в pdb) | 3-5 | 12-14 | 22-27 | 60-65 | 134-136 | 142-144 | 181-186 | 201-203 | 221-225 | 251-254 | 260-262 | 302-304 |
| Различия | ~ | ~ |
Из таблиц видно, что координаты всех бета-тяжей по данным файла PDB практически совпадают с координатами, определенными программой DSSP.Только первых 2 тяжа отличаются на 1-2 а.о (это объясняется алгоритмом программы DSSP, который не включает в элементы альфа-спирали крайние аминокислотные остатки. Этому правилу, однако, есть и исключения). Координаты альфа-спиралей по данным файла PDB лишь местами совпадают с координатами, определенными программой DSSP. Таким образом, можно сказать, что с поиском бета-тяжей программа справилась отлично, а вот для альфа-спиралей необходимо усовершенствовать поиск. Так же удивляет то, что и DSSP и в pdb-файле для одна альфа-спираль с другой (15-16 для А и 13-14 для B) идут практически одновременно - но считаются разными.
Рассмотрим колонку PHI выдачи DSSP, отметим остатки, имеющие положительное значение угла φ :
Цепь A: метионин 1 аргинин 10 глицин 21 глицин 58 аспартат 71 глицин 82 аспарагин 104 глицин 132 глицин 140 глицин 153 аргинин 157 лизин 175 глицин 190 глицин 199 глицин 218 глицин 226 пролин 233 глицин 248 аспартат 263 глицин 272 глицин 325 глицин 334 аспартат 349 фенилаланин 350 глицин 392 лейцин 394 Цепь B: метионин 1 аргинин 10 глицин 21 глицин 58 валин 86 аспарагин 104 глицин 132 глицин 140 глицин 153 аргинин 157 аргинин 176 глицин 190 серин 196 глицин 199 глицин 218 глицин 226 пролин 233 аспартат 247 глицин 248 аспартат 263 аргинин 264 аспарагин 273 фенилаланин 275 глицин 325 глицин 334 аспартат 349 фенилаланин 350 глицин 392 лейцин 394Следует заметить, что среди остатков, имеющих положительное значение угла phi, оказались не только глицины, но и аспарагин и лейцин, фенилаланин и др, имеющие достаточно большие по размеру радикалы, мешающие вращению вокруг N-Ca связи (что и определяет угол phi). Так же видим, что остатки, имеющие положительное значение угла, распологаются в цепях одинаково.
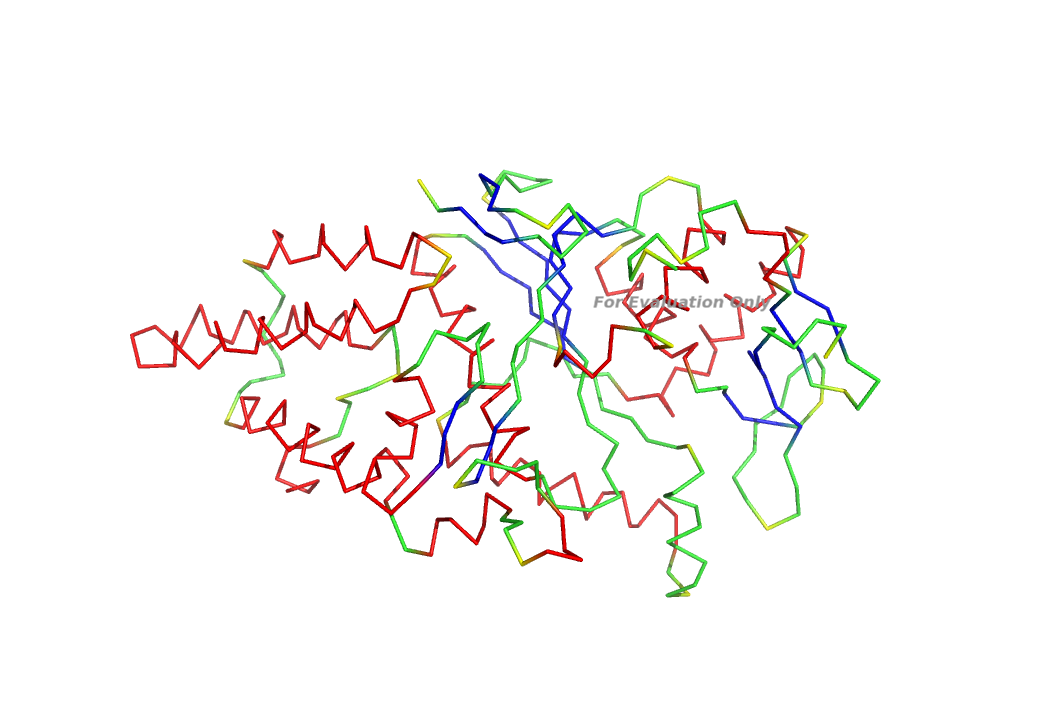
Создадим в PyMOL изображение, представляющее остовную модель (ribbon) цепи A белка 1LI5, на котором различными цветами выделены альфа-спирали (красного цвета), бета-тяжи (синего цвета), остатки с положительным phi (желтого цвета):

С помощью программы HBPlus определим водородные связи в белке 1LI5.
запустим программу с помощью команды: hbplus 1LI5.pdb Получим файл 1LI5.hb2Переведем полученные данные в файл Exel. Приведем примеры различных водородных связей, используя эти данные:
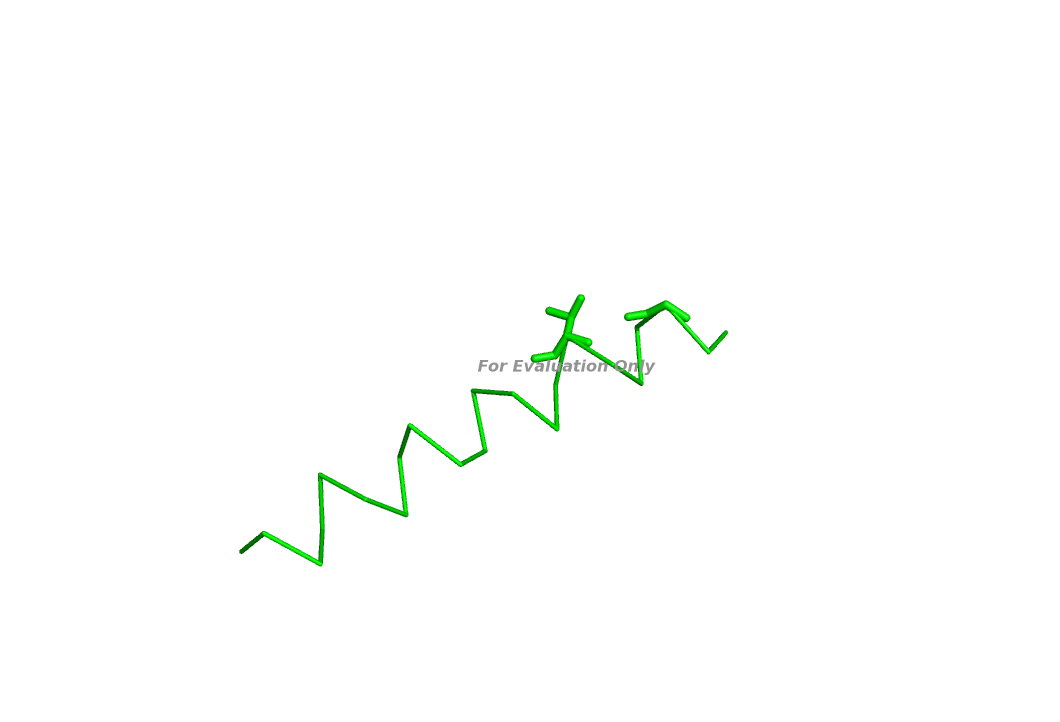
а) Водородные связи, участвующие в стабилизации вторичной структуры:
A0043 THR N A0039 GLY O A0043 THR OG1 A0039 GLY O
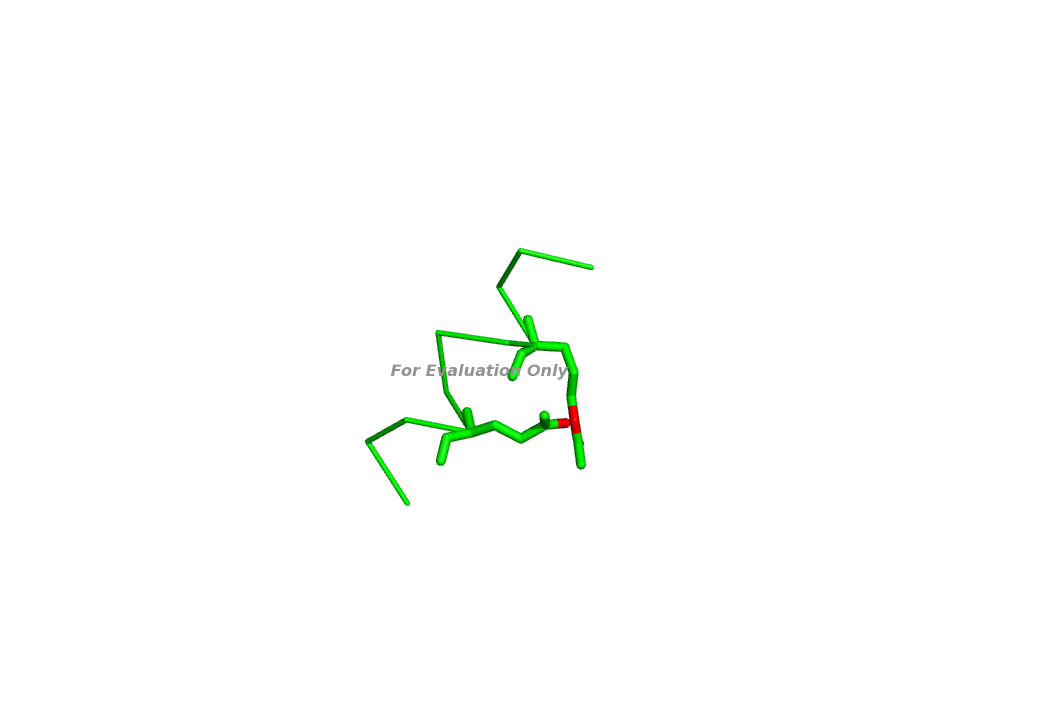
б) между боковыми цепями аминокислотных остатков:
A0313 ARG NE A0317 GLU OE2 Взаимодействующие атомы выделены красным
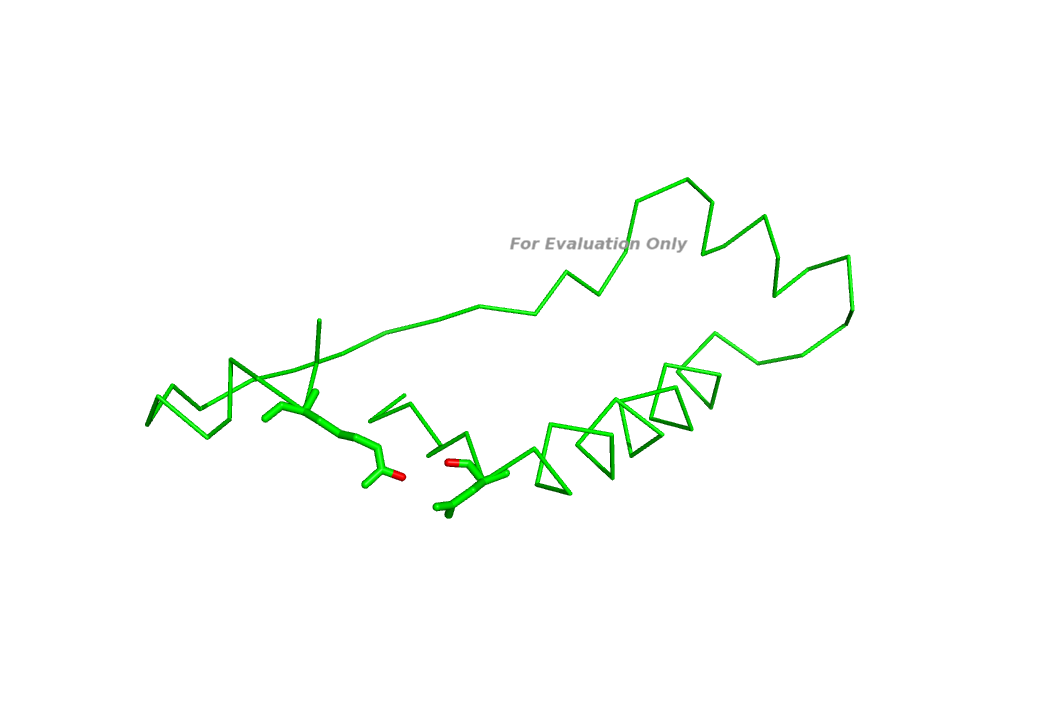
в) между боковой цепью одного остатка и остовным атомом другого:
A0052 ARG NH2 A0104 ASN O Взаимодействующие атомы выделены красным
В данной структуре нет малых органических молекул (лигандов).
Приведем пример взаимодействия одного остатка с двумя:
A0143 MET N A0135 TYR O A0176 ARG N A0135 TYR OH Соответсвующие взаимодействующие атомы выделены красным и синем.
Примечание. Возможно при выполнении этих упражнений пользоваться STRIDE вместо DSSP. STRIDE уже установлен на kodomo, при первом запуске он просит прочитать правила его использования и согласиться с ними. После этого его можно запускать командой: stride 1LI5.pdb > 1LI5.stride