Занятие 6. Изучение работы методов контроля температуры в GROMACS.
Цель - изучение реализации контроля температуры в молекулярной динамике на примере GROMACS.
Объект исследования - это одна молекула этана.
- Подготовка файлов координат и топологии.
На основе gro файла с 38 молекулами этана с прошлого занятия был создан индекс файл,
в котором будет группа из одной молекулы этана.
make_ndx -f box_38.gro -o 1.ndx
(После запуска команды появляется приглашение к вводу. Для ознакомления с помощью надо нажать кнопку "h" + enter.
Далее был выбран остаток номер 1 -> enter -> появилась новая группа).
Для создания gro файла с одной молекулой и задания ячейки была введена следующая команда:
editconf -f box_38.gro -o et1.gro -n 1.ndx
#зададим ячейку и расположим молекулу по центру ячейку
editconf -f et1.gro -o et.gro -d 2 -c
(При запуске ediconf надо выбрать номер, соответствующий группе из одной молекулы).
В результате был получен файл et.gro. Также я исправила файл топологии et.top из прошлого задания:
в разделе [ molecules ] надо было просто изменить количество молекул этана (вместо 38-ми 1).
- Разные параметры контроля температуры.
be.mdp - метод Берендсена для контроля температуры.
vr.mdp - метод "Velocity rescale" для контроля температуры.
nh.mdp - метод Нуза-Хувера для контроля температуры.
an.mdp - метод Андерсена для контроля температуры.
sd.mdp - метод стохастической молекулярной динамики.
Скрипт по работе с 5ю системами одновременно: смотреть.
- Разбор скрипта.
Входные файлы были построены для молекулярно-динамического движка mdrun с помощью grompp:
grompp -f ${i}.mdp -c et.gro -p et.top -o et_${i}.tpr
# где i: be,vr,nh,an,sd - см. выше список mdp файлов
Для каждого из 5 полученных tpr файлов запускается mdrun.
mdrun -deffnm et_${i} -v -nt 1
Для визуального анализа необходимо конвертировать каждую из 5 систем в pdb:
trjconv -f et_${i}.trr -s et_${i}.tpr -o et_${i}.pdb
Полученные pdb-файлы для дальнейшего просмотра в PyMOL:
- et_be.pdb
- et_an.pdb
- et_nh.pdb
- et_sd.pdb
- et_vr.pdb
Наблюдения, сделанные на основе визуального анализа:
1) В методе Берендсена в начале видны небольшие колебания и вращение молекулы, но достаточно быстро скорость вращения
начинает увеличиваться, а амплитуда колебаний затихать. Такая система выглядит довольно неправдоподобно.
2) В методе Андерсена видны небольшие колебания по длинам связей и валентным углам, однако молекула при этом не вращается.
Возможно, такое состояние характерно для достаточно низких температур, когда молекулы образуют кристалл.
3) В методе Нуза-Хувера длина связи С-С в молекуле этана практически не изменяется (по ркайней мере, визуально), можно наблюдать лишь вращение.
Хотя в основном молекула находится в более выгодной заторможенной конформации, иногда можно заметить заслонённую.
4) В методе стохастической молекулярной динамики трудно оценить по какому принципу молекула движется, т.к. она очень быстро перемещается, меняя свое направление
то ли случайно, то ли при столкновении с другими молекулами, которые просто здесь не представлены.
5) Метод "Velocity rescale" и метод Нуза-Хувера весьма похожи друг на друга, разница лишь в том, что в первом методе вращение по связи меньше, а
амплитуда колебаний больше. Невыгодные конформации, близкие к заслонённой, также видны достаточно редко.
Для сравнения потенциальной энергии связи и кинетической энергии для каждой из 5 систем:
g_energy -f et_${i}.edr -o et_${i}_en.xvg
На этом месте работа скрипта закончилась.
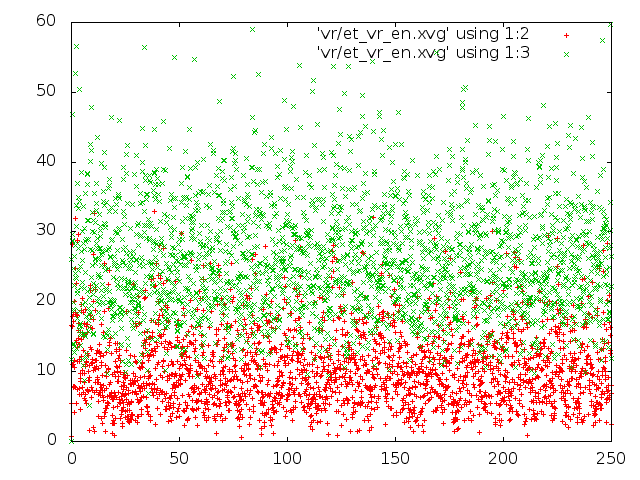
- Графики изменения энергий.
С помощью Gnuplot были построены графики изменения энергий (рекомендуемый вид - dot-plot). Зеленым цветом выделена кинетическая энергия, и зеленым
потенциальная.
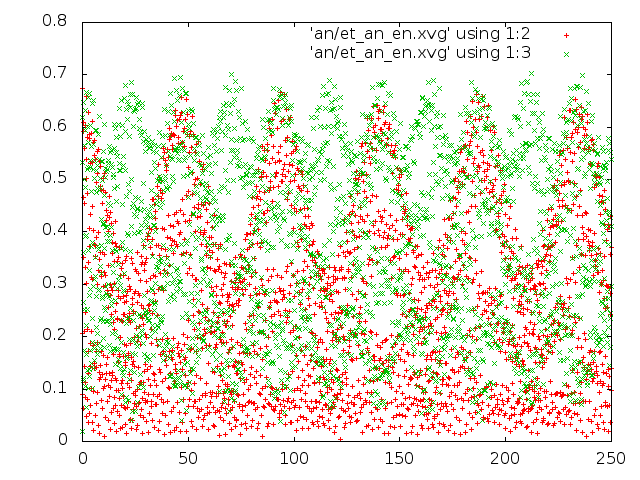
1) В методе Андерсена значения потенциальной и кинетической энергий примерно одинаковые и очень маленькие. Поэтому не верится, что такой термостат
хорошо подходит для передачи молекуле необходимой энергии для того, чтобы она попала в глобальный минимум;
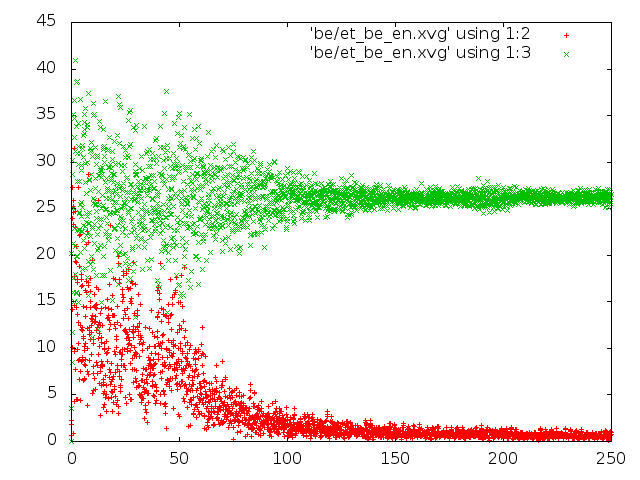
2) В методе Берендсена потенциальная энергия довольно быстро уменьшается, а вот кинетическая выходит на плато. Значения кинетической энергии выше,
чем в методе Андерсена, но все равно еще достаточно невелики;
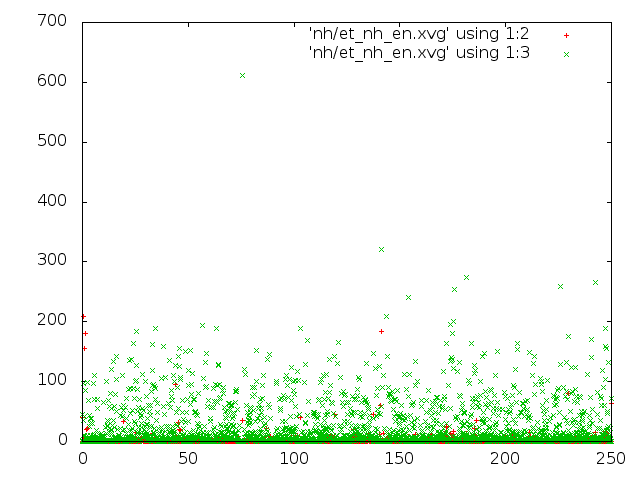
3) В методе Нуза-Хувера можно увидеть единичные достаточно высокие значения кинетической энергии.
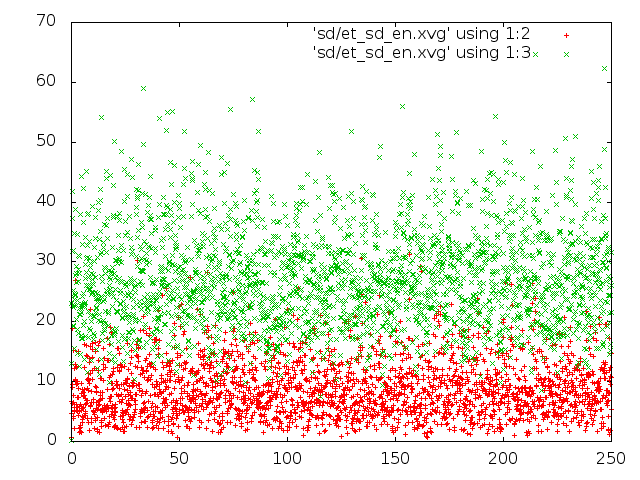
4) В методе стохастической молекулярной динамики видны средние, примерно постоянные, значения кинетической энергии;
5) В методе "Velocity rescale" ситуация сходна с тем, что происходит в методе стохастической молекулярной динамики.
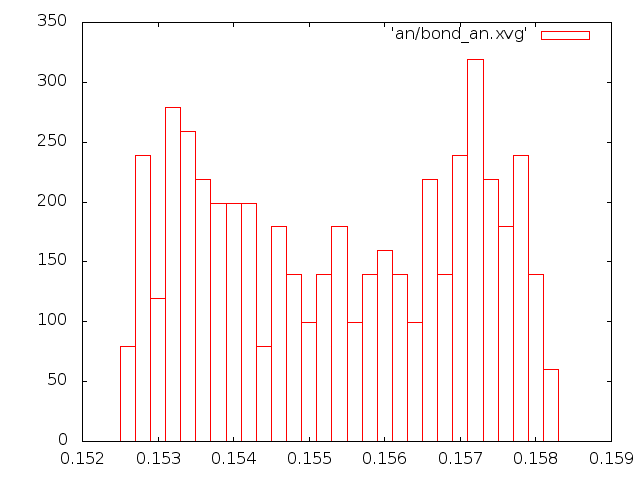
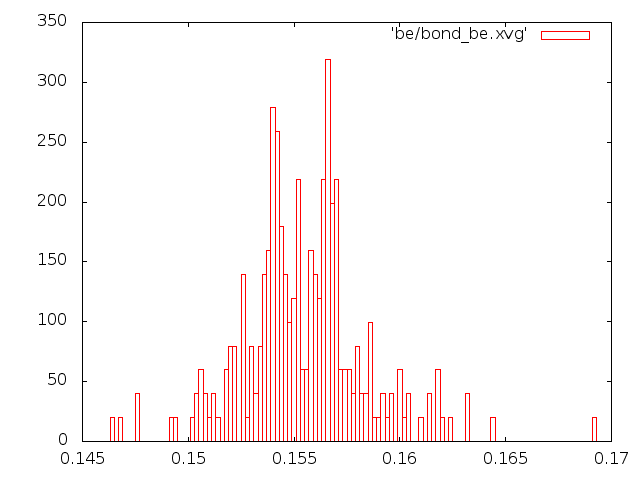
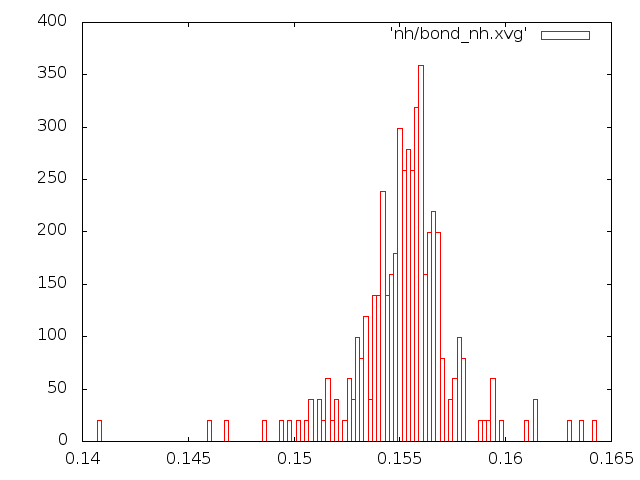
- Графики распределения длин связей.
Для анализа распределения длины связи С-С за время моделирования в текстовом редакторе был создан индекс-файл b.ndx с одной связью.
Его содержимое:
[ b ]
1 2
После этого была запущена утилита по анализу связей g_bond:
g_bond -f et_${i}.trr -s et_${i}.tpr -o bond_${i}.xvg -n b.ndx
Графики распределения длин связей были также построены в Gnuplot:
1) Метод Андерсена
2) Метод Берендсена
3) Метод Нуза-Хувера
4) Метод стохастической молекулярной динамики
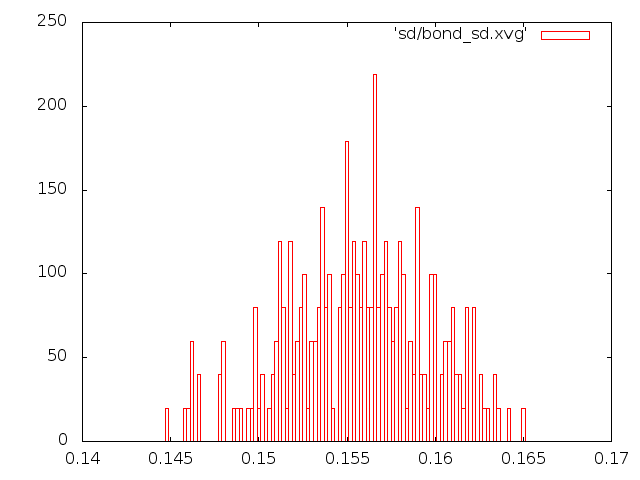
5) Метод "Velocity rescale"
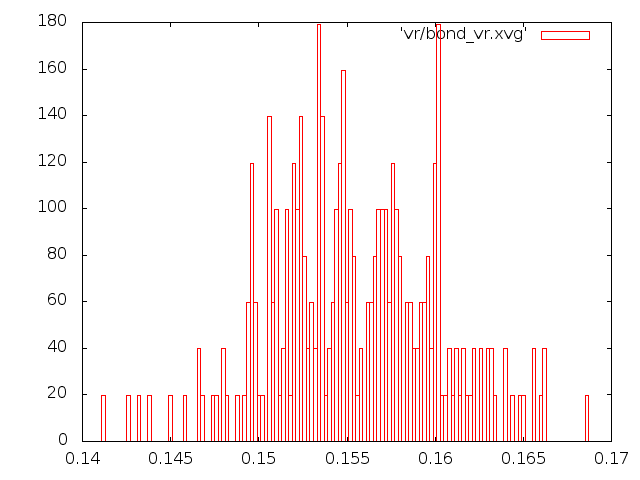
- Распределение Больцмана.
Форма распределения Больцмана:
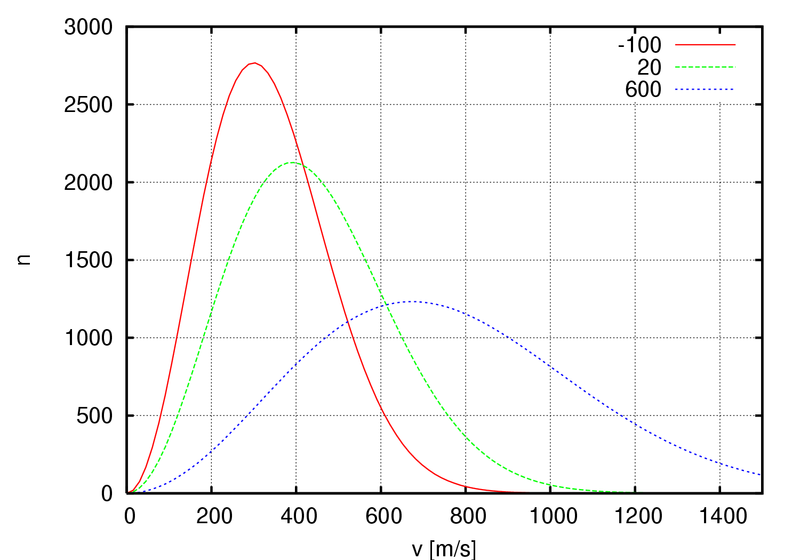
Этому распределению совершенно не соответствуют графики методов Андерсена и Берендсена, а вот с
графиками методов стохастической молекулярной динамики и "Velocity rescale" соответствие неплохое.
Исходя из всех полученных наблюдений, можно сказать, что наиболее вероятны 2 метода: стохастической молекулярной динамики и "Velocity rescale".
Однако в то же время визуальное представление метода стохастической молекулярной динамики в Pymol довольно сложно интерпретировать, по крайней мере,
в данном конкретном случае. Поэтому, по моему мнению, метод, который позволяет наиболее реалистично поддерживать температуру в системе, - "Velocity rescale".
|
|
