Занятие 10. Поверхность, гидрофобное взаимодействие, домены.
-
Для димера пуриновых репрессоров 1VPW в PyMol были созданы изображения:
а) поверхности контакта мономера белка (цепи А) с симметричным мономером
на фоне остовной (ribbon) модели мономера
(контактирующими будем считать атомы обоих мономеров белка, расположенные на расстоянии менее 5 Å)

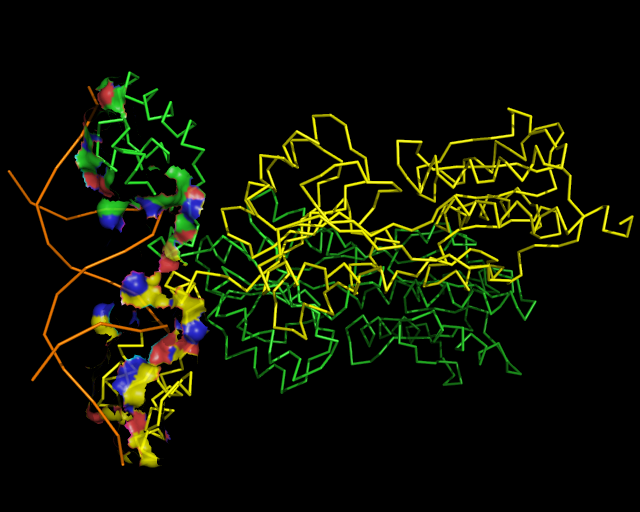
б) поверхности контакта димера белков с двойной спиралью ДНК на фоне
остовной модели части белка, вовлечённой в контакт
(контактирующими будем считать атомы белка и ДНК, находящиеся на расстоянии менее 5 Å)
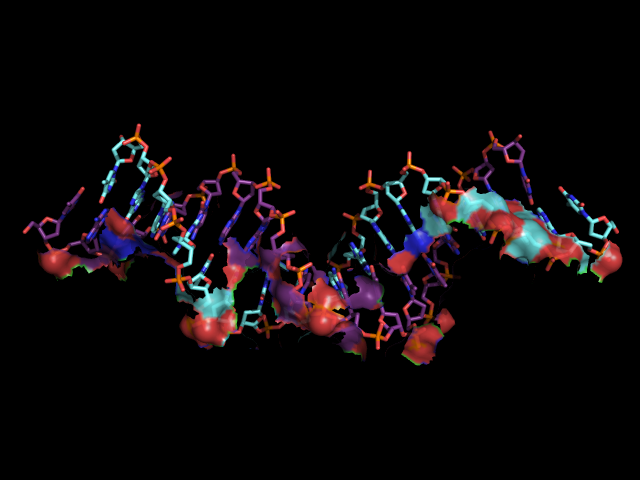
в) поверхности контакта ДНК с димером белков на фоне проволочной (sticks)
модели двойной спирали
(контактирующими будем считать атомы белка и ДНК, находящиеся на расстоянии менее 5 Å)
Для создания изображений была использована запись 1vpw_1.pdb,
содержащая биологическую единицу, в не запись 1vpw.pdb с ассиметрической единицей.
С помощью сервиса
PROTORP была
найдена площадь контакта мономеров белка из упражнения 1
(см. выдача сервиса).
Она равна 2546.57 Å2.
40.62% от нее приходится на гидрофобные взаимодействия (площадью гидрофобных взаимодейтсвий считаем ту долю площади, что приходится
на неполярные атомы).
С помощью сервиса
CluD,
были определены гидрофобные кластеры на интерфейсе мономеров белка 1VPW
(его биологической единица - 1vpw_1.pdb).
Выдача сервера CluD: смотреть.
Не смотря на что в запросе указывалось найти кластеры, состоящие из не менее 10 остатков, CluD выдал также
кластеры размером 3-8 а.о. Они были удалены вручную (см. здесь).
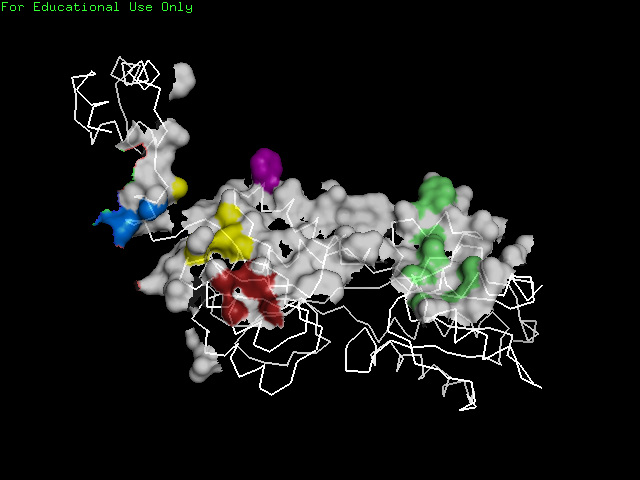
На Python был написан скрипт для получения PyMol скрипта,
который создает то же изображение, что в упр. 1а, но где поверхность, относящаяся к атомам, входящим в найденные
гидрофобные кластеры, выделена различными цветами.
На сервисе pDomains
были определены доменные структуры:
а) цепи A из записи 1KNP.
| Method | Domain Id | Fragment |
|---|
| CATH | 1KNPA1 |
5-244 , 349-424 | | 1KNPA2 |
245-348 | | 1KNPA3 |
425-533 | | | | SCOP | 1KNPA1 |
423-533 | | 1KNPA2 |
5-237 , 354-422 | | 1KNPA3 |
238-353 | | | | pdp | 1KNPA1 |
5-244 , 348-533 | | 1KNPA2 |
245-347 | | | | DHcL | 1KNPA1 |
5-160 | | 1KNPA2 |
161-533 | | | | dp | 1KNPA1 |
5-242 , 350-422 | | 1KNPA2 |
423-533 | | 1KNPA3 |
243-349 | | | | DDomain | 1KNPA1 |
5-533 | | | | NCBI | 1KNPA1 |
1-238 , 355-540 | | 1KNPA2 |
239-354 | | | | PUU | 1KNPA1 |
1-39 , 147-239 , 346-374 , 397-529 | | 1KNPA2 |
40-49 , 86-114 , 126-146 | | 1KNPA3 |
50-85 , 115-125 , 375-396 | | 1KNPA4 |
240-313 , 334-345 |
Как видно из таблицы, метод DDomain единственный определил, что цепь А состоит из одного домена, что довольно странно. Остальные методы
(кроме DHcL) опреледяют домен, расположенный приблизительно на 250-350 а.о. Метод DHcL включает этот участок в единый
домен 161-533. Метод PUU выдал довольно сложную доменню структуру, состоящую из 4 доменов, которые разбиты на
небольшие части и еще "перемешаны" между собой. Поэтому мне кажется, что структура, определенная данным методом,
не совсем корректна. 3 метода определили в структуре два домена, и оставшиеся 3 три домена. Методы NCBI и pdb показали очень схожие
доменные структуры:
То же самое можно сказать и о методах dp и CATH и БД SCOP, которые выделяют отдельно еще один домен:
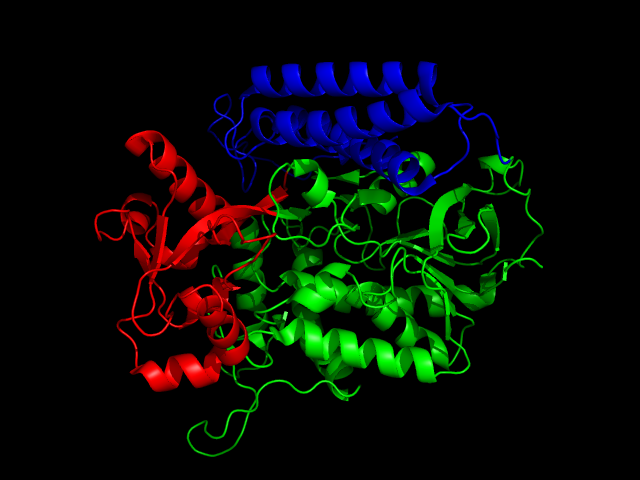
б) цепи А из записи 1VPW.
| Method | Domain Id | Fragment |
|---|
| CATH | 1VPWA1 |
2-58 | | 1VPWA2 |
59-160 , 291-322 | | 1VPWA3 |
161-290 , 323-339 | | | | SCOP | 1VPWA1 |
2-57 | | 1VPWA2 |
58-339 | | | | pdp | 1VPWA1 |
2-47 | | 1VPWA2 |
48-141 | | 1VPWA3 |
142-339 | | | | DHcL | 1VPWA1 |
2-117 | | 1VPWA2 |
118-339 | | | | dp | 1VPWA1 |
2-57 | | 1VPWA2 |
58-155 | | 1VPWA3 |
156-339 | | | | DDomain | 1VPWA1 |
2-339 | | | | NCBI | 1VPWA1 |
1-45 | | 1VPWA2 |
58-160 , 293-340 | | 1VPWA3 |
161-292 | | | | PUU | 1VPWA1 |
1-45 | | 1VPWA2 |
57-157 , 292-318 | | 1VPWA3 |
158-291 , 319-338 |
И снова метод DDomain единственный определил 1 домен в цепи А, что уже говорит о сомнительности в использовании данного метода.
Метод DHcL и БД SCOP определили двудоменную структуру, причем C-концевой домен от DHcL приблизительно на 60 а.о.
меньше C-концевого домена от SCOP (N-концевой домен, т.е. ДНК-связывающий, соответственно больше).
Т.к. большинство методов выдают
N-концевые домены размера, сходного с размером N-концевого домена от SCOP, далее метод DHcL рассматриваться не будет.
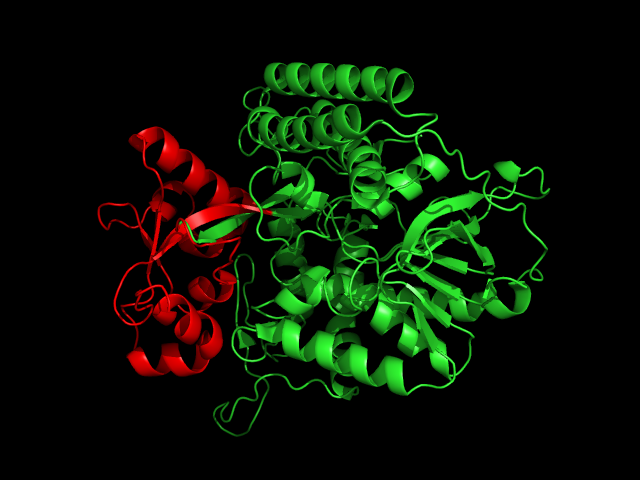
Все остальные методы определили три домена. Методы pdp и dp выявили их, как три идущих подряд домена:
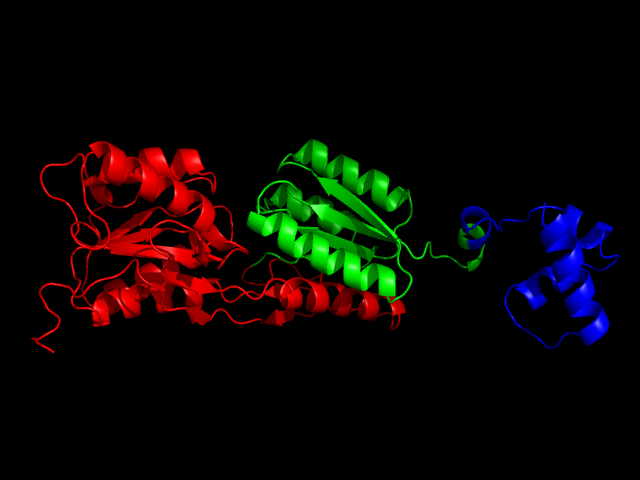
Синим цветом выделен N-концевой домен, а красным C-концевой.
Метод NCBI определяет доменную структуру следующим образом:
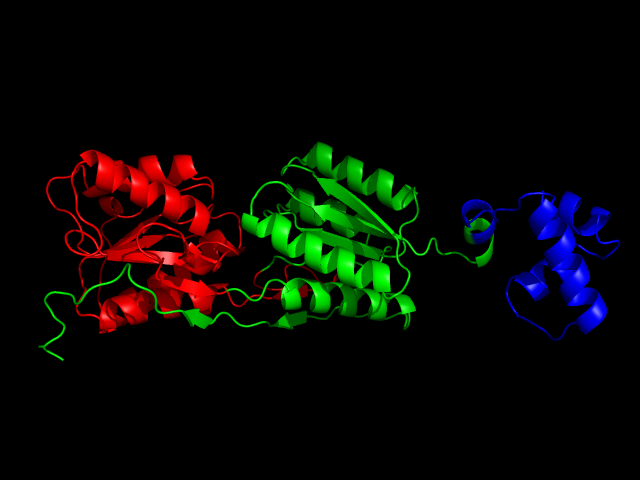
Видно, что C-хвост, выделенный зеленым цветом, структурно находится в другом домене. Это вычисляют методы PUU и CATH:
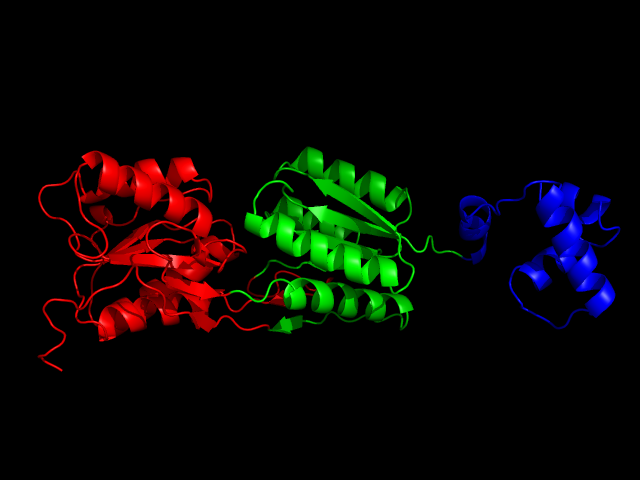
Итого, можно предположить, что в данном случае SCOP показал функциональные домены (ДНК- и эффектор-связывающие),
а другие методы (кроме DHcL и DDomain) структурные.
|
|









