Занятие 9. Качество структуры: PROCHECK, EDS, PDB_REDO.
- Анализ выдачи программы PROCHECK.
На сайте БД PDBSum была найдена
выдача
программы PROCHECK для записи PDB 1KNP.
Карта Рамачандрана для данной структуры:
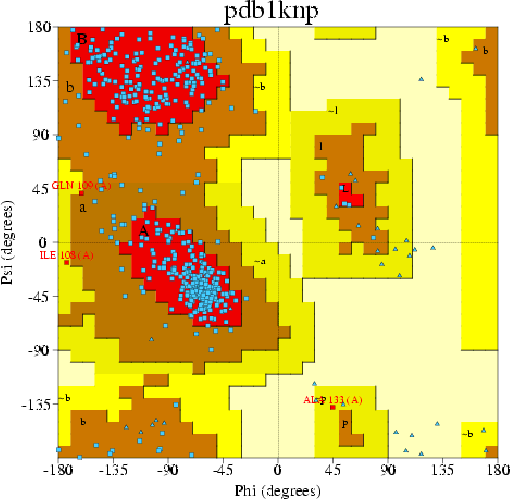
Модель считается хорошей, если более 90% остатков (отличных от Gly и Pro) попали в предпочитаемые области на карте.
Здесь этот процент равен 83.9%. Следовательно, нашу модель можно считать достаточно неплохой, однако далеко не самой оптимальной.
Замечу, что запрещенные области не содержат остатки, отличные от глицина и пролина, и что всего 0.6% остатков (Ile 108, Gln 109, Ala 133) содержатся в
допустимой области (соответственно, 15,5% находятся в разрешенной области).
- Работа на сервере EDS. Раздел Significant regions.
На сервере EDS была открыта страница
структуры 1KNP.
По данным, содержащимся в разделе Significant regions, 33 остатка имеют большой Z-score по RSR.
Для дальнейшего анализа был выбран аргинин 254, чей RSR составил 0.391, коэффициент корреляции для электронной плотности
("real-space correlation coefficient")- 0.668, Z-score пространственного R-фактора - 2.796895.
В PyMol было создано изображение его модели
с электронной плотностью уровня 0.5 (белый цвет) и уровня 1.0 (желтый цвет):
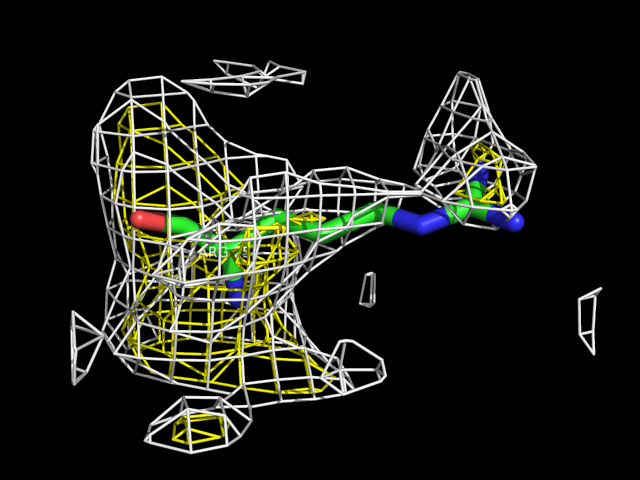
Как видно из картинки, электронная плотность обоих уровней не охватывает модель аргинина полностью,
NH2 и особенно NE в нее не вписаны, качество структуры для данного остатка низкое. Поэтому вполне закономерны плохие значения
таких характеристик, как Z-score и RSR.
- Работа на сервере EDS. Раздел PDB_REDO.
R-values etc.
| From PDB header | Calculated from data | After conservative optimisation | After full optimisation |
|---|
| R | 0.2300 | 0.2360 | 0.2215 | 0.2131 |
| R-free | 0.2810 | 0.2801 | 0.2589 | 0.2494 |
| σR-free | | 0.0056 | 0.0052 | 0.0050 |
| R-free Z-score | | 6.52 | 7.37 | 7.30 |
В первой колонке содержатся значения параметров, взятых из самой записи PDB, во второй - значения, посчитанные по имеющимся
данным. Видно, что они очень близки между собой. В последней колонке находятся значения параметров после полной оптимизации.
В этом случае, значения R-фактора и R-free уменьшились, иначе говоря улучшились. В то же время уменьшилась разница между ними
(0.0363 против 0.0510 ранее), что также говорит об улучшении качества структуры.
Однако значение Z-score увеличилось, что, наоборот свидетельствует об ухудшении оптимизации (положительное значение Z-score
говорит либо о заранее предвзятом выборе множества для расчета R-free,
либо о необходимости дальнейшей оптимизации (в идеале Z-score должен быть равен нулю)).
WHAT_CHECK validation
| Original PDB entry | Conservatively optimised | Fully optimised |
|---|
| 1st generation packing quality1 | -0.938 | -0.390 | -0.303 |
| 2nd generation packing quality1 | -1.744 | -0.953 | -0.798 |
| Ramachandran plot appearance1 | -3.858 | -1.955 | -1.670 |
| Chi-1/Chi-2 rotamer normality1 | -3.507 | -0.808 | 0.159 |
| Backbone conformation1 | -1.625 | -0.708 | -0.671 |
| Bond length RMS Z-score2 | 1.036 | 0.323 | 0.316 |
| Bond angle RMS Z-score2 | 1.287 | 0.596 | 0.589 |
| Total number of bumps3 | 302 | 52 | 52 |
| Unsatisfied H-bond donors/acceptors3 | 74 | 51 | 50 |
1 Чем выше, тем лучше
2 Должно быть меньше 1.000
3 Чем меньше, тем лучше - (*) Скачайте оптимизированную структуру и попробуйте выяснить, чем
она отличается от исходной. В частности, стал ли остаток, выбранный вами
в упр. 2, лучше вписываться в электронную плотность?
-
Выдача WHAT_CHECK для структуры из PDB.
В данной выдаче содержится множество различной информации от карты Рамачандрана и вторичной структуры до неправильной
номенклатуры атомов.
Здесь также можно узнать о необычных конформациях остатков, о проблемах, связанных с В-фактором, о пропущенных атомах
или даже о молекулах воды без водородной связи.
|
|
