1. EnsEMBL
Своё знакомство с данным браузером я начал с сервиса BLAST/BLAT.
В качестве объекта для работы был выбран самый длинный кодирующий участок 1450760..1451035 моего гена HLA_E (из первого занятия блока). Его последовательность была помещена в специальное окошко интерфеса
BLAST/BLAT, достаточно удобного и похожего на интерфейс BLAST'а на NCBI. Был осуществлен поиск по кДНК в человеческом геноме.
Вообще говоря, на странице сервиса BLAST/BLAT помимо различных способов задания искомых последовательностей можно выбрать
вид, по геному которого будет осуществляться поиск, причем их далеко не очень много (около 60ти), а также программы поиска последовательностей (BLASTN, TBLASTX и пр.) и базу данных: нуклеотидную (кДНК, РНК и пр.) или белковую.
Кроме того, можно настроить чувствительность поиска в разделе Search sensitivity.
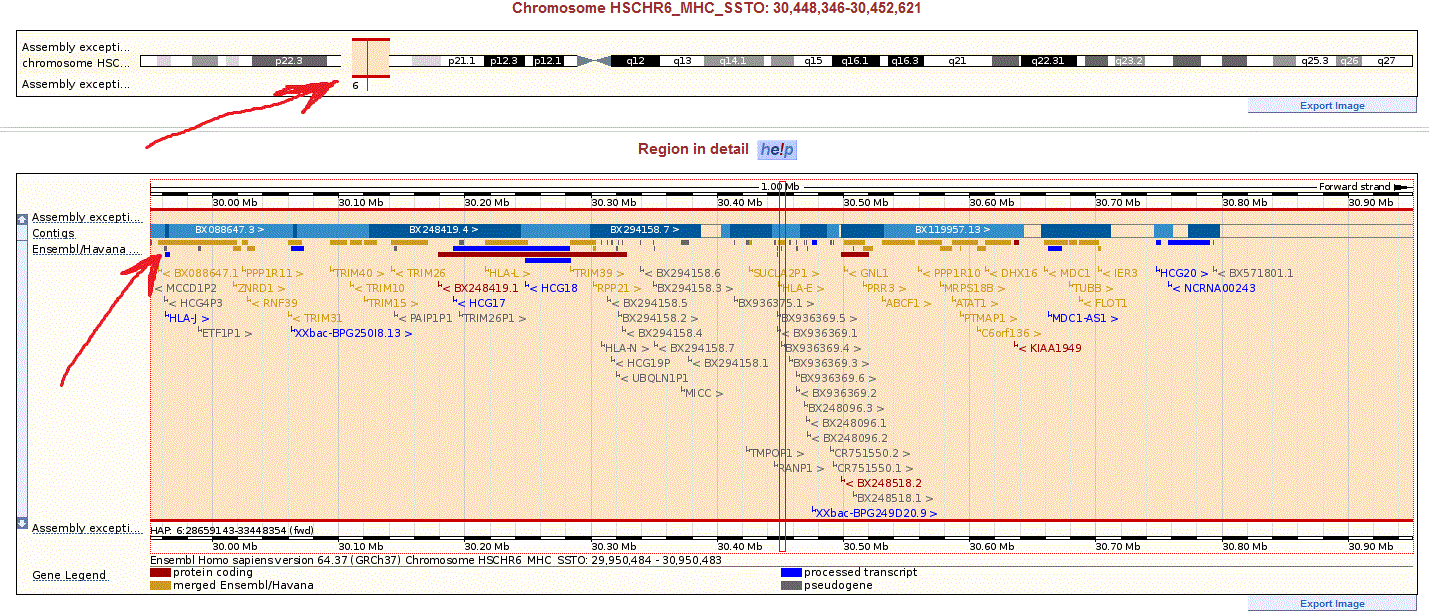
В выдаче наглядно продемонстрирована локализация данного фрагмента в кариотипе(см. ниже).
Query location : unnamed 3 to 275 (+)
Database location : ENST00000552883 293 to 565 (+)
Genomic location : HSCHR6_MHC_COX 30218875 to 30219147 (+)
Alignment score : 205
E-value : 1.1e-112
Alignment length : 273
Percentage identity: 93.77
Query: 3 CCCCCAAAGACACACGTGACTCACCACCCCATCTCTGACCATGAGGCCACCCTGAGGTGC 62
||||||||| |||| ||||| || ||||||||||||||||||||||||||||||||||||
Sbjct: 293 CCCCCAAAGGCACATGTGACCCAGCACCCCATCTCTGACCATGAGGCCACCCTGAGGTGC 352
Query: 63 TGGGCCCTGGGCTTCTACCCTGCGGAGATCACACTGACCTGGCAGCAGGATGGGGAGGGC 122
|||||||||||| ||||||||||||||||||||||||||||||||||||||||||||| |
Sbjct: 353 TGGGCCCTGGGCCTCTACCCTGCGGAGATCACACTGACCTGGCAGCAGGATGGGGAGGAC 412
Query: 123 CATACCCAGGACACGGAGCTCGTGGAGACCAGGCCTGCAGGGGATGGAACCTTCCAGAAG 182
|| ||||||||||||||||| ||||||||||||||||||||||| |||||||||||||||
Sbjct: 413 CAGACCCAGGACACGGAGCTTGTGGAGACCAGGCCTGCAGGGGACGGAACCTTCCAGAAG 472
Query: 183 TGGGCAGCTGTGGTGGTGCCTTCTGGAGAGGAGCAGAGATACACGTGCCATGTGCAGCAT 242
|||| ||||| ||||||||||| ||||||||||||||||||| ||||||||||||||||
Sbjct: 473 TGGGTGGCTGTAGTGGTGCCTTCCGGAGAGGAGCAGAGATACATGTGCCATGTGCAGCAT 532
Query: 243 GAGGGGCTACCCGAGCCCGTCACCCTGAGATGG 275
|||||||| || |||||| ||||||||||||||
Sbjct: 533 GAGGGGCTGCCAGAGCCCCTCACCCTGAGATGG 565
В Contig view наглядно представлено расположение заданного фрагмента в системе контигов, т.е. с какими генами он соседствует, это экзон или интрон. Стоит сказать,
что можно, кликнув на участок схемы, увеличить её масштаб и тем самым с большей подробностью рассмотреть структуру генома.
В верху страницы представлена хромосома с выделенным двумя параллельными красными линиями гомологичным фрагментом. Если опустить
глаза ниже, под заголовком Region in detail можно увидеть более подробную структуру хромосомы (а именно, какие
контиги известны), сама хромосома обозначена лентой с перемеживающимися синими и голубыми участками - разными контигами.
Под этой лентой-хромосомой размещены ID генов из Ensembl/Havana в точности там, где и лежат сами гены.
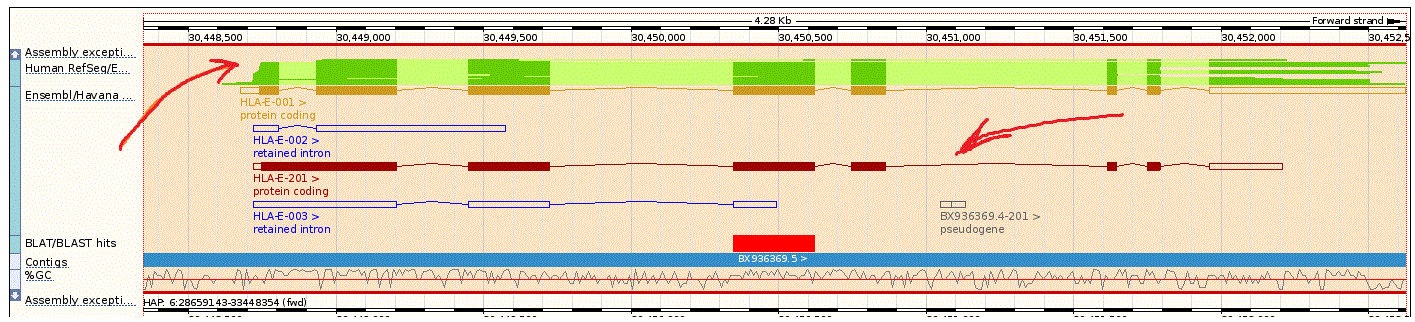
Еще ниже на следующей схеме зеленой лентой обозначена кодирующая ДНК из этого участка из EMBL/NCBI, выровненная с геномом.
А красной линий под ней - тот участок, который кодирует белок.
Нужно отметить, что все гены оснащены ссылками на различные базы данных, в частности, кодирующие белок - на UniProt (необходимо навести курсор для появления всплывающего окна).
Помимо поиска гомологичного участка посредством BLAST/BLAT можно попробовать ввести названия гена - в моем случае HLA_E - в строку быстрого поиска по какому-то из геномов - я выбрал человека.
Выдача будет организована в том же формате, что и была выше, т.е. так же наклядно представлено расположение гена в геноме и системе контигов.
Теперь немного поэкспериментируем.
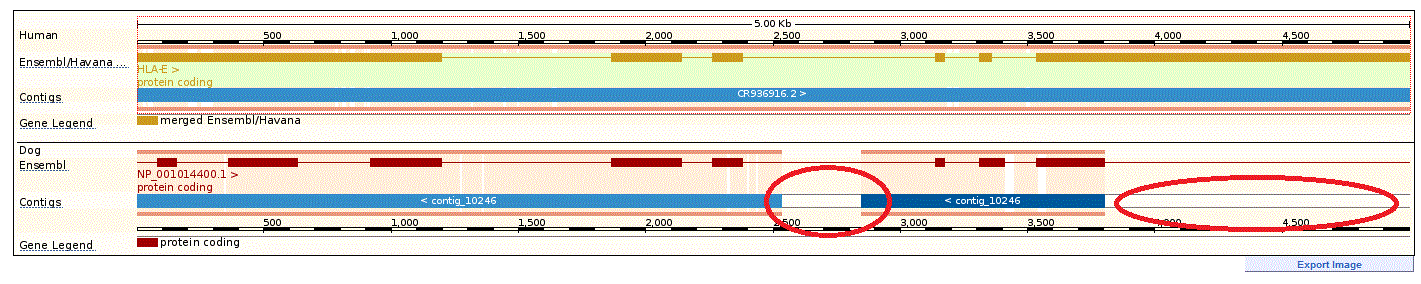
Слева в разделе Comparative Genomics кликнем на Aligments, а далее сравним этот человеческий ген с соответствующим у собаки.
В собачьей последовательности большие гэпы:
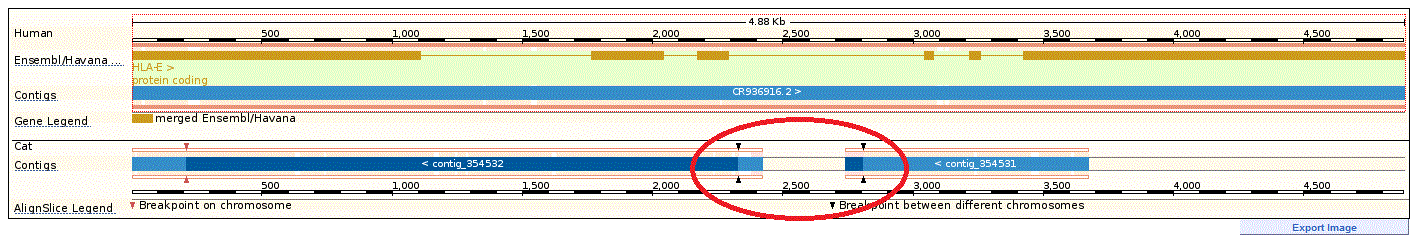
При сравнении с кошкой появилось нечто интересное - инверсия (в кружке):
В разделе Genetic Variation можно посмотреть, как могут варьировать определенные нуклеотиды среди индивидуумов вида. Например, фрагмент моего объкта - человеческого гена HLA_E.
Весьма полезным мне представляется раздел Help & Documentation , где можно найти подробную и понятную информацию
относительно того, как работать с теми или иными сервисами (чем я и воспользовался). Что меня приятно удивило, так это ролики из YouTube в подразделе Tutorials,
где ясно объясняется, как работать с сервисом и как анализировать выдачу, что показалось мне очень привлекательным особенно для написания своей курсовой.
2. Другие геномные браузеры
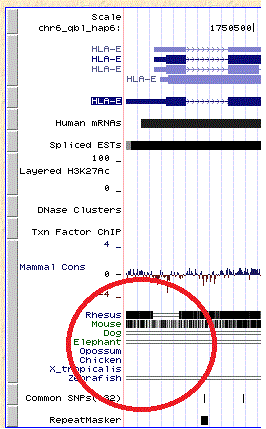
1.UCSC - по сравнению с "Ансамблем" визуализация результатов не такая легкая на восприятие, к тому же нет "Help".
Но сразу представлены данные сравнительной геномики. Так, для меня показалось интересным, что моего гена нет у слона, ноесть у мыши:
2.NCBI - достаточно удобный интерфейс, практически такой же, как и в "Ансамблем", но нет, к сожалению, таких же понятных Tutorials, поэтому нелегко понять.
3.Vega - очень похожа на "Ансамбль", но меньше инструментов для работы. Нужно сказать, что Vega специализируется на хорошо отаннотированных последовательностях прочитанных геномов позвоночных. Так, практически полностью прочитаны геномы человека, мыши и рыбы-зебры.