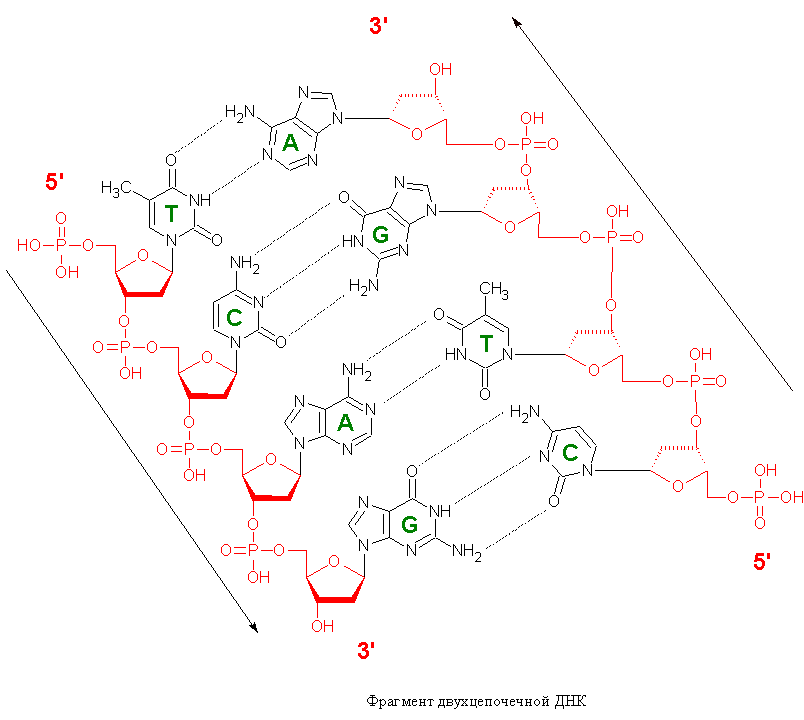
| A-форма | B-форма | *Z-форма | |
Тип спирали (правая или левая) |
Правая |
Правая |
Левая |
Шаг спирали (A) |
28.03 |
33.75 |
43.5 |
Число оснований на виток |
11 |
10 |
12 |
Ширина большой бороздки |
16.81(от фосфата аденина 38) |
17.21 (от фосфата гуанина 25) |
9.86 (от фосфата цитозина 10) |
Ширина малой бороздки |
7.98 (от фосфата аденина 6) |
11.9 (от фосфата цитозина 28) |
18.30 (от цитозина 14) |
| alpha | beta | gamma | delta | epsilon | zeta | chi | |
| А-ДНК | -51.70 | 174.79 | 41.70 | 79.10 | -147.83 | -75.09 | -157.23 |
| В-ДНК | -29.91 | 136.33 | 31.16 | 143.34 | -140.78 | -160.49 | -98.03 |

[1] 5'-GGCGCGUUAACAAAGCGGUUAUGUAGCGGAUUGCAAAUCCGUCUAGUCCGGUUCGACUCCGGAACGCGCCUCCA-3' [76],
где 1 и 76 - номера первого и последнего нуклеотида.
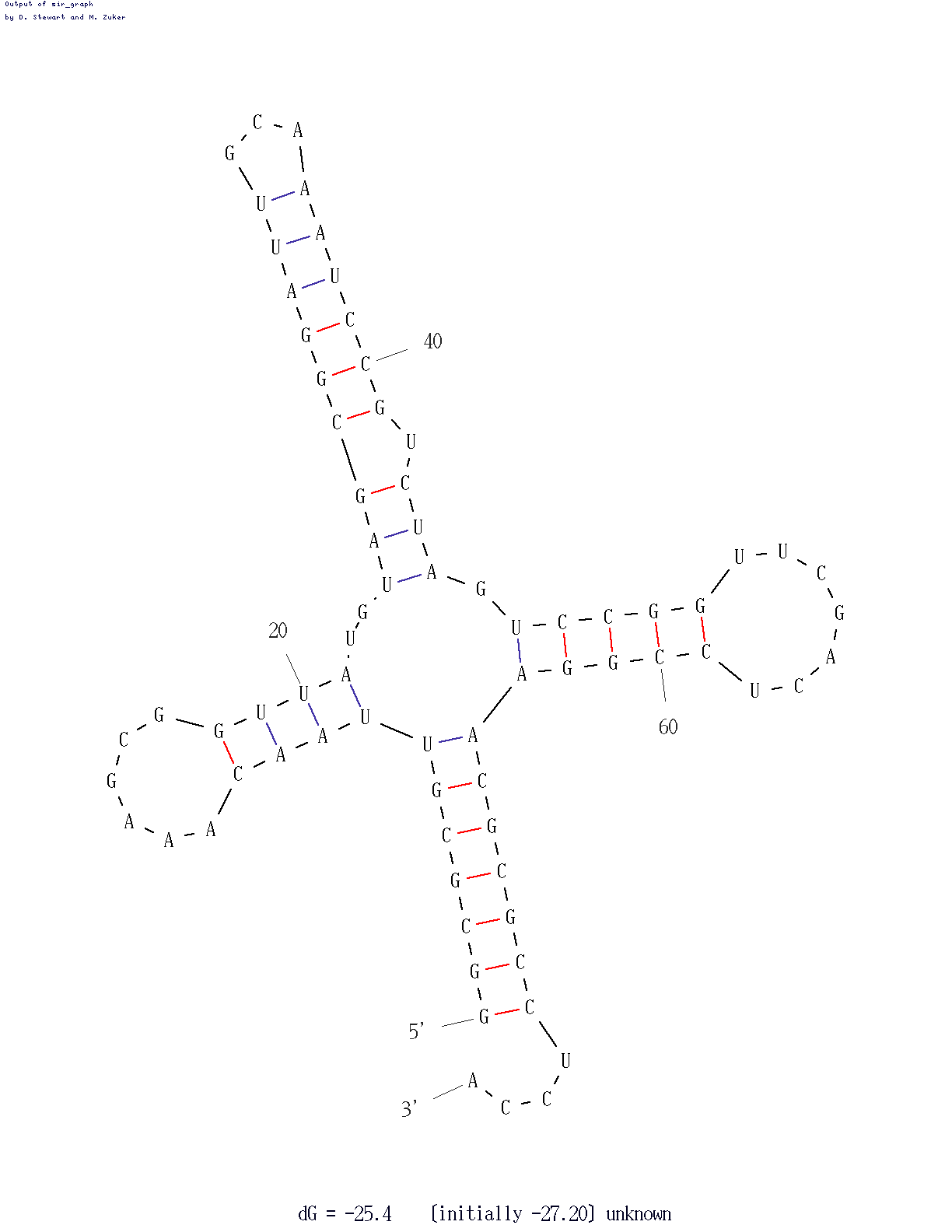
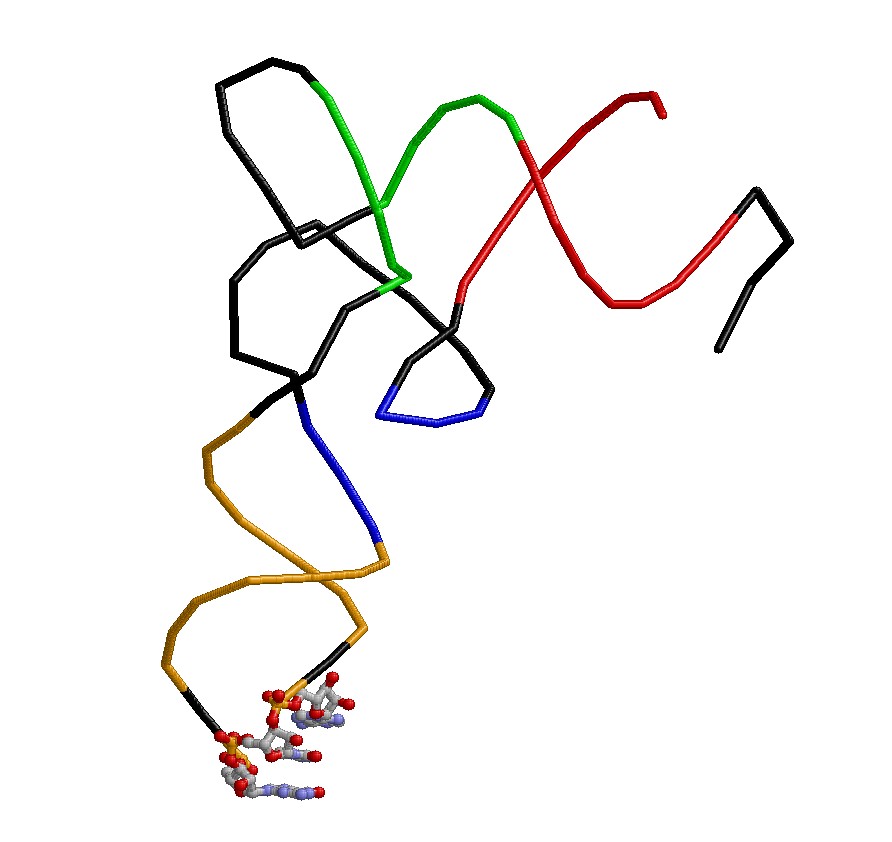
| Рис.1. Вторичная структура Цистеинил-тРНК из Еrichia coli | Скрипт для получения изображения |
|
bakground white restrict RNA wireframe off color black backbone 100 select (1-7, 66-72) and RNA color red select (49-53, 61-65) and RNA color green select (10-12, 23-25) and RNA color blue select (38-44, 26-32) and RNA color orange select (34, 35, 36) and RNA backbone off wireframe 100 cpk 150 color cpk Даный скрипт служит для получения в RasMol изображения остова исследуемой тРНК, где акцепторный стебель выделен красным, Т-стебель - зеленым, D-стебель - синим, антикодоновый - оранжевым. В шарнирной модели представлен антикодон (GCA)к цистеину. |
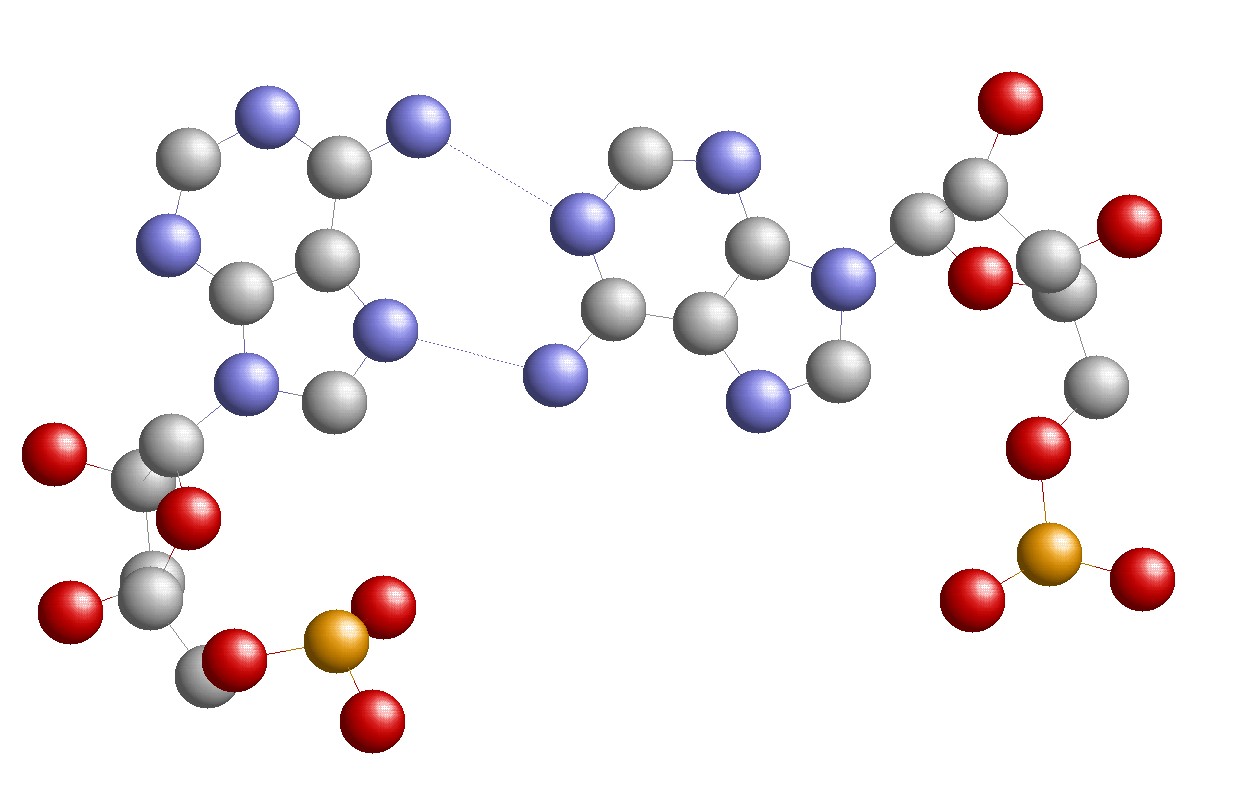
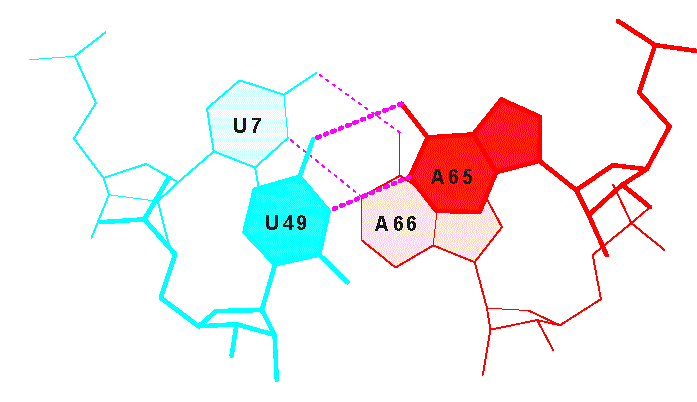
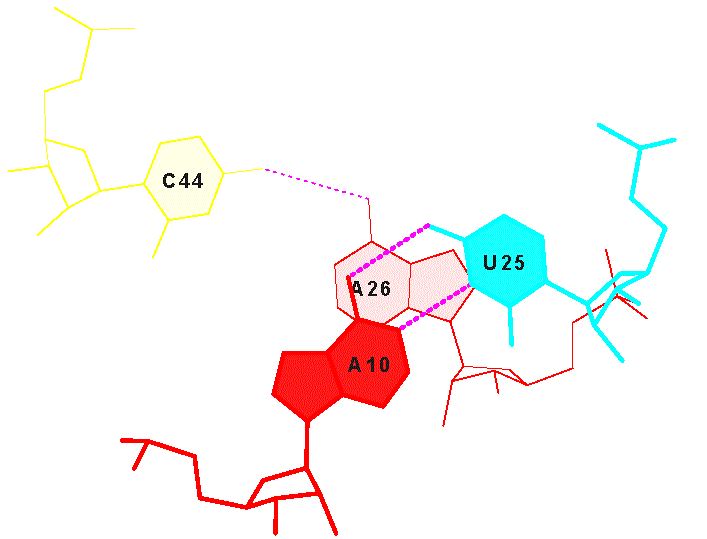
Участок структуры |
Позиции в структуре (по результатам find_pair) |
Результаты предсказания |
Результаты предсказания по алгоритму Зукера |
Акцепторный стебель |
|
|
предсказано 7 пар |
D-стебель |
5' 10-12 3' | Нет предсказаний |
Предсказано 4 пары, включающие 3 верных |
T-стебель |
5' 49-53 3' |
Нет предсказаний |
Предсказаны все 5 пар |
Антикодоновый стебель |
5' 38 - 44 3' |
Предсказано 4 из 7 реальных |
Предсказано 9 пар, включающих в себя 5 верных |
Общее число канонических пар нуклеотидов |
22 |
11 |
20 |