Качество структуры: PROCHECK, EDS, PDB_REDO
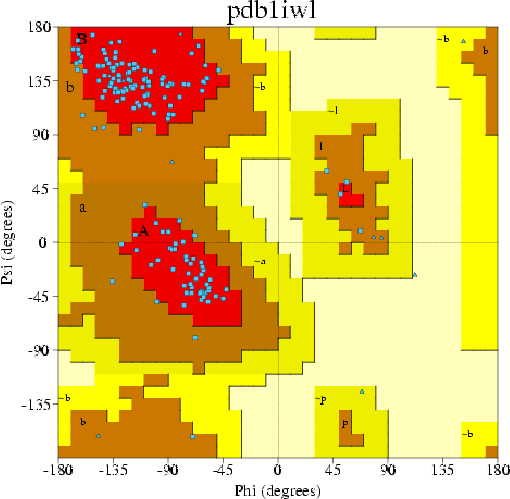
Ramachandran Plot statistics
![]()
No. of
residues %-tage
------ ------
Most favoured regions [A,B,L] 142 91.0%
Additional allowed regions [a,b,l,p] 14 9.0%
Generously allowed regions [~a,~b,~l,~p] 0 0.0%
Disallowed regions [XX] 0 0.0%
---- ------
Non-glycine and non-proline residues 156 100.0%
End-residues (excl. Gly and Pro) 4
Glycine residues 10
Proline residues 7
----
Total number of residues
Нет ни одного остатка в нежелательной области, причем 91% остатков (кроме Gly&Pro) в самой
предпочтительной области. Так что Procheck-проверку структура белка прошла.
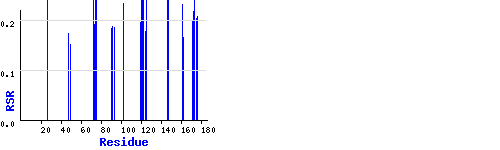
Пространственный R-фактор записи 1IWL
![]()
C помощью сервиса EDS были выявлены остатки с большим z-score по RSR их 26: возьмем
например 26 ASP RSR=0.403,Z-score=5.177778, R-value=0.626.
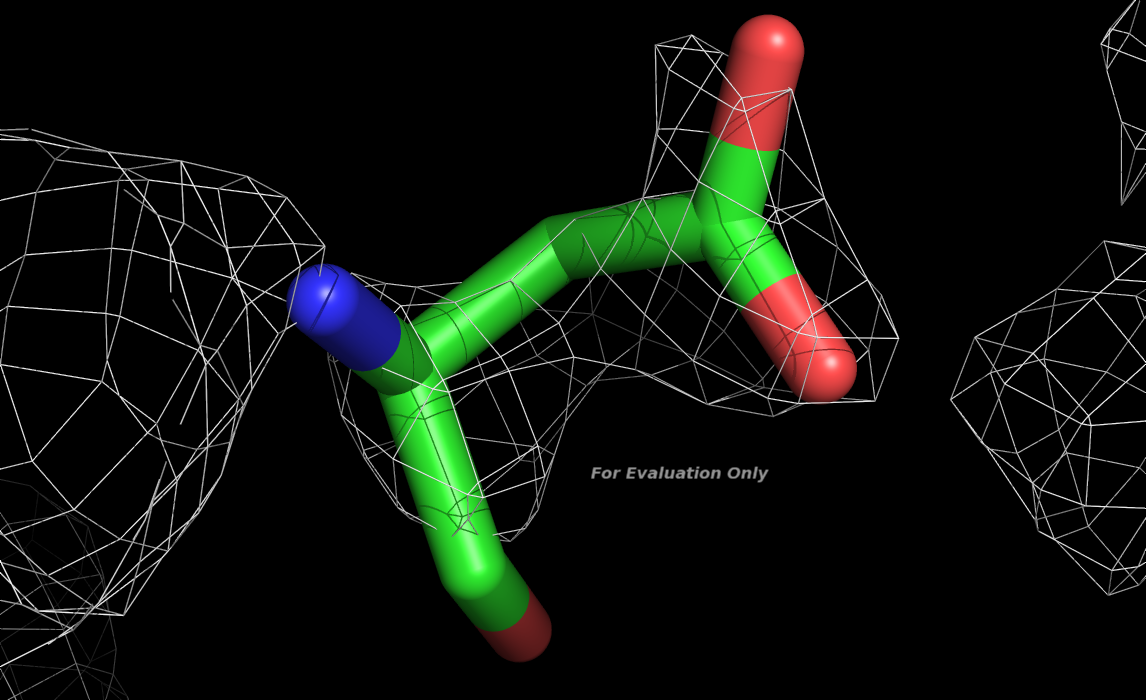
Большое значение RSR означает
необычно маленькое значение электронной плотности для данного остатка, на изображении
показана эл. плотность уровня 1.
![]()
Оптимизация WHAT_CHECK
R-values etc.
From PDB header Calculated from data After conservative optimisation After full optimisation
R 0.2220 0.2177 0.1569 0.1555
R-free 0.2490 0.2439 0.2054 0.2076
(sigma)R-free 0.0049 0.0042 0.0042
R-free Z-score 14.82 5.40 4.40
Все показатели значительно улучшены.
WHAT_CHECK validation
Original PDB entry Conservatively optimised Fully optimised
1st generation packing quality1 0.739 0.683 0.725
2nd generation packing quality1 -1.484 -1.503 -1.258
Ramachandran plot appearance1 -0.601 0.100 0.083
Chi-1/Chi-2 rotamer normality1 -0.862 -0.615 -0.171
Backbone conformation1 -0.134 -0.152 -0.056
Bond length RMS Z-score2 0.288 1.013 0.994
Bond angle RMS Z-score2 0.579 0.876 0.908
Total number of bumps3 31 32 38
Unsatisfied H-bond donors/acceptors3 9 7 8
1 Higher is better
2 Should be lower than 1.000
3 Fewer is better
Улучшились почти все показатели, кроме последних двух (количество "трясок", невзаимодействующие потенциальные доноры или акцепторы водородных связей).
©Мараховская Александра,2010


