Исследование структуры тРНК
Краткое описание структуры в файле 1c0a.pdb
В файле приведены координаты атомов следующих молекул:ASPARTYL TRNA - 1 молекула;
ASPARTYL TRNA SYNTHETASE - 1 молекула.
Молекулы получены из организма кишечной палочки - ESCHERICHIA COLI.
Для исследования была выбрана цепь B, представляющая аспартил-тРНК со следующей последовательностью:
[601] 5' - GGAGCGGUAG UUCAGUCGGU UAGAAUACCU GCCUGUCACG CAGGGGGUCG CGGGUUCGAG UCCCGUCCGU UCCGCCA - 3' [676],
где 601 и 676 - номера первого и последнего нуклеотида.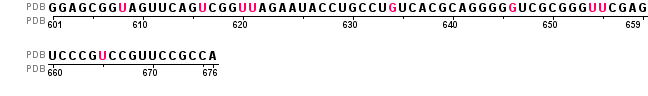
В последовательности на 3'-конце имеется триплет CCA, к которому присоединяется аминокислота. В PDB-файле приведены координаты его атомов.
Исследование вторичной структуры
С помощью программы find_pair пакета 3DNA были определены возможные водородные связи между азотистыми основаниями. (1C0A.out)
Strand I Strand II Helix
1 (0.016) B:.601_:[..G]G-----C[..C]:.672_:B (0.004) |
2 (0.010) B:.602_:[..G]G-----C[..C]:.671_:B (0.007) |
3 (0.006) B:.603_:[..A]A-----U[..U]:.670_:B (0.008) |
4 (0.011) B:.604_:[..G]G-*---U[..U]:.669_:B (0.006) |
5 (0.008) B:.605_:[..C]C-----G[..G]:.668_:B (0.006) |
6 (0.012) B:.606_:[..G]G-----C[..C]:.667_:B (0.006) |
7 (0.011) B:.607_:[..G]Gx----C[..C]:.666_:B (0.006) |
8 (0.007) B:.649_:[..G]G-*---P[PSU]:.665_:B (0.044) |
9 (0.003) B:.650_:[..C]C-----G[..G]:.664_:B (0.010) |
10 (0.006) B:.651_:[..G]G-----C[..C]:.663_:B (0.002) |
11 (0.009) B:.652_:[..G]G-----C[..C]:.662_:B (0.003) |
12 (0.009) B:.653_:[..G]G----xC[..C]:.661_:B (0.007) |
13 (0.010) B:.654_:[5MU]u-**-xA[..A]:.658_:B (0.006) |
14 (0.045) B:.655_:[PSU]Px**+xG[..G]:.618_:B (0.004) x
15 (0.004) B:.638_:[..C]C-*---C[..C]:.632_:B (0.005) |
16 (0.005) B:.639_:[..G]G-----C[..C]:.631_:B (0.008) |
17 (0.004) B:.640_:[..C]C-----G[..G]:.630_:B (0.009) |
18 (0.005) B:.641_:[..A]A-----U[..U]:.629_:B (0.008) |
19 (0.007) B:.642_:[..G]G-----C[..C]:.628_:B (0.004) |
20 (0.006) B:.643_:[..G]G-----C[..C]:.627_:B (0.008) |
21 (0.005) B:.644_:[..G]Gx*---A[..A]:.626_:B (0.004) |
22 (0.004) B:.610_:[..G]G-*---U[..U]:.625_:B (0.008) |
23 (0.011) B:.611_:[..U]U-----A[..A]:.624_:B (0.003) |
24 (0.007) B:.612_:[..U]U-----A[..A]:.623_:B (0.013) |
25 (0.006) B:.613_:[..C]C----xG[..G]:.622_:B (0.007) |
26 (0.006) B:.614_:[..A]A-**-xu[4SU]:.608_:B (0.006) |
27 (0.007) B:.615_:[..G]Gx**+xC[..C]:.648_:B (0.006) x
28 (0.010) B:.619_:[..G]G-----C[..C]:.656_:B (0.004) +
В соответствии с полученными данными:
акцепторный стебель состоит из участка 601-607 и комплементарного ему участка 666-672;
Т-стебель состоит из участка 649-652 и комплементарного ему участка 662-665;
D-стебель состоит из участка 610-613 и комплементарного ему участка 622-625;
антикодоновый стебель состоит из участка 639-643 и комплементарного ему участка 627-631.
Эти выводы мы делаем на основании статьи и, в частности, данного изображения:
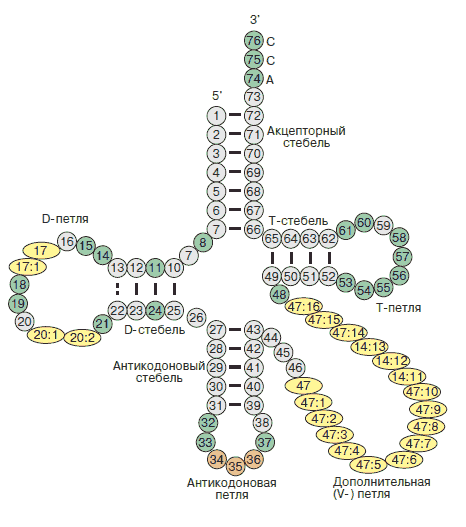
Далее был создан скрипт для получения в RasMol изображения остова цепи B исследуемой тРНК, где
акцепторный стебель выделен красным, Т-стебель - зеленым, D-стебель - синим, антикодоновый - оранжевым.
|
|
Тескт скрипта: background white restrict *B wireframe off cpk off backbone 100 define stem_acc (601-607,666-672) define stem_t (649-653, 661-665) define stem_d (610-613, 622-625) define stem_ant (639-643, 627-631) select stem_acc color red select stem_t color green select stem_d color blue select stem_ant color orange select not (stem_acc, stem_t, stem_d, stem_ant) color grey |
Неканоническая пара G - U (604 - 669):
Можно отметить отсутствие остатка тимидина в Т-петле и отсутствие дигидроуридинов в D-петле, возможно наличие вариабельной петли (вероятное месторасположение отмечено на рисунке красным):
Исследование третичной структуры
Программа analyze обнаружила 27 возможных стекинг-взаимодействий.
step i1-i2 i1-j2 j1-i2 j1-j2 sum
1 GG/CC 3.25( 1.63) 0.00( 0.00) 1.29( 0.00) 0.00( 0.00) 4.54( 1.63)
2 GA/UC 2.44( 1.00) 0.00( 0.00) 0.00( 0.00) 1.06( 0.31) 3.50( 1.31)
3 AG/UU 1.01( 1.00) 0.00( 0.00) 0.37( 0.00) 0.27( 0.00) 1.65( 1.00)
4 GC/GU 6.94( 3.74) 0.00( 0.00) 0.00( 0.00) 7.26( 4.40) 14.20( 8.14)
5 CG/CG 0.00( 0.00) 0.00( 0.00) 5.18( 2.30) 0.00( 0.00) 5.18( 2.30)
6 GG/CC 4.02( 2.78) 0.00( 0.00) 0.38( 0.00) 0.02( 0.00) 4.43( 2.78)
7 GG/PC 1.28( 0.12) 0.00( 0.00) 0.63( 0.00) 0.03( 0.00) 1.94( 0.12)
8 GC/GP 5.83( 2.80) 0.00( 0.00) 0.00( 0.00) 7.19( 3.81) 13.02( 6.60)
9 CG/CG 0.06( 0.00) 0.00( 0.00) 4.24( 1.54) 0.11( 0.00) 4.41( 1.54)
10 GG/CC 4.18( 2.83) 0.00( 0.00) 0.43( 0.00) 0.01( 0.00) 4.62( 2.83)
11 GG/CC 4.42( 3.05) 0.00( 0.00) 0.41( 0.00) 0.00( 0.00) 4.83( 3.05)
12 Gu/AC 7.68( 2.61) 0.00( 0.00) 0.00( 0.00) 5.71( 2.57) 13.39( 5.18)
13 uP/GA 5.83( 1.71) 0.00( 0.00) 0.00( 0.00) 6.19( 3.13) 12.02( 4.84)
14 PC/CG 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
15 CG/CC 2.11( 1.29) 0.00( 0.00) 0.00( 0.00) 2.29( 0.52) 4.39( 1.81)
16 GC/GC 5.05( 2.41) 0.00( 0.00) 0.00( 0.00) 6.27( 3.17) 11.32( 5.57)
17 CA/UG 0.09( 0.00) 0.00( 0.00) 2.94( 1.76) 0.00( 0.00) 3.03( 1.76)
18 AG/CU 0.52( 0.52) 0.00( 0.00) 0.66( 0.00) 0.00( 0.00) 1.18( 0.52)
19 GG/CC 2.44( 1.00) 0.00( 0.00) 0.28( 0.00) 0.08( 0.00) 2.80( 1.00)
20 GG/AC 1.22( 0.01) 0.00( 0.00) 0.00( 0.00) 4.38( 2.82) 5.61( 2.82)
21 GG/UA 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.91( 0.52) 0.91( 0.52)
22 GU/AU 6.75( 4.00) 0.00( 0.00) 0.00( 0.00) 5.59( 4.39) 12.34( 8.39)
23 UU/AA 1.26( 0.17) 0.00( 0.00) 0.00( 0.00) 2.04( 1.80) 3.30( 1.97)
24 UC/GA 1.27( 0.13) 0.00( 0.00) 0.00( 0.00) 4.55( 3.31) 5.82( 3.44)
25 CA/uG 0.00( 0.00) 1.48( 0.00) 5.68( 2.83) 0.00( 0.00) 7.16( 2.83)
26 AG/Cu 2.23( 0.60) 0.00( 0.00) 0.35( 0.00) 0.00( 0.00) 2.58( 0.60)
27 GG/CC 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00) 0.00( 0.00)
Исследуем возможность стекинг-взаимодействия между основаниями конца акцепторного стебля и начала Т-стебля.
Для этого рассмотрим водородные связи на данном участке:
7 (0.011) B:.607_:[..G]Gx----C[..C]:.666_:B (0.006) | 8 (0.007) B:.649_:[..G]G-*---P[PSU]:.665_:B (0.044) |Найдем стекинг-взаимодействие G607-G649 и C666-P665:
step i1-i2 i1-j2 j1-i2 j1-j2 sum 7 GG/PC 1.28( 0.12) 0.00( 0.00) 0.63( 0.00) 0.03( 0.00) 1.94( 0.12)
С помощью программы stack2img получим изображение этого стекинг-взаимодействия:
Площадь перекрытия - 1.94 квадратных ангстрем, это значение относительно невелико, что мы и можем наблюдать на рисунке.
Соответственно, возможность образования стекинг-взаимодействия достаточно низкая. 2. Есть ли дополнительные водородные связи между основаниями D- и Т-петель? Если есть, то укажите номера нуклеотидов в структуре, количество канонических и неканонических пар, приведите картинку, иллюстрирующую водородные связи в какой-либо из таких пар, на картинке приведите только основания и их номера.