Проект "Поиск белка с заданной функциональной специфичностью."
1. Описание функциональных особенностей заданной группы.
Заданный белок прототип - PurR_Ecoli - транскрипционный рецептор, принадлежт семейству GalR/LacI. Его димер контролирует несколько генов, участвующих в биосинтезе пуриновых и пиримидиновых нуклеотидов и самого PurR. Связывание с двумя продуктами метаболизма пурина, гипоксантином и гуанином, обеспечивает необходимую конформацию для cвязывания с ДНК.Структурные формулы эффекторов из БД KEGG:
гипоксантин
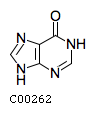
гуанин
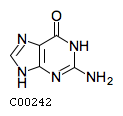
2. Создание множественного выравнивания доменов с разметкой по группам специфичности.
1. Доменная структура белка-прототипа PurR_Ecoli из БД PFAM:LacI - эффекторсвязывающий домен (3 - 48 а.о.), Peripla_BP_1 - ДНК-связывающий домен (59 - 329 а.о.).
На странице с описанием эффекторсвязываюего домена получим выравнивание всех доменов этого типа в формате FASTA. Для получения выравнивания доменов белков группы специфичности gntr из полного выравнивания напишем скрипт:
#!/bin/bash
for i in `cat gntr`; do
grep -A 20 ${i} PF00532_full.fasta >> PF00532_full_filtered.ali
done
Полученное выравнивание.
2. Выравнивания всех групп специфичности были импортированы в GeneDoc, гэпы удалены; заданная для исследования группа purr расположена сверху, остальные далее в алфавитном порядке. С помощью раскраски Shade Group Conserved позиции, консервативные для последовательностей одной группы, были покрашены в цвет группы, а позиции, консервативные для всех последовательностей, покрашены черным цветом. Множественное выравнивание эффекторсвязывающих доменов: eff.JPG,
и ДНК-связывающих доменов: dna.JPG.
3. Лого-изображение полных выравниваний:
эффекторсвязывающий домен,
ДНК-связывающий домен.
Лого-изображение выравниваний доменов группы специфичности purr:
эффекторсвязывающий домен,
ДНК-связывающий домен.
3. Поиск белка группы специфичности purr в протеоме Staphylococcus xylosus.
Был создан профиль для ДНК- и эффекторсвязывающих доменов группы специфичности purr:1. с помощью программы pfw был добавлен вес в выравнивания доменов;
2. программа pfmake использовалась для построения профиля;
3. autoscale - для нормирования полученного профиля относительно случайной базы данных.
Далее из БД UniProt был получен протеом Xanthomonas campestris pv. campestris (strain B100).
В данном протеоме был проведен поиск белков группы специфичности purr c помощью программы pfsearch (оптимальное пороговое значение - 10).
Для ДНК-связывающего домена нашлось 9 возможных гомологов, для эффекторсвязывающего - 10, причем 9 из них совпадают с находками для ДНК- связывающего домена, что повышает вероятность того, что среди них будет белок со специфичностью purr.
Выравнивание ДНК-связывающего домена, полученное с помощью ClustalW2: dna.aln, dna.GIF. Эффекторсвязывающего домена: eff.aln, eff.GIF.
Назад