К сожалению количество "точек" по которым можно было бы создавать выравнивание 3D структуры в LYS_BPH19 оказалось равным 1. Была взята последовательность LYS_MERLU, но и у нее в выравнивании была только одна консервативная группа связывания с лигандом. В дальнейшем вся работа происходит надо LYS_OSTED.
После получения выравнивания 1LMP и LYS_OSTED программой ClastalW необходимо было добавить строки с параметрами для Modeller. Конечный файл выравнивания выглядит так.
Для дальейшей работы так же пригодится модифицированный файл структуры 1lmp.
Для создания скрипта необходимо было найти аминокислоты, которые связывались бы с лигандом, а так же были консервативными. Данные материалы были получены при изучении pdb структуры и выравнивания:
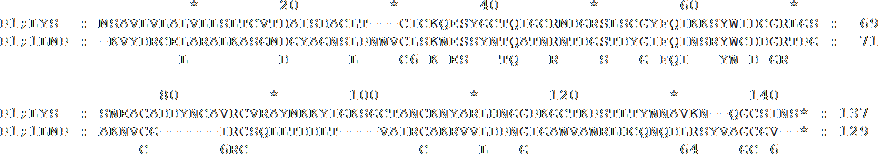
Сам скрипт можно посмотреть здесь
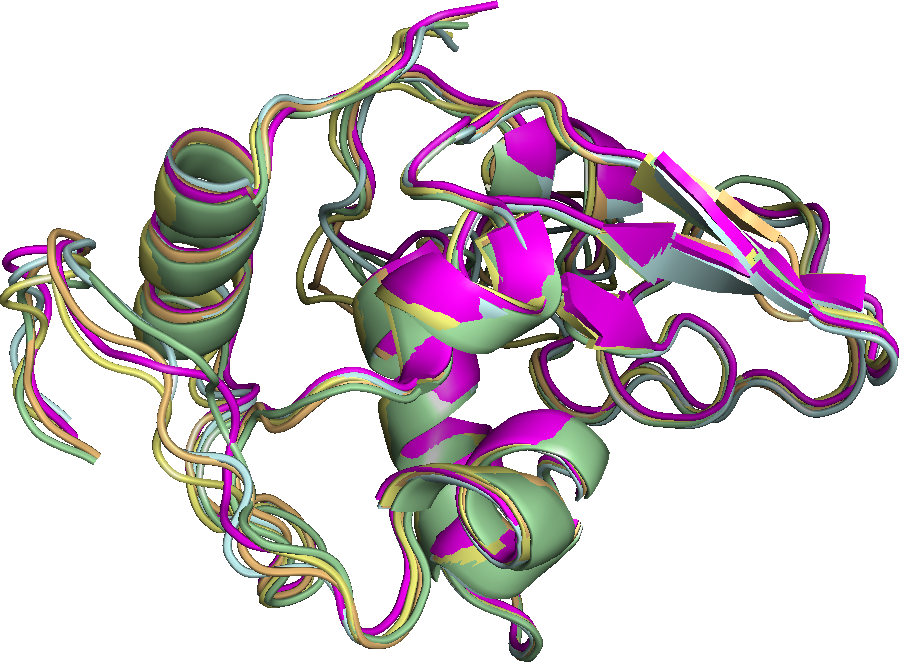
Все 5 структур получились очень похожи. Небольшие изменения есть в длинне альфа испиралей, а так же в существовании и размерах бетта-листа.
Используя сервис WHATIF были получены данные о правильности моделирования:
1 структура:
Structure Z-scores, positive is better than average: 1st generation packing quality : -3.884 (poor) Ramachandran plot appearance : -2.523 chi-1/chi-2 rotamer normality : -4.230 (bad) Backbone conformation : -4.982 (bad) RMS Z-scores, should be close to 1.0: Bond lengths : 0.946 Bond angles : 1.326 Omega angle restraints : 0.858 Side chain planarity : 0.559 (tight) Improper dihedral distribution : 0.999 Inside/Outside distribution : 1.206 (unusual)2 структура:
Structure Z-scores, positive is better than average: 1st generation packing quality : -3.523 Ramachandran plot appearance : -1.905 chi-1/chi-2 rotamer normality : -3.150 (poor) Backbone conformation : -4.146 (bad) RMS Z-scores, should be close to 1.0: Bond lengths : 0.945 Bond angles : 1.263 Omega angle restraints : 0.803 Side chain planarity : 0.426 (tight) Improper dihedral distribution : 0.861 Inside/Outside distribution : 1.213 (unusual)3 структура:
Structure Z-scores, positive is better than average: 1st generation packing quality : -3.639 Ramachandran plot appearance : -1.721 chi-1/chi-2 rotamer normality : -4.176 (bad) Backbone conformation : -4.554 (bad) RMS Z-scores, should be close to 1.0: Bond lengths : 0.936 Bond angles : 1.339 Omega angle restraints : 0.776 Side chain planarity : 0.338 (tight) Improper dihedral distribution : 0.888 Inside/Outside distribution : 1.233 (unusual)4 структура:
Structure Z-scores, positive is better than average: 1st generation packing quality : -3.690 Ramachandran plot appearance : -2.396 chi-1/chi-2 rotamer normality : -3.673 (poor) Backbone conformation : -5.275 (bad) RMS Z-scores, should be close to 1.0: Bond lengths : 0.968 Bond angles : 1.363 Omega angle restraints : 0.906 Side chain planarity : 0.249 (tight) Improper dihedral distribution : 0.949 Inside/Outside distribution : 1.217 (unusual)5 структура:
Structure Z-scores, positive is better than average: 1st generation packing quality : -3.610 Ramachandran plot appearance : -1.851 chi-1/chi-2 rotamer normality : -3.703 (poor) Backbone conformation : -4.814 (bad) RMS Z-scores, should be close to 1.0: Bond lengths : 0.939 Bond angles : 1.268 Omega angle restraints : 0.778 Side chain planarity : 0.658 (tight) Improper dihedral distribution : 0.945 Inside/Outside distribution : 1.230 (unusual)
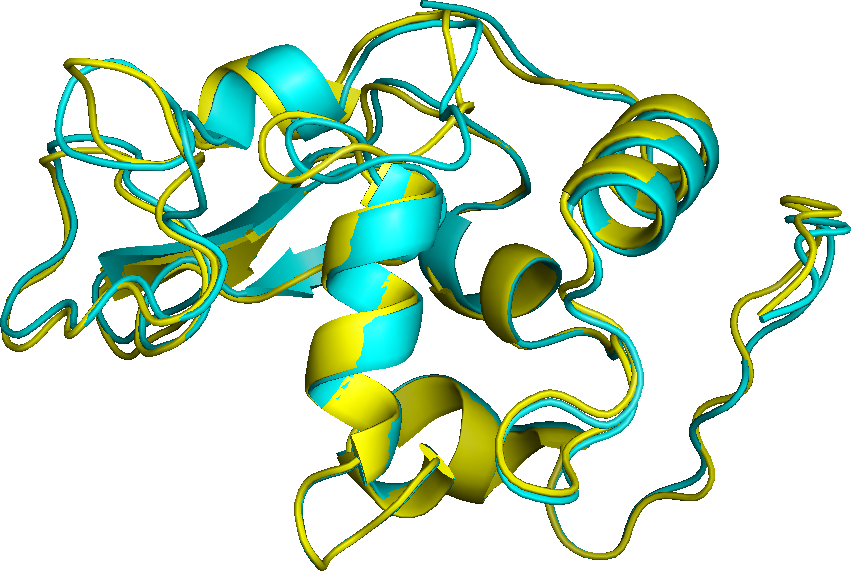
По полученным данным, лучшей структурой оказывается вторая, она представлена на изображении желтым, исходная циановым.