При просмотре log файла найдены 3 лучших расположения лиганда и разница между ними:
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -5.6 0.000 0.000
2 -5.5 1.944 5.393
3 -5.4 2.759 4.435
Все состояни зафиксированы и представлены на изображении:
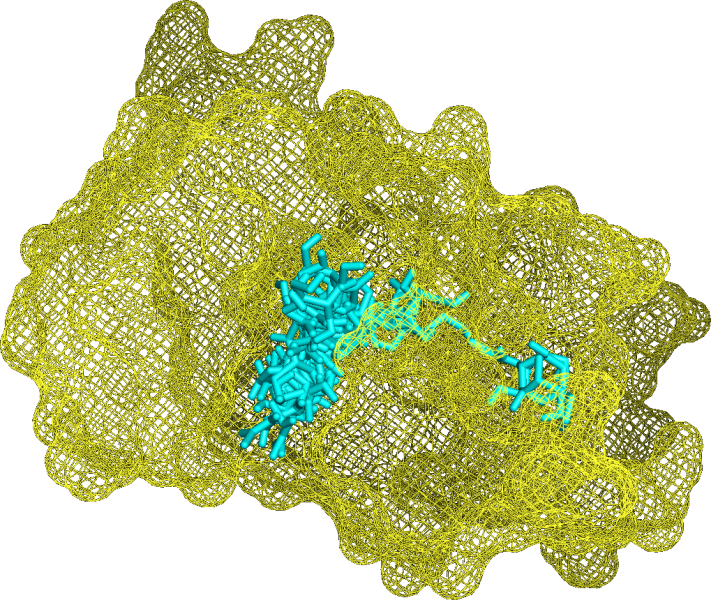
Из log файла получаем 3 лучших расположения лиганда в этом случае.
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -6.0 0.000 0.000
2 -5.9 1.337 3.398
3 -5.5 1.188 2.105
Изменения в структуре белка обозначены фиолетовым, новое расположение лигда - красным: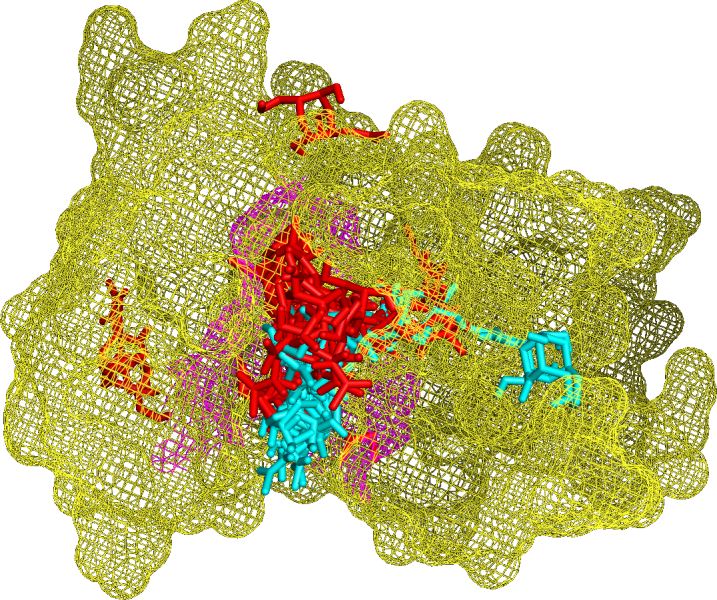
Теоретически подвижный докинг лучше, однако в результате подвижности мы получаем много лишних возможных взаимодействий с лигандом вне активного центра. К сожалению, докинг не смог расположить лиганд так, как он располагался в исходной модели.
NAG содержит в себе СH3C(=O)NH группу с метильным радикалом. Созданы лиганды где радикал изменен на:
OH, NH2, H, Ph
Для всех измененных лигандов был проведен обычный докинг:
OH:
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -5.7 0.000 0.000
2 -5.7 4.273 7.992
3 -5.5 5.925 7.789
NH2:
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -5.9 0.000 0.000
2 -5.8 2.174 5.360
3 -5.8 4.400 8.065
H:
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -5.2 0.000 0.000
2 -5.2 2.118 4.166
3 -5.2 2.917 3.961
Ph:
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -6.9 0.000 0.000
2 -6.8 2.188 2.728
3 -6.4 8.440 11.256
Единственное, что можно сказать: фенил неплохо увеличивает афинность, однако афинность не самый лучший показатель связывания.Так же был проведен докинг с подвижными радикалами:
OH:
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -5.8 0.000 0.000
2 -5.7 1.809 3.471
3 -5.6 1.309 2.320
NH2:
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -6.1 0.000 0.000
2 -5.9 1.602 3.439
3 -5.5 1.066 2.043
Ph:
mode | affinity | dist from best mode
| (kcal/mol) | rmsd l.b.| rmsd u.b.
-----+------------+----------+----------
1 -6.5 0.000 0.000
2 -6.4 6.341 8.512
3 -6.4 3.959 6.404
Для лиганда OH и NH2 афинность увеличилась (в среднем), однако для Ph упала. Скорее всего целесообразнее использовать подвижный докинг, но используя нечно иное, чем Vina.