Содержит последовательность мономера пуринового репрессора, связанного с одной цепью ДНК.
На сервере PDBe не оказалось биологической единицы для данной записи. Поэтому на сайте RCSB был скачан файл авторов структуры с биологической единицей 1jfs.pdb1. Но в этом файле различные ассиметрические единицы находились в различных моделях, поэтому был написан скрипт на языке Perl biol.pl, который объединяет две модели в одну, переименновывая цепи атомов второй модели A -> C, B -> D. Таким образом был получен необходимый для работы файл biol.pdb
Изображения поверхностей контакта
Для каждого пункта были написаны скрипты для PyMOL, после чего улучшалось качество поверхности командойset surface_quality, 1и создавалось изображение.
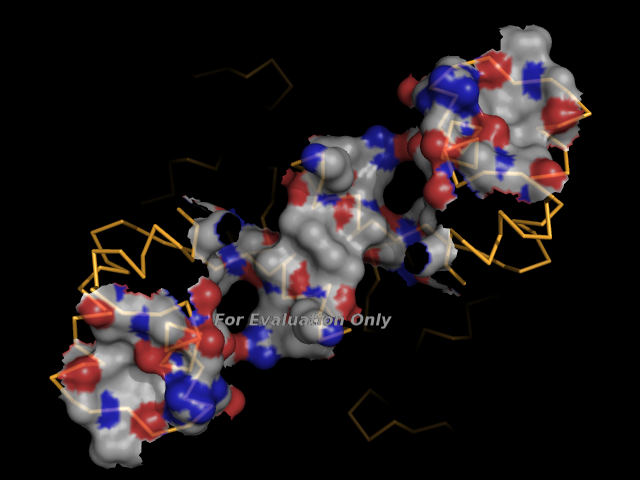
- Поверхность контакта мономера белка с симметричным мономером на фоне остовной
(ribbon) модели мономера (monomer.pml).
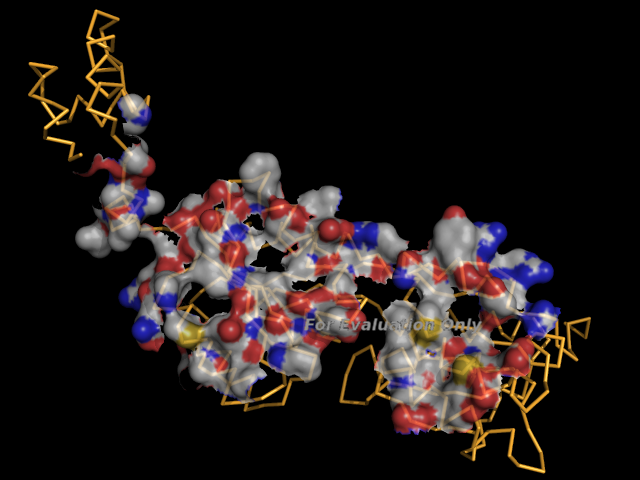
- Поверхность контакта димера белков с двойной спиралью ДНК на фоне остовной модели части белка,
вовлечённой в контакт (prot.pml).
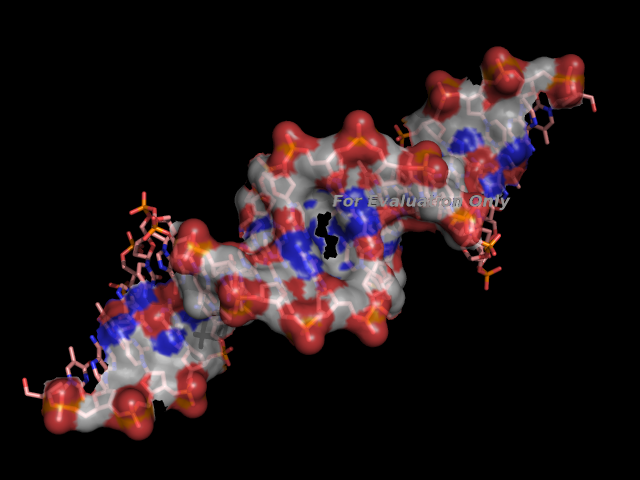
- Поверхность контакта ДНК с димером белков на фоне проволочной (sticks) модели двойной спирали
(dna.pml).
Cервис PROTORP
Данный сервис позволяет узнать различные характеристики взаимодействия двух цепей из файла PDB, например:Площадь контакта белковых мономеров составляет 2551.62 Å2, 42.72% этой площади приходится на гидрофобное взаимодействие.
Cервис CluD
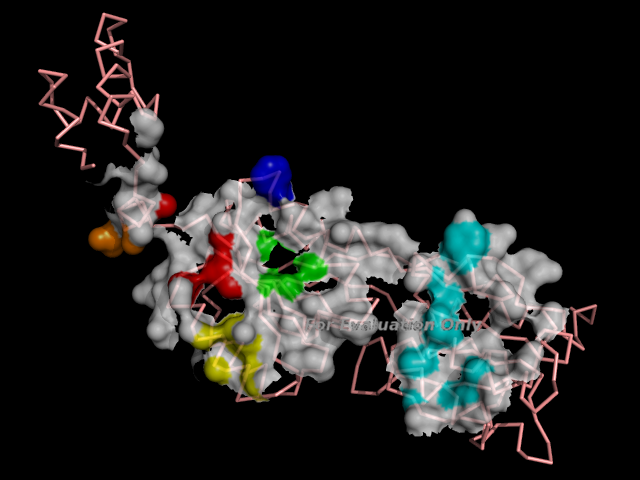
С помощью этого сервиса были определены гидрофобные кластеры на интерфейсе мономеров белка объёмом не менее 10 атомов.Выходной файл сервиса - таблица с атомами, относящимися к гидрофобным кластерам. Довольно странно, что этот файл содержал атомы кластеров, объем которых ниже заданного программе. Такие атомы не учитывались в упражнении.
Создано то же изображение, что в упр. 1A, на котором поверхность, относящаяся к атомам, входящим в найденные гидрофобные кластеры, выделена различными цветами для каждого кластера (clud.pml).
Cервис pDomains
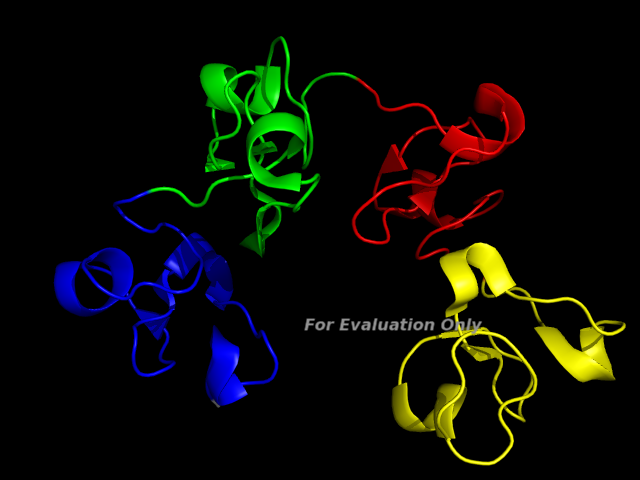
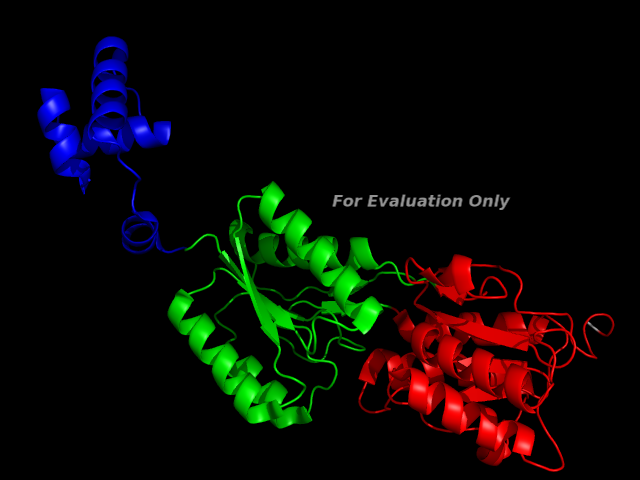
Данный сервис позволяет определить доменную структуру одной цепи из файла PDB, используя различные методы.- Для цепи A белка пуринового репрессора различные методы определили доменную структуру
по-разному. Наиболее адекватными мне показались результаты метода CATH. Он выделил
следующие домены:
- синий 2-58
- зеленый 59-160 , 291-322
- красный 161-290 , 323-339
- Мой белок с точки зрения доменной структуры не слишком интересен, так как представляет из себя
бета-бочонок. Поэтому в банке PDB была найдена запись 2x52.pdb.
Сервисы метода сходным образом определили доменную структуру цепи A этой записи. Я выбрал метод pdb,
так как его результаты выглядят немного адекватнее. Он выделил следующие домены:
- синий 2-42
- зеленый 43-85
- красный 86-128
- желтый 129-171