В моей записи содержится всего один домен, поэтому возьмем для работы также запись 1T9H.
Классификация доменов согласно SCOP
Моя записьДомен: Внешняя мембранная протеаза OMPT из E.coli (Outer membrane protease OMPT from Escherichia coli)
- Расположен во обоих цепях A и B записи, занимает цепи целиком.
- Классификация по SCOP
- Класс: Белки и пептиды на мембране и на поверхности клетки (Membrane and cell surface proteins and peptides)
- Укладка: Трансмембранные бета-бочонки (Transmembrane beta-barrels)
- Суперсемейство: OMPT-подобные (OMPT-like)
- Семейство: Внешняя мембранная протеаза OMPT (Outer membrane protease OMPT)
- В данном суперсемействе содержится 2 семейства.
- Данную укладку имеет 6 суперсемейств.
Запись 1T9H
- Домен: N-концевой домен вероятной ГТФазы EngC (YjeQ) из B.subtilis (Probable GTPase EngC (YjeQ), N-terminal domain from Bacillus subtilis)
- Расположен в цепи A в позициях 1-67.
- Классификация по SCOP
- Класс: Все бета-структурные белки (All beta proteins)
- Укладка: OB-укладка (OB-fold)
- Суперсемейство: Белки, связывающие нуклеиновые кислоты (Nucleic acid-binding proteins)
- Семейство: Подобные ДНК-связывающему домену при холодовом шоке (Cold shock DNA-binding domain-like)
- В данном суперсемействе содержится 16 семейств.
- Данную укладку имеет 16 суперсемейств.
- Домен: C-концевой домен вероятной ГТФазы EngC (YjeQ) из B.subtilis (Probable GTPase EngC (YjeQ), C-terminal domain from Bacillus subtilis)
- Расположен в цепи A в позициях 68-298.
- Классификация по SCOP
- Класс: Альфа-спиральные и бета-структурные белки (a/b) (Alpha and beta proteins (a/b))
- Укладка: Нуклеозид-трифофат гидролазы, содержащие P-петлю (P-loop containing nucleoside triphosphate hydrolases)
- Суперсемейство: Нуклеозид-трифофат гидролазы, содержащие P-петлю (P-loop containing nucleoside triphosphate hydrolases)
- Семейство: G-белки (G proteins)
- В данном суперсемействе содержится 24 семейства.
- Данную укладку имеет 1 суперсемейство.
Классификация доменов согласно CATH
Моя записьДомен 0 из цепи A (цепь B аналогична)
- Расположен в позициях 1-297, в цепи B в позициях 11-297.
- Классификация по CATH: 2.40.128.90.1.1.1.1.1
- Класс: Главным образом бета-структурные (Mainly Beta)
- Архитектура: Бета-бочонок (Beta Barrel)
- Топология: Липокалин (Lipocalin)
- Суперсемейство: OMPT-подобные (OMPT-like)
- В данном суперсемействе содержится 1 семейство.
- Данную топологию имеет 9 суперсемейств.
Запись 1T9H
- Домен 1 в цепи A
- Расположен в позициях -7 - 65.
- Классификация по CATH: 2.40.50.140.28.1.1.1.1
- Класс: Главным образом бета-структурные (Mainly Beta)
- Архитектура: Бета-бочонок (Beta Barrel)
- Топология: OB-укладка, Дигидролипоамидная ацетилтрансфераза (OB fold (Dihydrolipoamide Acetyltransferase, E2P))
- Суперсемейство: Белки, связывающие нуклеиновые кислоты (Nucleic acid-binding proteins)
- В данном суперсемействе содержится 89 семейство.
- Данную укладку имеет 17 суперсемейств.
- Домен 2 в цепи A
- Расположен в позициях 66-230.
- Классификация по CATH: 3.40.50.300.106.1.1.1.1
- Класс: Альфа-спиральные и бета-структурные (Alpha Beta)
- Архитектура: Трехслойный (альфа-бета-альфа) сэндвич (3-Layer(aba) Sandwich)
- Топология: Укладка Россмана (Rossmann fold)
- Суперсемейство: Нуклеозид-трифофат гидролазы, содержащие P-петлю (P-loop containing nucleoside triphosphate hydrolases)
- В данном суперсемействе содержится 208 семейств.
- Данную укладку имеет 120 суперсемейств.
- Домен 3 в цепи A
- Расположен в позициях 231-298.
- Классификация по CATH: 1.10.40.50.1.1.1.1.1
- Класс: Главным образом альфа-спиральные (Alpha Alpha)
- Архитектура: Ортогональное расслоение (Orthogonal Bundle)
- Топология: Белок R1 рибонуклеотидной редуктазы, домен 1 (Ribonucleotide Reductase Protein R1; domain 1)
- Суперсемейство: Вероятная ГТФаза EngC, домен 3 (Probable gtpase engc; domain 3)
- В данном суперсемействе содержится 2 семейства.
- Данную укладку имеет 4 суперсемейства.
Сравнение SCOP и CATH
Моя записьДомены выделены одинаково (полные цепи), но классификация различна. Одинаковы только суперсемейства (OMPT-подобные). Кроме того, сходны архитектура CATH (Бета-бочонок) и укладка SCOP (Трансмембранные бета-бочонки). Также любопытно различие в классах: класс в CATH - "Главным образом бета-структурные", но в SCOP есть практически такой же класс (Все бета-структурные белки), что видно из классификации в записи 1T9H, а домен моей записи почему-то был отнесен к другому классу (Белки и пептиды на мембране и на поверхности клетки).
Запись 1T9H
Выделено разное количество доменов.
- Первые домены почти совпадают. Классы, укладка (SCOP) и топология (CATH), а также суперсемейства практически идентичны.
- Оставшаяся часть цепи имеет один домен по SCOP и два - по CATH. По классификации похожи домен из SCOP и первый на участке домен из CATH. У них одинаковые класс и суперсемейство.
Выравнивание двух доменов с одной топологией по CATH, но из разных суперсемейств
Для работы выбрано:- Топология по CATH: Укладка Россмана.
- Домен: 1t9hA02 (домен 2 цепи A записи 1T9H), позиции: 66-230.
- Домен: 2qipA00 (единственный домен цепи A записи QIP), позиции 2-162.
Главное геометрическое ядро, полученное на сервисе Geometrical core
| Pos. | 1T9H_A | 2QIP_A |
| 87 | ASP79 | LYS8 |
| 88 | GLN80 | ILE9 |
| 90 | VAL82 | ILE11 |
| 92 | VAL84 | VAL13 |
| 113 | LEU96 | ASP31 |
| 114 | LEU97 | TYR32 |
| 115 | ASP98 | ASN33 |
| 116 | ARG99 | GLN34 |
| 117 | PHE100 | PHE35 |
| 118 | LEU101 | TRP36 |
| 119 | VAL102 | TYR37 |
| 120 | LEU103 | VAL38 |
| 121 | VAL104 | ALA39 |
| 187 | ASP157 | ASP98 |
| 190 | PRO160 | GLU101 |
| 191 | HIS161 | ILE102 |
| 192 | PHE162 | ALA103 |
| 193 | GLN163 | PRO104 |
| 196 | THR166 | ASP107 |
| 197 | THR167 | ARG108 |
Скрипт PyMOL для создания изображения: gc.pml
Командой pair_fit были совмещены атомы геометрического ядра, rmsd = 1.263.
Показаны только интересующие нас домены, чтоб не загромождать изображение. Раскраска:
- 1T9H: бета-тяжи красные, остальное зеленое
- 2QIP: бета-тяжи розовые, остальное голубое
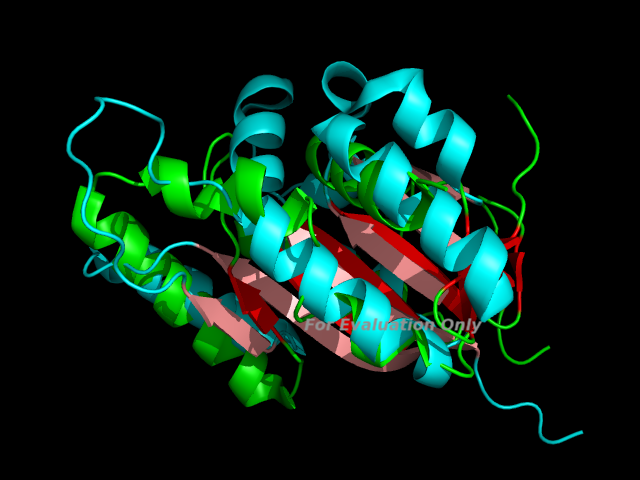
Данные домены имеют довольно много элементов вторичной структуры, поэтому было трудно сделать хорошую картинку. Желательно, рассматривать данное выравнивание программой PyMOL с использованием предоставленного скрипта.
В целом, домены выравнились неплохо. Близко лежат бета-тяжи в центре глобул. Альфа-спирали в доменах немного различаются, но некоторые совпадающие совместились хорошо.