Занятие 8.
- Объясните, что значит код заданного фермента.
Открыла страницу БД UNIPROT с описанием DACA_ECOLI белка
1)- сам код EC=3.4.16.4;
- расшифровка для каждого его пункта на русском и английском языках;
EC=3-Hydrolases (Гидролаза- класс ферментов, катализирующий гидролиз ковалентной связи)
EC=3.4-Peptidases (Пептидазы- гидролизуемая связь- пептидная)
EC=3.4.16-Serine-type carboxypeptidases (Карбоксипептидаза серинового типа - ферменты класса гидролаз, катализирующие гидролитич. отщепление С-концевых аминокислотных остатков в молекуле белков и пептидов)
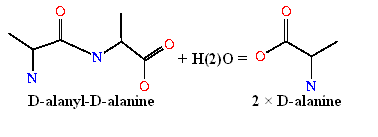
EC=3.4.16.4-serine-type D-Ala-D-Ala carboxypeptidase (Сериновый тип D-аланин-D-аланин карбоксипептидаза- у карбоксипептидазы активный центр серин, отщепление по D-аланин-D-аланин) - уравнение катализируемой реакции;
(Ac)2-L-Lys-D-Ala(<=здесь отщепление=>)D-Ala отщепление - Фермент бактериальный, связывающийся с мембраной, ингибируется пенициллином и другими бета-лактамными антибиотиками, которые ацилируют активный сайт серин.
- На сайте PDB было получено графическое изображение
катализируемой реакции
2)- сам код EC=3.5.2.6;
- расшифровка для каждого его пункта на русском и английском языках;
EC=3-Hydrolases(Гидролаза- класс ферментов, катализирующий гидролиз ковалентной связи)
EC=3.5-Acting on carbon-nitrogen bonds, other than peptide bonds(гидродизуемая связь- азот-углеродная, кроме пептидных)
EC=3.5.2- In cyclic amides(В циклических амидах действует)
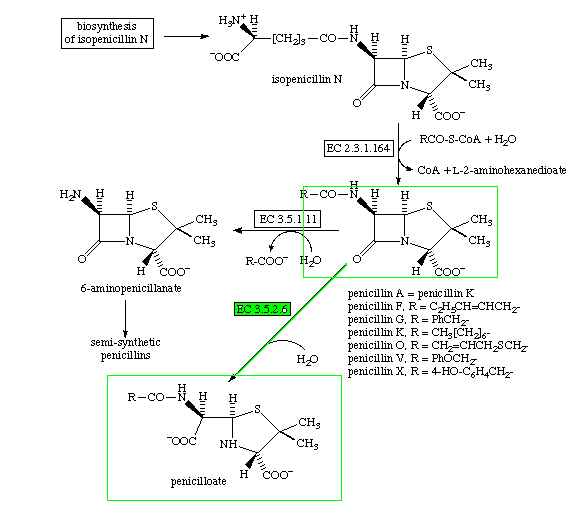
EC=3.5.2.6-beta-lactamase(бета-лактамаза- гидролизуют бета-лактамное кольцо)
бета-лактамное кольцо является частью структуры нескольких семейств антибиотиков, в частности пенициллинов, цефалоспоринов, карбапенов и монобактамов. Эти антибиотики ингибируют процессы синтеза клеточной стенки бактерий, особенно грам-положительных. - уравнение катализируемой реакции;
A beta-lactam + H2O = a substituted(замещенный) beta-amino acid - Метаболический путь.(зеленым цветом выделен фермент, а в зеленой рамочке
субстрат и продукт реакции гидролиза)
- Определите, в каких метаболических путях участвует изучаемый фермент.
Идентификатор KEGG- eco00550 - названиe на русском и английском языках- Peptidoglycan biosynthesis (Биосинтез пептидогликана)
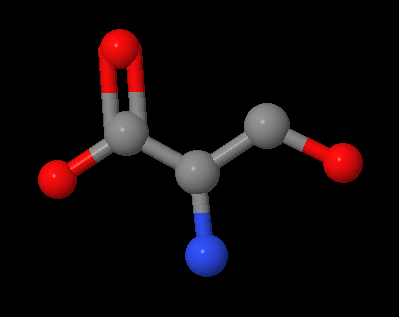
- название L-Serine (серин в виде оптического изомера L)
- идентификатор C00065
- структурная формулу.
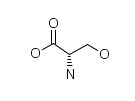
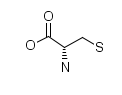
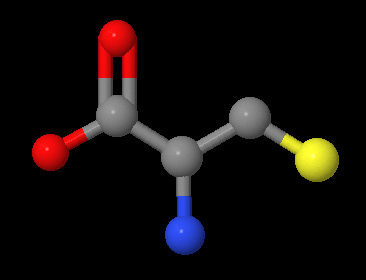
- название L-Cysteine (цистеин в виде оптического изомера L)
- идентификатор C00097
- структурная формулу.
- Найдите метаболический путь от одного заданного вещества к другому Для поиска реактантов (или, как часто говорят, интермедиатов или лигандов), а также ферментов в БД KEGG Pathway есть два инструмента: "Search objects in pathways" и "Color objects in pathways". Предлагается работать со вторым.
- Сравните метаболические пути у разных организмов Определим, возможна ли выбранная в упр.4 цепочка ферментативных реакций у организмов, перечисленных в таблице ниже.
- Сравните ферменты из далеких организмов
- Для сравнения был выбран фермент с EC=4.1.1.21. Этот фермент является лиазой.
- Сравним доменную организацию (по PFAM) найденных белков,используя режим SW_InterProMatches
для просмотра находок.
Для белка человека в Pfam найдены 2 домена: SAICAR synthetase (синтетаза) PF01259 и AIR carboxylase (карбоксилаза) PF00731
Для белка археи в Pfam найден 1 домен: AIR carboxylase (карбоксилаза) PF00731
У белка человека и археи есть одинаковй домен AIR carboxylase - С помощью инструментов KEGG найдем для выбранного человеческого белка лучшего ортолога
из архей, а для архейного –- лучшего ортолога у эукариот.
Ген, соответствующий белку PUR6_HUMAN, PAICS , id в KEGG - hsa:10606. Ген, соответствующий белку PUR6_ARCFU, purE , в KEGG - afu:AF1271
Лучший ортолог для гена белка PUR6_ARCFU из эукариот cme:CME023C, синтетаза\карбоксилаза
процент идентичности: 0.477
длина выравнивания: 597
организм: Cyanidioschyzon merolae (фотосинтезирующий организм, живет в кислых горячих водах)
3.Найдите в KEGG структурные формулы заданных соединений.
Открыла страницу оглавления KEGG. Найдите ссылку на базу данных химических соединений (KEGG LIGAND). Провела поиск по каждому из названий веществВ отчете для каждого соединения привела следующие сведения: 1)
2)
В поле запроса ввела найденные в упр.3 идентификаторы соединений. Указала, что хочу раскрасить первое соединение красным, а второе - зеленым. Синтаксис показан на примерах справа от поля запроса. После ввода запроса на экране появится список карт. Открыла ту, для которой указаны два идентификатора.
Выбранная цепочка ферментативных реакций:
путь: метаболизм глицина, серина и треонина
цепочка: L-серин →L-цистеин
Найденный путь надо отметить на карте.
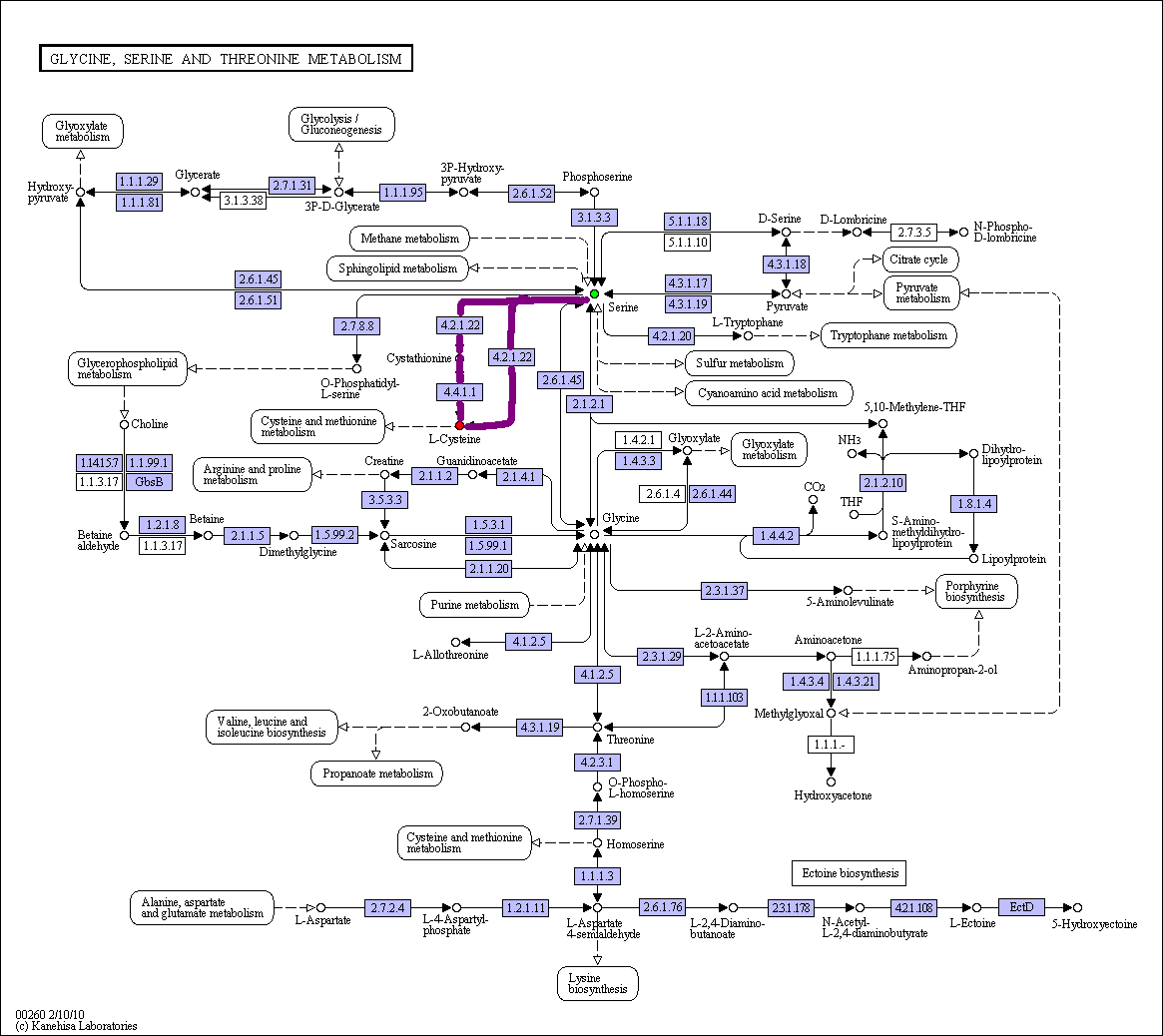 Код промежуточного соединения C02291.
Раскрашенная карта (первое-красным, последнее-зеленым, промежуточное-желтым).
Код промежуточного соединения C02291.
Раскрашенная карта (первое-красным, последнее-зеленым, промежуточное-желтым).
| Организм | Возможна ли цепочка реакций (да/нет/неизвестно) |
Обоснование |
| Escherichia coli K-12 MG1655 | нет eco00260.gif |
нет ферментов 4.2.1.22, 4.4.1.1, 2.6.1.45 |
| Archaeoglobus fulgidus | нет afu00260.gif |
нет ферментов 4.2.1.22, 4.4.1.1, 2.6.1.45, остальные есть |
| Arabidopsis thaliana | нет ath00260.gif |
нет ферментов 4.2.1.22, 4.4.1.1, остальные есть |
| Homo sapiens | нет hsa00260.gif |
нет фермента 2.6.1.45, остальные есть |
В SRS был создан запрос для поиска фермента среди белков человека и археи Archaeoglobus fulgidus.(Опция маски (Use wildcards) необходима, чтобы находились только ферменты с кодом 4.1.1.21, а не 4.1.1.21*)
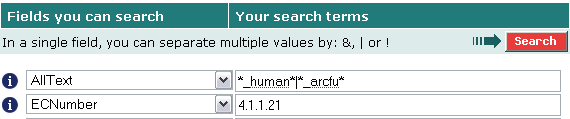
Было найдено 2 находки (одна относится к человеку, другая - к архее) PUR6_ARCFU и PUR6_HUMAN
Лучший ортолог для гена белка PUR6_HUMAN из архей sto:ST1493, синтетаза
процент идентичности: 0.380длина выравнивания: 234
организм: Sulfolobus tokodaii (живет в горячих водах)